تحليل التمييز الخطي في r (خطوة بخطوة)
التحليل التمييزي الخطي هو طريقة يمكنك استخدامها عندما يكون لديك مجموعة من المتغيرات المتوقعة وترغب في تصنيف متغير الاستجابة إلى فئتين أو أكثر.
يقدم هذا البرنامج التعليمي مثالاً خطوة بخطوة لكيفية إجراء تحليل التمييز الخطي في R.
الخطوة 1: تحميل المكتبات الضرورية
أولاً، سنقوم بتحميل المكتبات اللازمة لهذا المثال:
library (MASS)
library (ggplot2)
الخطوة 2: تحميل البيانات
في هذا المثال، سوف نستخدم مجموعة بيانات القزحية المضمنة في R. ويوضح التعليمة البرمجية التالية كيفية تحميل مجموعة البيانات هذه وعرضها:
#attach iris dataset to make it easy to work with attach(iris) #view structure of dataset str(iris) 'data.frame': 150 obs. of 5 variables: $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ... $ Sepal.Width: num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ... $Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ... $Petal.Width: num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ... $ Species: Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 ...
يمكننا أن نرى أن مجموعة البيانات تحتوي على 5 متغيرات و150 ملاحظة في المجموع.
في هذا المثال، سنقوم ببناء نموذج تحليل تمييزي خطي لتصنيف الأنواع التي تنتمي إليها زهرة معينة.
سوف نستخدم المتغيرات المتوقعة التالية في النموذج:
- طول
- عرض
- البتلة.الطول
- البتلة.العرض
وسنستخدمها للتنبؤ بمتغير استجابة الأنواع ، الذي يدعم الفئات الثلاثة المحتملة التالية:
- سيتوسا
- المبرقشة
- فرجينيا
الخطوة 3: قياس البيانات
أحد الافتراضات الرئيسية للتحليل التمييزي الخطي هو أن كل متغير من المتغيرات المتوقعة له نفس التباين. هناك طريقة بسيطة لضمان استيفاء هذا الافتراض وهي قياس كل متغير بحيث يكون متوسطه 0 وانحرافه المعياري 1.
يمكننا القيام بذلك بسرعة في R باستخدام وظيفة المقياس () :
#scale each predictor variable (ie first 4 columns)
iris[1:4] <- scale(iris[1:4])
يمكننا استخدام الدالة application() للتحقق من أن كل متغير متنبئ لديه الآن متوسط 0 وانحراف معياري 1:
#find mean of each predictor variable apply(iris[1:4], 2, mean) Sepal.Length Sepal.Width Petal.Length Petal.Width -4.484318e-16 2.034094e-16 -2.895326e-17 -3.663049e-17 #find standard deviation of each predictor variable apply(iris[1:4], 2, sd) Sepal.Length Sepal.Width Petal.Length Petal.Width 1 1 1 1
الخطوة 4: إنشاء عينات التدريب والاختبار
بعد ذلك، سنقوم بتقسيم مجموعة البيانات إلى مجموعة تدريب لتدريب النموذج عليها ومجموعة اختبار لاختبار النموذج عليها:
#make this example reproducible set.seed(1) #Use 70% of dataset as training set and remaining 30% as testing set sample <- sample(c( TRUE , FALSE ), nrow (iris), replace = TRUE , prob =c(0.7,0.3)) train <- iris[sample, ] test <- iris[!sample, ]
الخطوة 5: ضبط نموذج LDA
بعد ذلك، سنستخدم الدالة lda() من الحزمة MASS لتكييف نموذج LDA مع بياناتنا:
#fit LDA model model <- lda(Species~., data=train) #view model output model Call: lda(Species ~ ., data = train) Prior probabilities of groups: setosa versicolor virginica 0.3207547 0.3207547 0.3584906 Group means: Sepal.Length Sepal.Width Petal.Length Petal.Width setosa -1.0397484 0.8131654 -1.2891006 -1.2570316 versicolor 0.1820921 -0.6038909 0.3403524 0.2208153 virginica 0.9582674 -0.1919146 1.0389776 1.1229172 Coefficients of linear discriminants: LD1 LD2 Sepal.Length 0.7922820 0.5294210 Sepal.Width 0.5710586 0.7130743 Petal.Length -4.0762061 -2.7305131 Petal.Width -2.0602181 2.6326229 Proportion of traces: LD1 LD2 0.9921 0.0079
وإليك كيفية تفسير نتائج النموذج:
الاحتمالات السابقة للمجموعة: تمثل نسب كل نوع في مجموعة التدريب. على سبيل المثال، 35.8% من جميع الملاحظات في مجموعة التدريب كانت لأنواع فيرجينيكا .
متوسطات المجموعة: تعرض متوسط قيم كل متغير متنبئ لكل نوع.
معاملات التمييز الخطي: تعرض المجموعة الخطية من متغيرات التوقع المستخدمة لتدريب قاعدة قرار نموذج LDA. على سبيل المثال:
- LD1: 0.792 * طول الكأس + 0.571 * عرض الكأس – 4.076 * طول البتلة – 2.06 * عرض البتلة
- LD2: 0.529 * طول الكأس + 0.713 * عرض الكأس – 2.731 * طول البتلة + 2.63 * عرض البتلة
نسبة التتبع: تعرض النسبة المئوية للفصل الذي حققته كل وظيفة تمييزية خطية.
الخطوة 6: استخدم النموذج لعمل تنبؤات
بمجرد أن نلائم النموذج باستخدام بيانات التدريب الخاصة بنا، يمكننا استخدامه للتنبؤ ببيانات الاختبار الخاصة بنا:
#use LDA model to make predictions on test data predicted <- predict (model, test) names(predicted) [1] "class" "posterior" "x"
يؤدي هذا إلى إرجاع قائمة بثلاثة متغيرات:
- الطبقة: الطبقة المتوقعة
- الخلفي: الاحتمال الخلفي الذي تنتمي إليه الملاحظة لكل فئة
- س: التمييز الخطي
يمكننا تصور كل من هذه النتائج بسرعة للملاحظات الستة الأولى في مجموعة بيانات الاختبار الخاصة بنا:
#view predicted class for first six observations in test set head(predicted$class) [1] setosa setosa setosa setosa setosa setosa Levels: setosa versicolor virginica #view posterior probabilities for first six observations in test set head(predicted$posterior) setosa versicolor virginica 4 1 2.425563e-17 1.341984e-35 6 1 1.400976e-21 4.482684e-40 7 1 3.345770e-19 1.511748e-37 15 1 6.389105e-31 7.361660e-53 17 1 1.193282e-25 2.238696e-45 18 1 6.445594e-22 4.894053e-41 #view linear discriminants for first six observations in test set head(predicted$x) LD1 LD2 4 7.150360 -0.7177382 6 7.961538 1.4839408 7 7.504033 0.2731178 15 10.170378 1.9859027 17 8.885168 2.1026494 18 8.113443 0.7563902
يمكننا استخدام الكود التالي لمعرفة النسبة المئوية للملاحظات التي تنبأ بها نموذج LDA بشكل صحيح حول الأنواع:
#find accuracy of model
mean(predicted$class==test$Species)
[1] 1
وتبين أن النموذج تنبأ بشكل صحيح بالأنواع بنسبة 100% من الملاحظات في مجموعة بيانات الاختبار الخاصة بنا.
في العالم الحقيقي، نادرًا ما يتنبأ نموذج LDA بنتائج كل فئة بشكل صحيح، ولكن مجموعة بيانات القزحية هذه مبنية ببساطة بطريقة تميل خوارزميات التعلم الآلي إلى الأداء بشكل جيد للغاية.
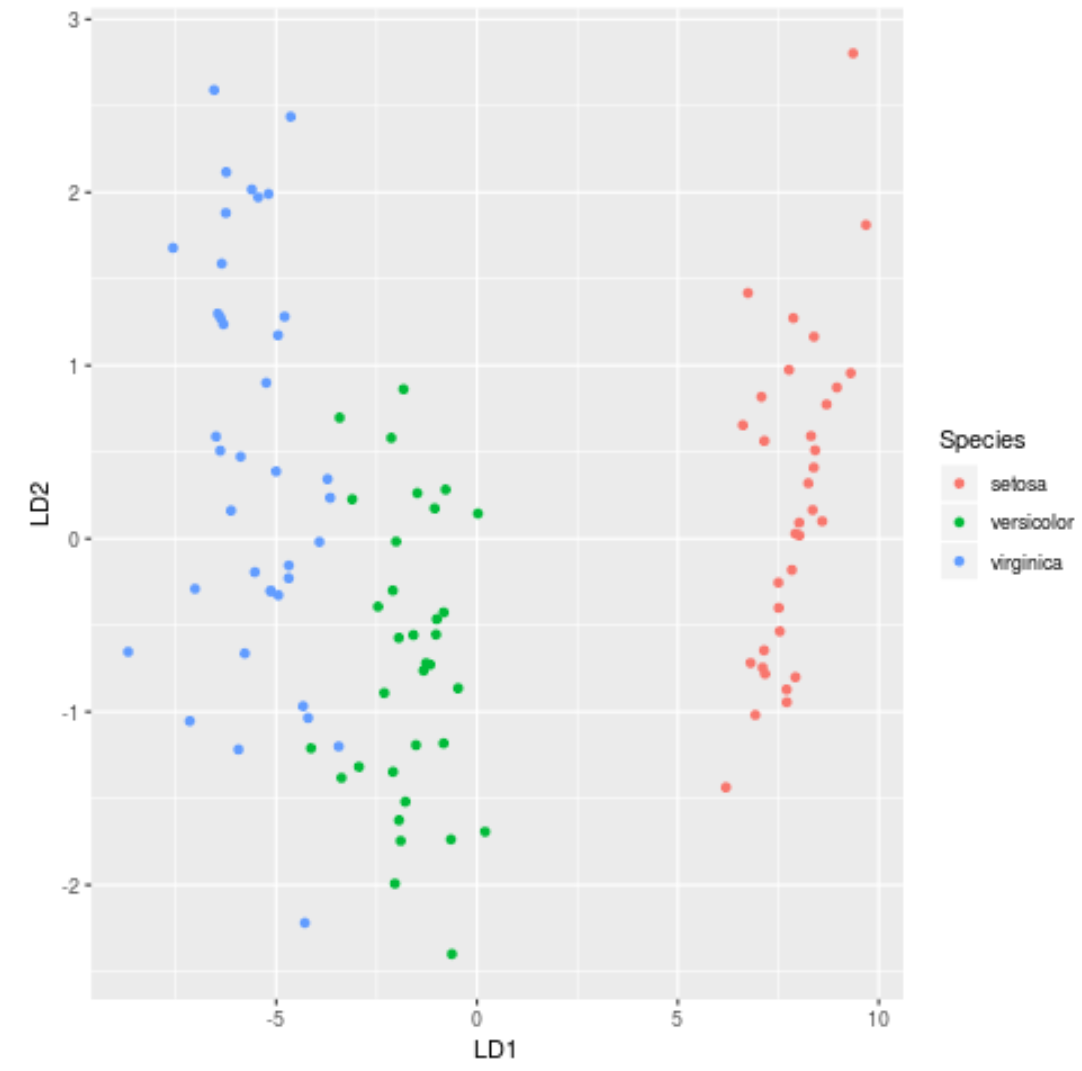
الخطوة 7: تصور النتائج
أخيرًا، يمكننا إنشاء مخطط LDA لتصور المميزات الخطية للنموذج وتصور مدى نجاحه في الفصل بين الأنواع الثلاثة المختلفة في مجموعة البيانات الخاصة بنا:
#define data to plot lda_plot <- cbind(train, predict(model)$x) #createplot ggplot(lda_plot, aes (LD1, LD2)) + geom_point( aes (color=Species))
يمكنك العثور على رمز R الكامل المستخدم في هذا البرنامج التعليمي هنا .