انحدار المكون الرئيسي في r (خطوة بخطوة)
بالنظر إلى مجموعة من متغيرات التوقع p ومتغير الاستجابة، يستخدم الانحدار الخطي المتعدد طريقة تعرف باسم المربعات الصغرى لتقليل مجموع المربعات المتبقية (RSS):
RSS = Σ(y i – ŷ i ) 2
ذهب:
- Σ : رمز يوناني معناه المجموع
- y i : قيمة الاستجابة الفعلية للملاحظة رقم i
- ŷ i : قيمة الاستجابة المتوقعة بناءً على نموذج الانحدار الخطي المتعدد
ومع ذلك، عندما تكون متغيرات التوقع مترابطة بشكل كبير، يمكن أن تصبح العلاقة الخطية المتعددة مشكلة. وهذا يمكن أن يجعل تقديرات معامل النموذج غير موثوقة وتظهر تباينًا كبيرًا.
إحدى الطرق لتجنب هذه المشكلة هي استخدام انحدار المكونات الرئيسية ، والذي يجد مجموعات خطية M (تسمى “المكونات الرئيسية”) من تنبؤات p الأصلية ثم يستخدم المربعات الصغرى لتناسب نموذج الانحدار الخطي باستخدام المكونات الرئيسية كمتنبئات.
يوفر هذا البرنامج التعليمي مثالاً خطوة بخطوة لكيفية تنفيذ انحدار المكونات الرئيسية في R.
الخطوة 1: تحميل الحزم اللازمة
أسهل طريقة لإجراء انحدار المكونات الرئيسية في R هي استخدام الوظائف الموجودة في الحزمة pls .
#install pls package (if not already installed) install.packages(" pls ") load pls package library(pls)
الخطوة 2: ضبط نموذج PCR
في هذا المثال، سوف نستخدم مجموعة بيانات R المضمنة والتي تسمى mtcars والتي تحتوي على بيانات عن أنواع مختلفة من السيارات:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
في هذا المثال، سوف نقوم بتركيب نموذج انحدار المكونات الرئيسية (PCR) باستخدام hp كمتغير الاستجابة والمتغيرات التالية كمتغيرات متوقعة:
- ميلا في الغالون
- عرض
- القرف
- وزن
- com.qsec
يوضح الكود التالي كيفية ملاءمة نموذج PCR لهذه البيانات. لاحظ الحجج التالية:
- مقياس = TRUE : هذا يخبر R بأن كل متغير من متغيرات التوقع يجب أن يتم قياسه ليكون متوسطه 0 وانحراف معياري قدره 1. وهذا يضمن عدم وجود متغير متنبئ له تأثير كبير في النموذج إذا تم قياسه بوحدات مختلفة. .
- validation=”CV” : هذا يخبر R باستخدام التحقق المتقاطع k-fold لتقييم أداء النموذج. لاحظ أن هذا يستخدم k=10 طيات بشكل افتراضي. لاحظ أيضًا أنه يمكنك تحديد “LOOCV” بدلاً من ذلك لإجراء التحقق من صحة الإجازة الواحدة .
#make this example reproducible set.seed(1) #fit PCR model model <- pcr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
الخطوة 3: اختر عدد المكونات الرئيسية
بمجرد تعديل النموذج، نحتاج إلى تحديد عدد المكونات الرئيسية التي تستحق الاحتفاظ بها.
للقيام بذلك، ما عليك سوى إلقاء نظرة على اختبار جذر متوسط مربع الخطأ (اختبار RMSE) المحسوب عن طريق التحقق من صحة k-cross:
#view summary of model fitting
summary(model)
Data:
Y dimension: 32 1
Fit method: svdpc
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comp 2 comps 3 comps 4 comps 5 comps
CV 69.66 44.56 35.64 35.83 36.23 36.67
adjCV 69.66 44.44 35.27 35.43 35.80 36.20
TRAINING: % variance explained
1 comp 2 comps 3 comps 4 comps 5 comps
X 69.83 89.35 95.88 98.96 100.00
hp 62.38 81.31 81.96 81.98 82.03
هناك جدولان مثيران للاهتمام في النتيجة:
1. التحقق من الصحة: RMSEP
يخبرنا هذا الجدول باختبار RMSE المحسوب عن طريق التحقق من صحة k-fold. يمكننا أن نرى ما يلي:
- إذا استخدمنا المصطلح الأصلي فقط في النموذج، فإن قيمة RMSE للاختبار هي 69.66 .
- إذا أضفنا المكون الرئيسي الأول، ينخفض اختبار RMSE إلى 44.56.
- إذا أضفنا المكون الرئيسي الثاني، ينخفض اختبار RMSE إلى 35.64.
يمكننا أن نرى أن إضافة مكونات رئيسية إضافية يؤدي في الواقع إلى زيادة في RMSE للاختبار. وبالتالي، يبدو أنه سيكون من الأمثل استخدام مكونين رئيسيين فقط في النموذج النهائي.
2. التدريب: شرح نسبة التباين
يوضح لنا هذا الجدول نسبة التباين في متغير الاستجابة الموضح بالمكونات الرئيسية. يمكننا أن نرى ما يلي:
- وباستخدام المكون الرئيسي الأول فقط يمكننا تفسير 69.83% من التباين في متغير الاستجابة.
- وبإضافة المكون الرئيسي الثاني يمكننا تفسير 89.35% من التباين في متغير الاستجابة.
لاحظ أننا سنظل قادرين على تفسير المزيد من التباين باستخدام المزيد من المكونات الرئيسية، ولكن يمكننا أن نرى أن إضافة أكثر من مكونين رئيسيين لا يؤدي في الواقع إلى زيادة النسبة المئوية للتباين الموضح كثيرًا.
يمكننا أيضًا تصور اختبار RMSE (جنبًا إلى جنب مع اختبار MSE واختبار R-squared) كدالة لعدد المكونات الرئيسية باستخدام وظيفة validationplot() .
#visualize cross-validation plots
validationplot(model)
validationplot(model, val.type="MSEP")
validationplot(model, val.type="R2")
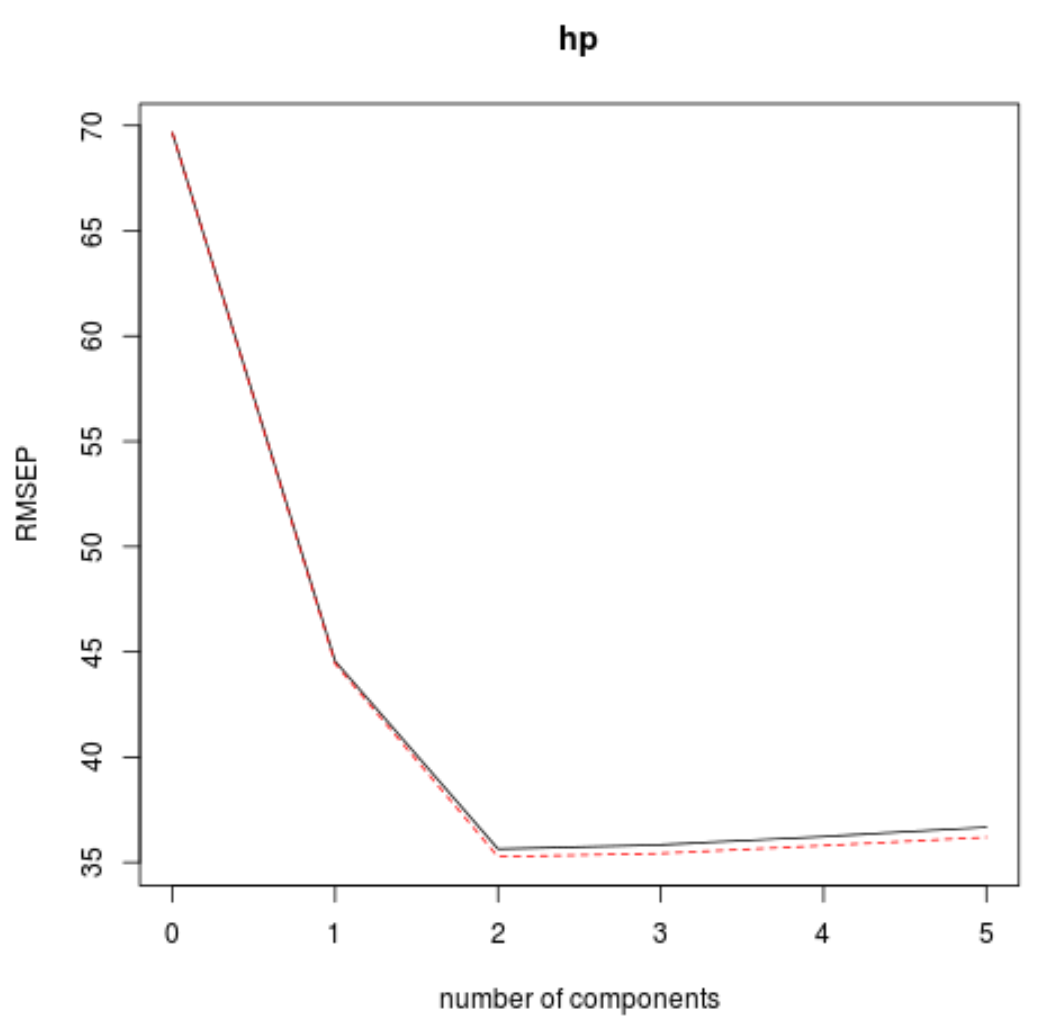
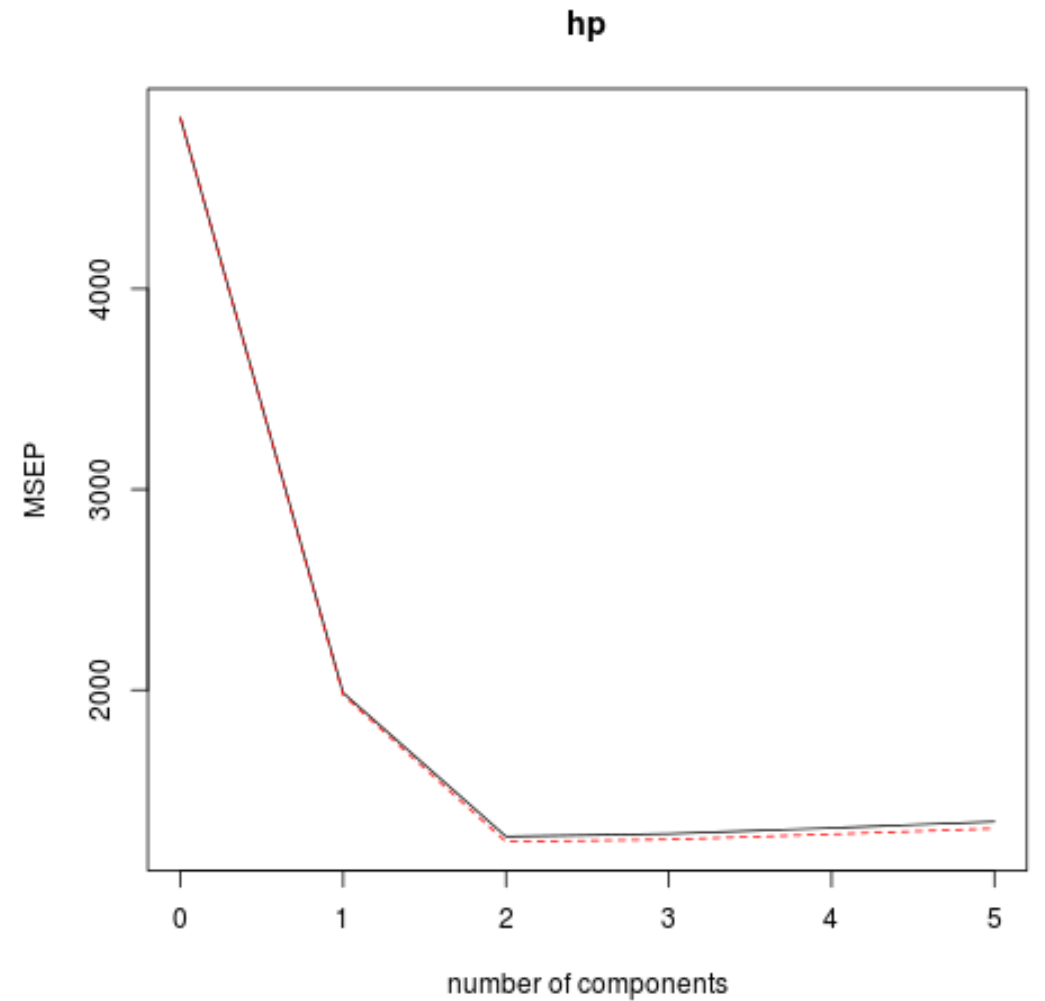
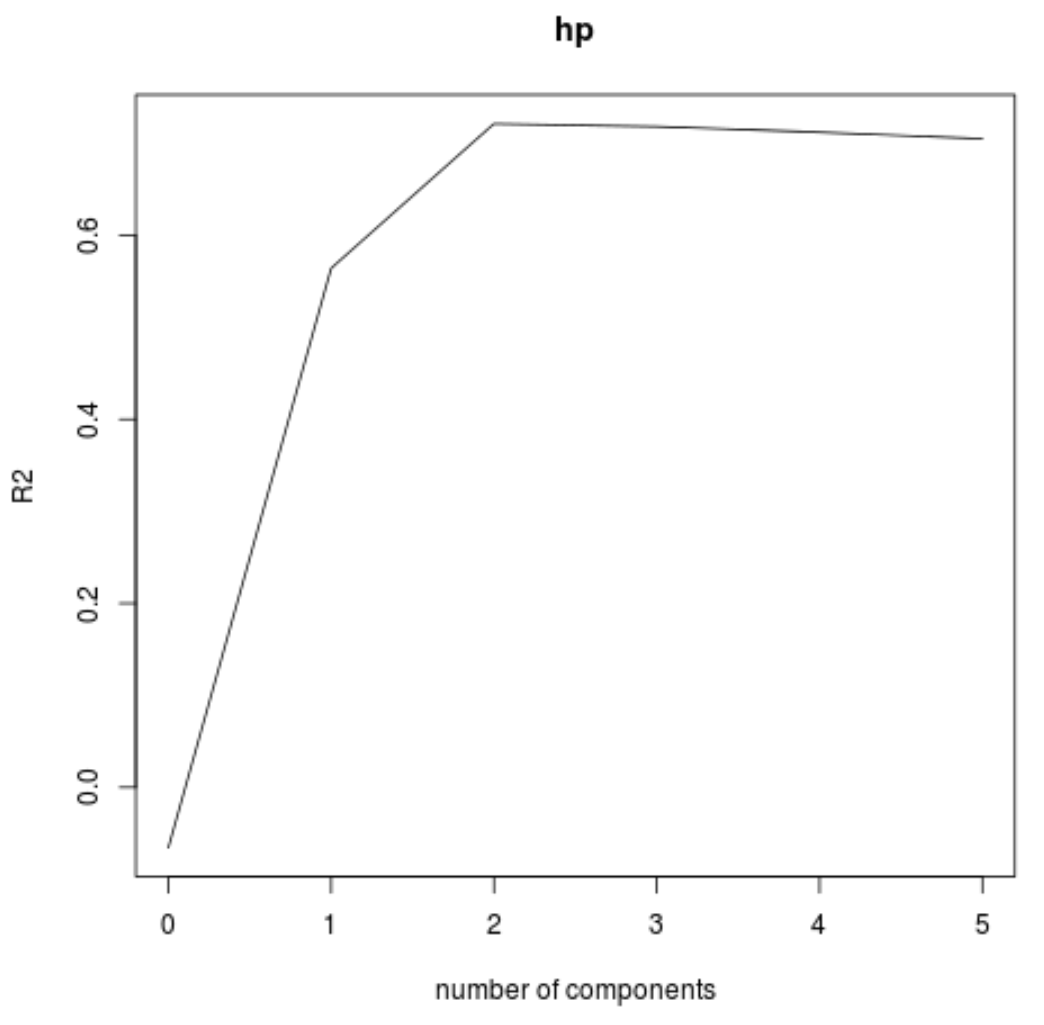
في كل رسم بياني، يمكننا أن نرى أن ملاءمة النموذج تتحسن بإضافة مكونين رئيسيين، ولكنه يميل إلى التدهور عندما نضيف المزيد من المكونات الرئيسية.
وبالتالي، فإن النموذج الأمثل يشمل فقط المكونين الرئيسيين الأولين.
الخطوة 4: استخدم النموذج النهائي لعمل تنبؤات
يمكننا استخدام نموذج PCR النهائي المكون من عنصرين رئيسيين للتنبؤ بالملاحظات الجديدة.
يوضح الكود التالي كيفية تقسيم مجموعة البيانات الأصلية إلى مجموعة تدريب واختبار واستخدام نموذج PCR مع مكونين رئيسيين لعمل تنبؤات على مجموعة الاختبار.
#define training and testing sets train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")] y_test <- mtcars[26: nrow (mtcars), c("hp")] test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")] #use model to make predictions on a test set model <- pcr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ") pcr_pred <- predict(model, test, ncomp= 2 ) #calculate RMSE sqrt ( mean ((pcr_pred - y_test)^2)) [1] 56.86549
نرى أن RMSE للاختبار تبين أنه 56.86549 . هذا هو متوسط الانحراف بين قيمة حصان المتوقعة وقيمة حصان المرصودة لملاحظات مجموعة الاختبار.
يمكن العثور هنا على الاستخدام الكامل لرمز R في هذا المثال.