如何在 r 中创建随机森林(逐步)
当一组预测变量和响应变量之间的关系非常复杂时,我们经常使用非线性方法对它们之间的关系进行建模。
其中一种方法是构建 决策树。然而,使用单个决策树的缺点是它往往会遭受 高方差的影响。
也就是说,如果我们将数据集分成两半并将决策树应用于两半,结果可能会非常不同。
我们可以用来减少单个决策树方差的一种方法是构建随机森林模型,其工作原理如下:
1.从原始数据集中获取b 个引导样本。
2.为每个引导样本创建决策树。
- 构建树时,每次考虑拆分时,仅将m 个预测变量的随机样本视为从p 个预测变量的完整集合中进行拆分的候选者。一般情况下,我们选择m等于√p 。
3.对每棵树的预测进行平均以获得最终模型。
事实证明,随机森林往往会产生比单个决策树甚至袋装模型更准确的模型。
本教程提供了如何在 R 中为数据集创建随机森林模型的分步示例。
第1步:加载必要的包
首先,我们将加载本示例所需的包。对于这个简单的例子,我们只需要一个包:
library (randomForest)
第2步:调整随机森林模型
在此示例中,我们将使用名为“空气质量”的内置 R 数据集,其中包含纽约市 153 天的空气质量测量值。
#view structure of air quality dataset str(airquality) 'data.frame': 153 obs. of 6 variables: $ Ozone: int 41 36 12 18 NA 28 23 19 8 NA ... $Solar.R: int 190 118 149 313 NA NA 299 99 19 194 ... $ Wind: num 7.4 8 12.6 11.5 14.3 14.9 8.6 13.8 20.1 8.6 ... $ Temp: int 67 72 74 62 56 66 65 59 61 69 ... $Month: int 5 5 5 5 5 5 5 5 5 5 ... $Day: int 1 2 3 4 5 6 7 8 9 10 ... #find number of rows with missing values sum(! complete . cases (airquality)) [1] 42
该数据集有 42 行存在缺失值。因此,在拟合随机森林模型之前,我们将用列中位数填充每列中的缺失值:
#replace NAs with column medians for (i in 1: ncol (air quality)) { airquality[,i][ is . na (airquality[, i])] <- median (airquality[, i], na . rm = TRUE ) }
相关: 如何在R中估算缺失值
以下代码演示了如何使用randomForest包中的randomForest()函数在 R 中拟合随机森林模型。
#make this example reproducible set.seed(1) #fit the random forest model model <- randomForest( formula = Ozone ~ ., data = airquality ) #display fitted model model Call: randomForest(formula = Ozone ~ ., data = airquality) Type of random forest: regression Number of trees: 500 No. of variables tried at each split: 1 Mean of squared residuals: 327.0914 % Var explained: 61 #find number of trees that produce lowest test MSE which.min(model$mse) [1] 82 #find RMSE of best model sqrt(model$mse[ which . min (model$mse)]) [1] 17.64392
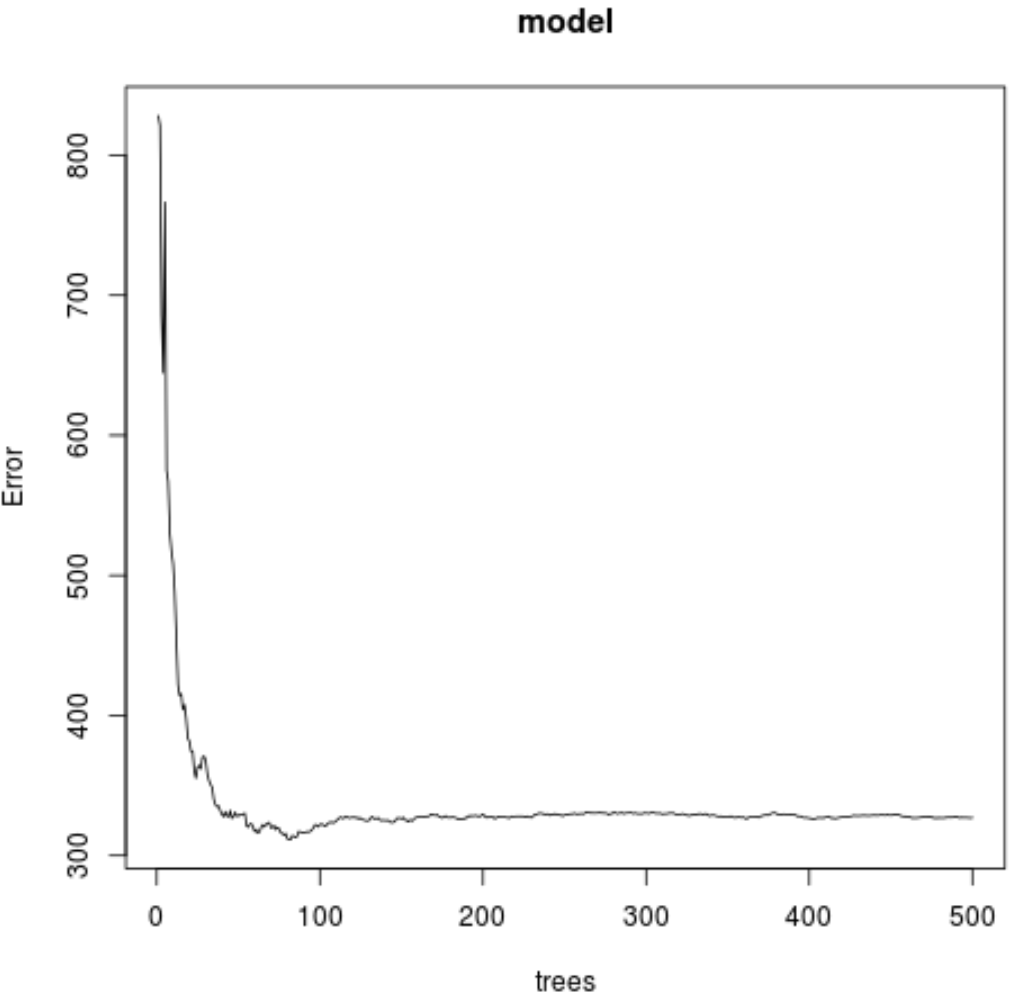
从结果中,我们可以看到产生最低测试均方误差 (MSE) 的模型使用了82棵树。
我们还可以看到该模型的均方根误差为17.64392 。我们可以将其视为臭氧预测值与实际观测值之间的平均差异。
我们还可以使用以下代码根据所使用的树的数量生成 MSE 测试图:
#plot the MSE test by number of trees
plot(model)
我们可以使用varImpPlot()函数创建一个图,显示最终模型中每个预测变量的重要性:
#produce variable importance plot
varImpPlot(model)
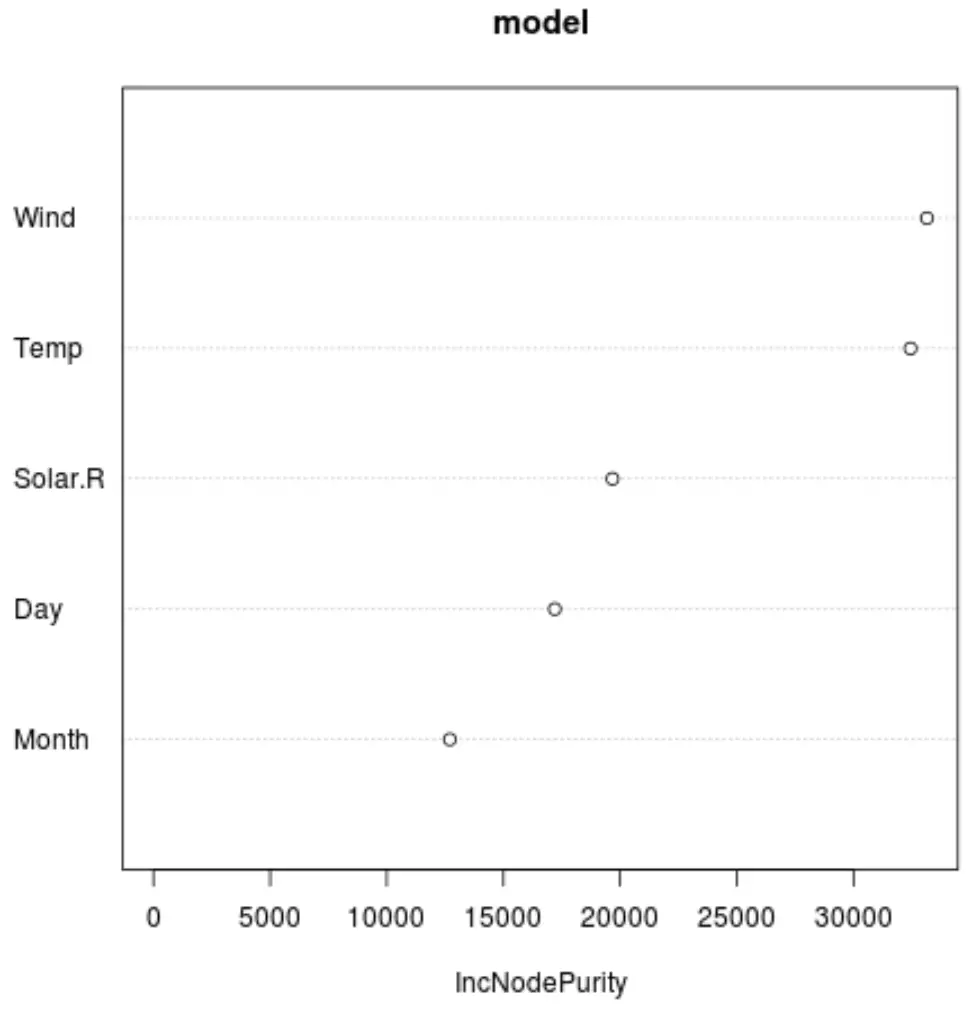
x 轴显示回归树节点纯度的平均增量,作为 y 轴上显示的不同预测变量的分割函数。
从图中我们可以看到Wind是最重要的预测变量,紧随其后的是Temp 。
第三步:调整模型
默认情况下, randomForest()函数使用 500 棵树和(总预测变量/3)随机选择的预测变量作为每次分割的潜在候选变量。我们可以使用tuneRF()函数调整这些参数。
以下代码展示了如何使用以下规范找到最佳模型:
- ntreeTry:要构建的树的数量。
- mtryStart:每次划分时要考虑的预测变量的初始数量。
- stepFactor:增加因子,直到估计的袋外误差停止改善一定量。
- 改善:为了继续增加步长因子,必须改善袋子出口误差的量。
model_tuned <- tuneRF(
x=airquality[,-1], #define predictor variables
y=airquality$Ozone, #define response variable
ntreeTry= 500 ,
mtryStart= 4 ,
stepFactor= 1.5 ,
improve= 0.01 ,
trace= FALSE #don't show real-time progress
)
此函数生成以下图,其中显示在 x 轴上构建树时每次分割使用的预测变量的数量,以及 y 轴上估计的袋外误差:
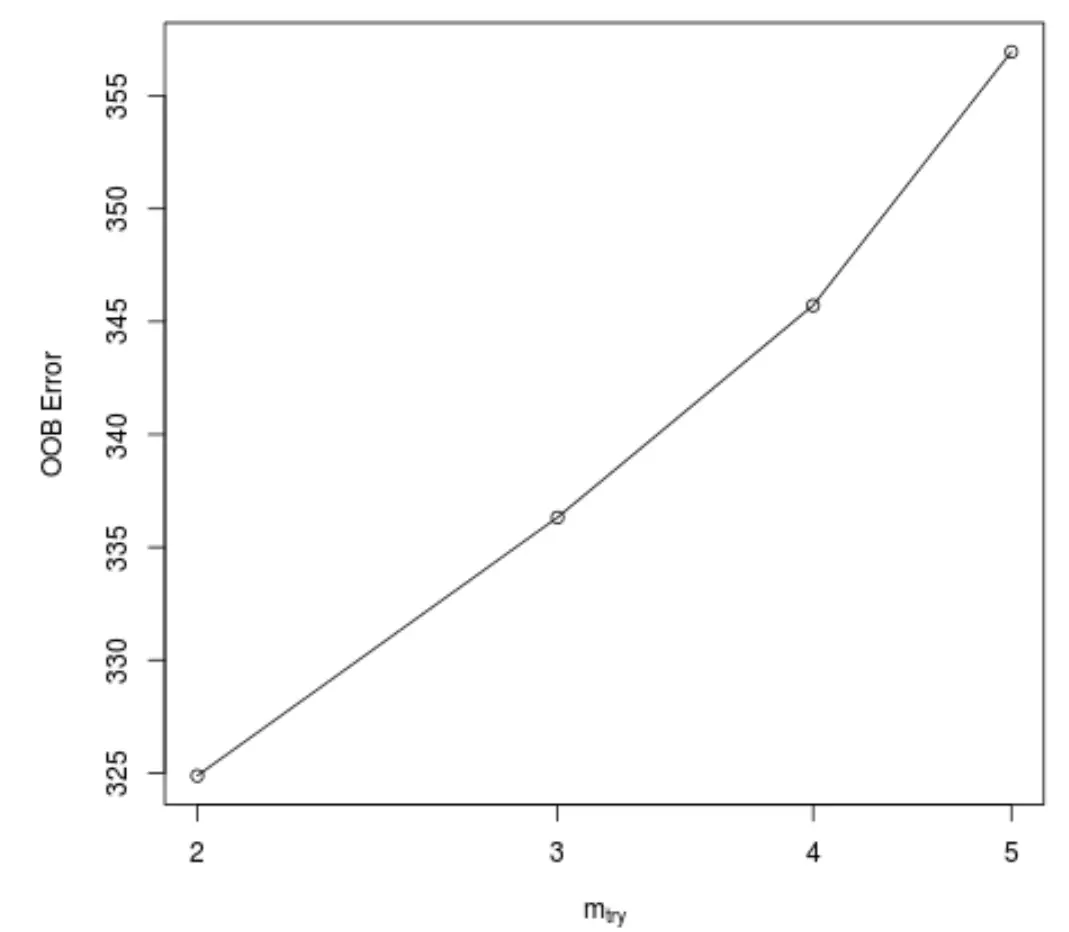
我们可以看到,在构建树时,在每次分割时使用2 个随机选择的预测变量可以获得最低的 OOB 误差。
这实际上对应于初始randomForest()函数使用的默认设置(总预测变量/3 = 6/3 = 2)。
第 4 步:使用最终模型进行预测
最后,我们可以使用调整后的随机森林模型来对新的观察结果进行预测。
#define new observation new <- data.frame(Solar.R=150, Wind=8, Temp=70, Month=5, Day=5) #use fitted bagged model to predict Ozone value of new observation predict(model, newdata=new) 27.19442
根据预测变量的值,拟合的随机森林模型预测这一天的臭氧值为27.19442 。
此示例中使用的完整 R 代码可以在此处找到。