Hauptkomponentenregression in python (schritt für schritt)
Bei einem Satz von p Prädiktorvariablen und einer Antwortvariablen verwendet die multiple lineare Regression eine Methode, die als kleinste Quadrate bekannt ist, um die verbleibende Quadratsumme (RSS) zu minimieren:
RSS = Σ(y i – ŷ i ) 2
Gold:
- Σ : Ein griechisches Symbol für Summe
- y i : der tatsächliche Antwortwert für die i-te Beobachtung
- ŷ i : Der vorhergesagte Antwortwert basierend auf dem multiplen linearen Regressionsmodell
Wenn jedoch Prädiktorvariablen stark korreliert sind, kann Multikollinearität zu einem Problem werden. Dies kann dazu führen, dass Modellkoeffizientenschätzungen unzuverlässig werden und eine hohe Varianz aufweisen.
Eine Möglichkeit, dieses Problem zu vermeiden, besteht darin, die Hauptkomponentenregression zu verwenden, die M lineare Kombinationen (sogenannte „Hauptkomponenten“) der ursprünglichen p Prädiktoren findet und dann die Methode der kleinsten Quadrate verwendet, um ein lineares Regressionsmodell unter Verwendung der Hauptkomponenten als Prädiktoren anzupassen.
Dieses Tutorial bietet ein schrittweises Beispiel für die Durchführung einer Hauptkomponentenregression in Python.
Schritt 1: Importieren Sie die erforderlichen Pakete
Zuerst importieren wir die Pakete, die zur Durchführung der Hauptkomponentenregression (PCR) in Python erforderlich sind:
import numpy as np
import pandas as pd
import matplotlib. pyplot as plt
from sklearn. preprocessing import scale
from sklearn import model_selection
from sklearn. model_selection import RepeatedKFold
from sklearn.model_selection import train_test_split
from sklearn. PCA import decomposition
from sklearn. linear_model import LinearRegression
from sklearn. metrics import mean_squared_error
Schritt 2: Daten laden
Für dieses Beispiel verwenden wir einen Datensatz namens mtcars , der Informationen zu 33 verschiedenen Autos enthält. Wir werden hp als Antwortvariable und die folgenden Variablen als Prädiktoren verwenden:
- mpg
- Anzeige
- Scheisse
- Gewicht
- qsec
Der folgende Code zeigt, wie dieser Datensatz geladen und angezeigt wird:
#define URL where data is located
url = "https://raw.githubusercontent.com/Statorials/Python-Guides/main/mtcars.csv"
#read in data
data_full = pd. read_csv (url)
#select subset of data
data = data_full[["mpg", "disp", "drat", "wt", "qsec", "hp"]]
#view first six rows of data
data[0:6]
mpg disp drat wt qsec hp
0 21.0 160.0 3.90 2.620 16.46 110
1 21.0 160.0 3.90 2.875 17.02 110
2 22.8 108.0 3.85 2.320 18.61 93
3 21.4 258.0 3.08 3.215 19.44 110
4 18.7 360.0 3.15 3.440 17.02 175
5 18.1 225.0 2.76 3.460 20.22 105
Schritt 3: Passen Sie das PCR-Modell an
Der folgende Code zeigt, wie das PCR-Modell an diese Daten angepasst wird. Beachte das Folgende:
- pca.fit_transform(scale(X)) : Dies teilt Python mit, dass jede der Prädiktorvariablen so skaliert werden soll, dass sie einen Mittelwert von 0 und eine Standardabweichung von 1 aufweist. Dadurch wird sichergestellt, dass keine Prädiktorvariable einen zu großen Einfluss auf das Modell hat, wenn das kommt vor. in verschiedenen Einheiten gemessen werden.
- cv = RepeatedKFold() : Dies weist Python an, die k-fache Kreuzvalidierung zu verwenden, um die Modellleistung zu bewerten. Für dieses Beispiel wählen wir k = 10 Falten, dreimal wiederholt.
#define predictor and response variables
X = data[["mpg", "disp", "drat", "wt", "qsec"]]
y = data[["hp"]]
#scale predictor variables
pca = pca()
X_reduced = pca. fit_transform ( scale (X))
#define cross validation method
cv = RepeatedKFold(n_splits= 10 , n_repeats= 3 , random_state= 1 )
regr = LinearRegression()
mse = []
# Calculate MSE with only the intercept
score = -1*model_selection. cross_val_score (regr,
n.p. ones ((len(X_reduced),1)), y, cv=cv,
scoring=' neg_mean_squared_error '). mean ()
mse. append (score)
# Calculate MSE using cross-validation, adding one component at a time
for i in np. arange (1, 6):
score = -1*model_selection. cross_val_score (regr,
X_reduced[:,:i], y, cv=cv, scoring=' neg_mean_squared_error '). mean ()
mse. append (score)
# Plot cross-validation results
plt. plot (mse)
plt. xlabel ('Number of Principal Components')
plt. ylabel ('MSE')
plt. title ('hp')
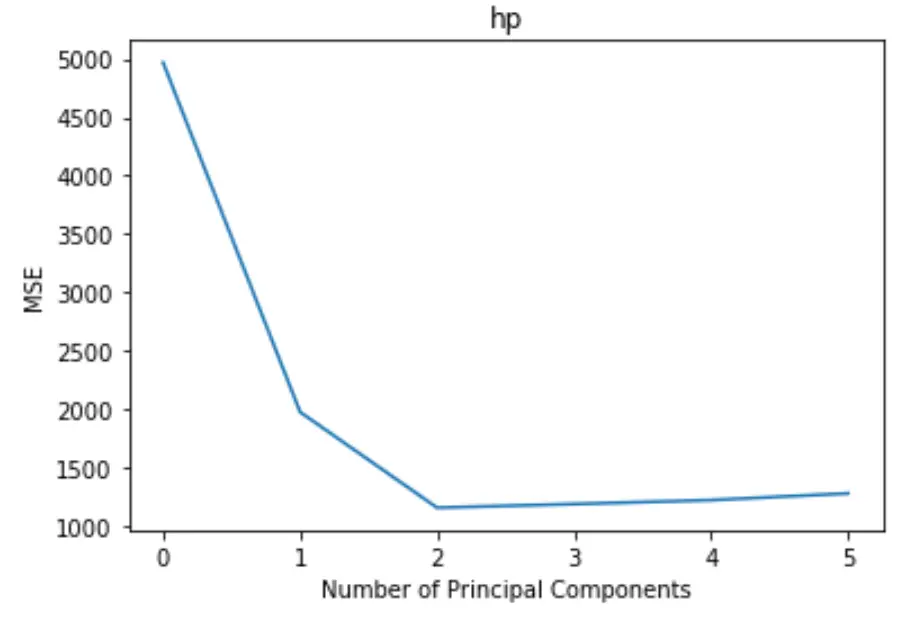
Das Diagramm zeigt die Anzahl der Hauptkomponenten entlang der x-Achse und den MSE-Test (mittlerer quadratischer Fehler) entlang der y-Achse.
Aus der Grafik können wir ersehen, dass der MSE des Tests durch das Hinzufügen von zwei Hauptkomponenten abnimmt, aber zu steigen beginnt, wenn wir mehr als zwei Hauptkomponenten hinzufügen.
Somit umfasst das optimale Modell nur die ersten beiden Hauptkomponenten.
Wir können auch den folgenden Code verwenden, um den Prozentsatz der Varianz in der Antwortvariablen zu berechnen, indem wir jede Hauptkomponente zum Modell hinzufügen:
n.p. cumsum (np. round (pca. explained_variance_ratio_ , decimals= 4 )* 100 )
array([69.83, 89.35, 95.88, 98.95, 99.99])
Wir können Folgendes sehen:
- Wenn wir nur die erste Hauptkomponente verwenden, können wir 69,83 % der Variation der Antwortvariablen erklären.
- Durch Hinzufügen der zweiten Hauptkomponente können wir 89,35 % der Variation der Antwortvariablen erklären.
Beachten Sie, dass wir immer noch in der Lage sein werden, eine größere Varianz zu erklären, indem wir mehr Hauptkomponenten verwenden, aber wir können sehen, dass das Hinzufügen von mehr als zwei Hauptkomponenten den Prozentsatz der erklärten Varianz tatsächlich nicht wesentlich erhöht.
Schritt 4: Verwenden Sie das endgültige Modell, um Vorhersagen zu treffen
Wir können das endgültige Zwei-Hauptkomponenten-PCR-Modell verwenden, um Vorhersagen über neue Beobachtungen zu treffen.
Der folgende Code zeigt, wie Sie den Originaldatensatz in einen Trainings- und einen Testsatz aufteilen und das PCR-Modell mit zwei Hauptkomponenten verwenden, um Vorhersagen für den Testsatz zu treffen.
#split the dataset into training (70%) and testing (30%) sets
X_train,X_test,y_train,y_test = train_test_split (X,y,test_size= 0.3 , random_state= 0 )
#scale the training and testing data
X_reduced_train = pca. fit_transform ( scale (X_train))
X_reduced_test = pca. transform ( scale (X_test))[:,:1]
#train PCR model on training data
regr = LinearRegression()
reg. fit (X_reduced_train[:,:1], y_train)
#calculate RMSE
pred = regr. predict (X_reduced_test)
n.p. sqrt ( mean_squared_error (y_test, pred))
40.2096
Wir sehen, dass der RMSE-Test 40,2096 ergibt. Dies ist die durchschnittliche Abweichung zwischen dem vorhergesagten HP- Wert und dem beobachteten HP- Wert für die Testsatzbeobachtungen.
Den vollständigen in diesem Beispiel verwendeten Python-Code finden Sie hier .