Bootstrapping in r (mit beispielen)
Bootstrapping ist eine Methode, mit der der Standardfehler jeder Statistik geschätzt und einKonfidenzintervall für die Statistik erstellt werden kann.
Der grundlegende Prozess für das Bootstrapping ist wie folgt:
- Nehmen Sie k Replikatproben mit Ersetzung aus einem bestimmten Datensatz.
- Berechnen Sie für jede Stichprobe die interessierende Statistik.
- Dies ergibt k verschiedene Schätzungen für eine bestimmte Statistik, die Sie dann verwenden können, um den Standardfehler der Statistik zu berechnen und ein Konfidenzintervall für die Statistik zu erstellen.
Wir können in R mit den folgenden Funktionen aus der Bootstrap-Bibliothek booten:
1. Bootstrap-Beispiele generieren.
boot(Daten, Statistiken, R, …)
Gold:
- Daten: ein Vektor, eine Matrix oder ein Datenblock
- Statistik: eine Funktion, die die zu initiierende(n) Statistik(en) erstellt
- A: Anzahl der Bootstrap-Wiederholungen
2. Generieren Sie ein Bootstrap-Konfidenzintervall.
boot.ci(Boot-Objekt, Konfiguration, Typ)
Gold:
- Bootobject: Ein von der Funktion boot() zurückgegebenes Objekt
- conf: Das zu berechnende Konfidenzintervall. Der Standardwert ist 0,95
- Typ: Typ des zu berechnenden Konfidenzintervalls. Zu den Optionen gehören „Standard“, „Basic“, „Stud“, „Perc“, „Bca“ und „All“ – die Standardeinstellung ist „All“.
Die folgenden Beispiele zeigen, wie Sie diese Funktionen in der Praxis nutzen können.
Beispiel 1: Bootstrap einer einzelnen Statistik
Der folgende Code zeigt, wie der Standardfehler für dasR-Quadrat eines einfachen linearen Regressionsmodells berechnet wird:
set.seed(0) library (boot) #define function to calculate R-squared rsq_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (summary(fit)$r.square) #return R-squared of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=rsq_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = rsq_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 0.7183433 0.002164339 0.06513426
Aus den Ergebnissen können wir sehen:
- Das geschätzte R-Quadrat für dieses Regressionsmodell beträgt 0,7183433 .
- Der Standardfehler für diese Schätzung beträgt 0,06513426 .
Wir können die Verteilung der Bootstrapping-Beispiele auch schnell visualisieren:
plot(reps)
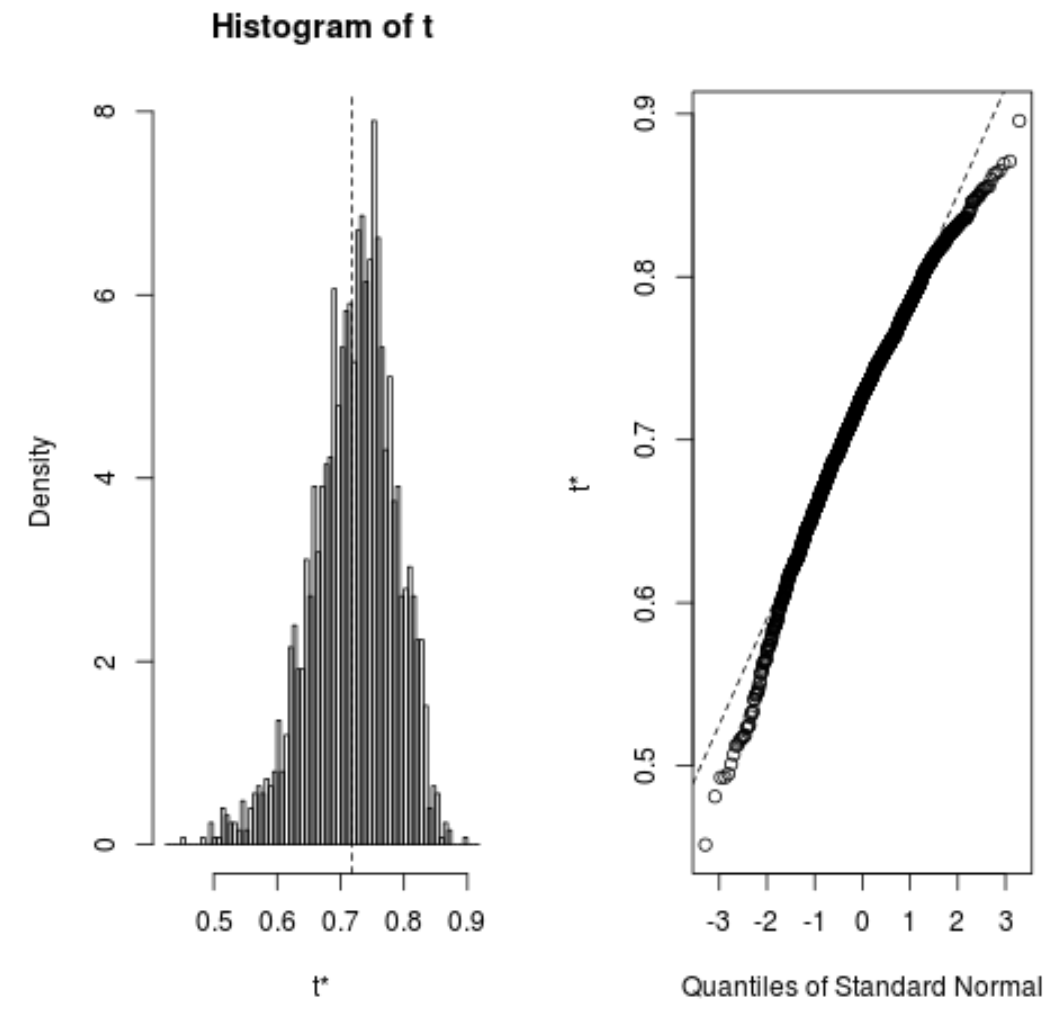
Wir können auch den folgenden Code verwenden, um das 95 %-Konfidenzintervall für das geschätzte R-Quadrat des Modells zu berechnen:
#calculate adjusted bootstrap percentile (BCa) interval boot.ci(reps, type=" bca ") CALL: boot.ci(boot.out = reps, type = "bca") Intervals: Level BCa 95% (0.5350, 0.8188) Calculations and Intervals on Original Scale
Aus dem Ergebnis können wir ersehen, dass das Bootstrapping-Konfidenzintervall von 95 % für die wahren R-Quadrat-Werte (0,5350, 0,8188) beträgt.
Beispiel 2: Bootstrap mehrerer Statistiken
Der folgende Code zeigt, wie der Standardfehler für jeden Koeffizienten in einem multiplen linearen Regressionsmodell berechnet wird:
set.seed(0) library (boot) #define function to calculate fitted regression coefficients coef_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (coef(fit)) #return coefficient estimates of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=coef_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = coef_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 29.59985476 -5.058601e-02 1.49354577 t2* -0.04121512 6.549384e-05 0.00527082
Aus den Ergebnissen können wir sehen:
- Der geschätzte Koeffizient für den Modellabschnitt beträgt 29,59985476 und der Standardfehler dieser Schätzung beträgt 1,49354577 .
- Der geschätzte Koeffizient für die Prädiktorvariable disp im Modell beträgt -0,04121512 und der Standardfehler dieser Schätzung beträgt 0,00527082 .
Wir können die Verteilung der Bootstrapping-Beispiele auch schnell visualisieren:
plot(reps, index=1) #intercept of model plot(reps, index=2) #disp predictor variable
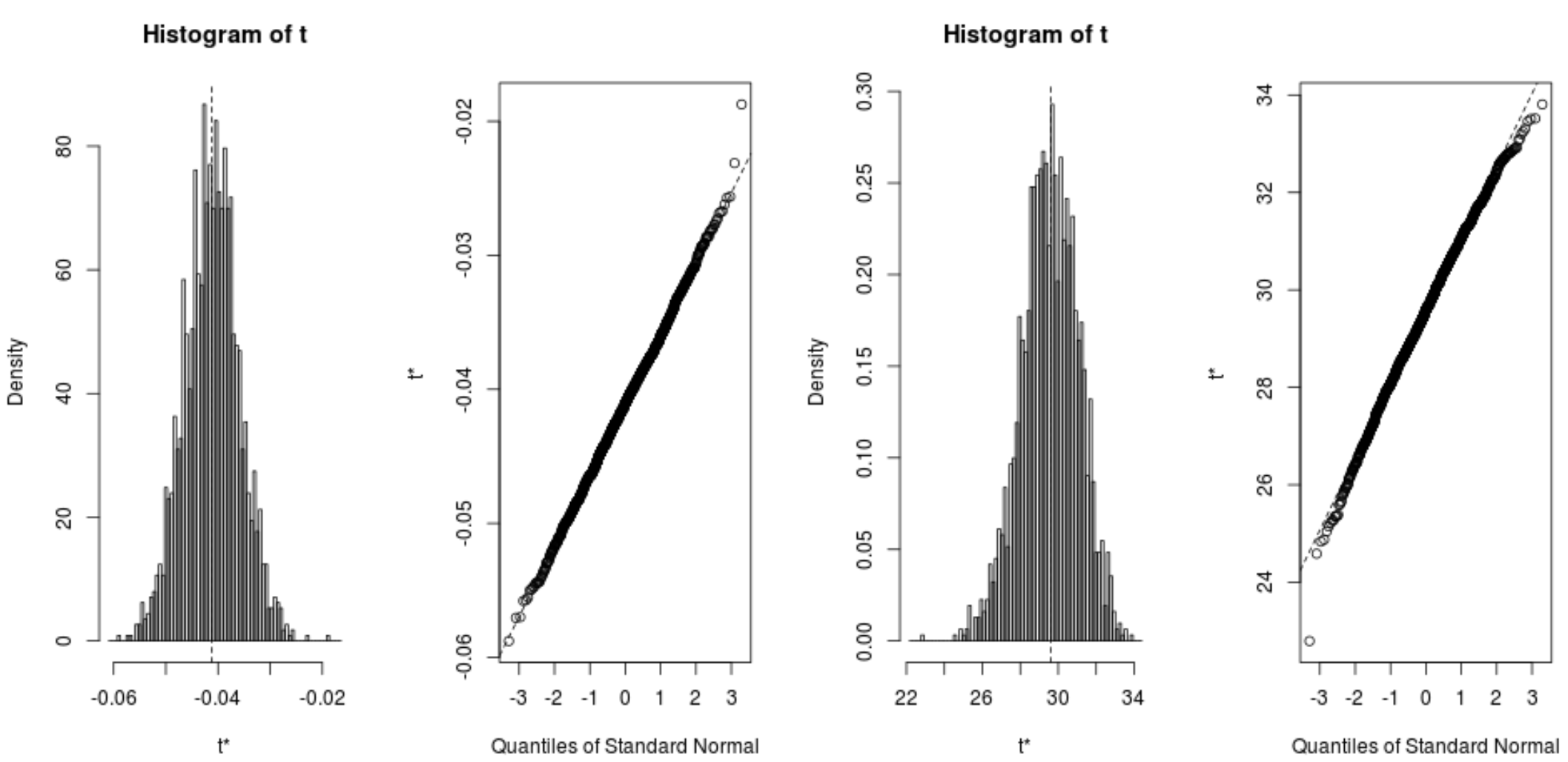
Wir können auch den folgenden Code verwenden, um die 95 %-Konfidenzintervalle für jeden Koeffizienten zu berechnen:
#calculate adjusted bootstrap percentile (BCa) intervals boot.ci(reps, type=" bca ", index=1) #intercept of model boot.ci(reps, type=" bca ", index=2) #disp predictor variable CALL: boot.ci(boot.out = reps, type = "bca", index = 1) Intervals: Level BCa 95% (26.78, 32.66) Calculations and Intervals on Original Scale BOOTSTRAP CONFIDENCE INTERVAL CALCULATIONS Based on 2000 bootstrap replicates CALL: boot.ci(boot.out = reps, type = "bca", index = 2) Intervals: Level BCa 95% (-0.0520, -0.0312) Calculations and Intervals on Original Scale
Aus den Ergebnissen können wir ersehen, dass die Bootstrapping-95-%-Konfidenzintervalle für die Modellkoeffizienten wie folgt lauten:
- IC zum Abfangen: (26,78, 32,66)
- CI für Disp : (-.0520, -.0312)
Zusätzliche Ressourcen
So führen Sie eine einfache lineare Regression in R durch
So führen Sie eine multiple lineare Regression in R durch
Einführung in Konfidenzintervalle