So berechnen sie die cook-distanz in python
Die Cook-Distanz wird verwendet, um einflussreiche Beobachtungen in einem Regressionsmodell zu identifizieren.
Die Formel für die Cook-Distanz lautet:
d i = (r i 2 / p*MSE) * (h ii / (1-h ii ) 2 )
Gold:
- r i ist der i -te Rest
- p ist die Anzahl der Koeffizienten im Regressionsmodell
- MSE ist der mittlere quadratische Fehler
- h ii ist der i-te Hebelwert
Im Wesentlichen misst die Cook-Distanz, wie stark sich alle angepassten Werte des Modells ändern, wenn die i- te Beobachtung entfernt wird.
Je größer der Wert der Cook-Distanz ist, desto einflussreicher ist eine bestimmte Beobachtung.
Als allgemeine Regel gilt, dass jede Beobachtung mit einer Cook-Distanz von mehr als 4/n (wobei n = Gesamtbeobachtungen) einen großen Einfluss hat.
Dieses Tutorial bietet ein schrittweises Beispiel für die Berechnung der Cook-Distanz für ein bestimmtes Regressionsmodell in Python.
Schritt 1: Geben Sie die Daten ein
Zuerst erstellen wir einen kleinen Datensatz, mit dem wir in Python arbeiten können:
import pandas as pd #create dataset df = pd. DataFrame ({' x ': [8, 12, 12, 13, 14, 16, 17, 22, 24, 26, 29, 30], ' y ': [41, 42, 39, 37, 35, 39, 45, 46, 39, 49, 55, 57]})
Schritt 2: Passen Sie das Regressionsmodell an
Als nächstes passen wir ein einfaches lineares Regressionsmodell an:
import statsmodels. api as sm
#define response variable
y = df[' y ']
#define explanatory variable
x = df[' x ']
#add constant to predictor variables
x = sm. add_constant (x)
#fit linear regression model
model = sm. OLS (y,x). fit ()
Schritt 3: Kochentfernung berechnen
Als nächstes berechnen wir die Cook-Distanz für jede Beobachtung im Modell:
#suppress scientific notation
import numpy as np
n.p. set_printoptions (suppress= True )
#create instance of influence
influence = model. get_influence ()
#obtain Cook's distance for each observation
cooks = influence. cooks_distance
#display Cook's distances
print (cooks)
(array([0.368, 0.061, 0.001, 0.028, 0.105, 0.022, 0.017, 0. , 0.343,
0. , 0.15 , 0.349]),
array([0.701, 0.941, 0.999, 0.973, 0.901, 0.979, 0.983, 1. , 0.718,
1. , 0.863, 0.713]))
Standardmäßig zeigt die Funktion „cooks_distance()“ für jede Beobachtung ein Array von Werten für die Cook-Distanz an, gefolgt von einem Array entsprechender p-Werte.
Zum Beispiel:
- Cook-Distanz für Beobachtung Nr. 1: 0,368 (p-Wert: 0,701)
- Cook-Distanz für Beobachtung Nr. 2: 0,061 (p-Wert: 0,941)
- Cook-Distanz für Beobachtung Nr. 3: 0,001 (p-Wert: 0,999)
Und so weiter.
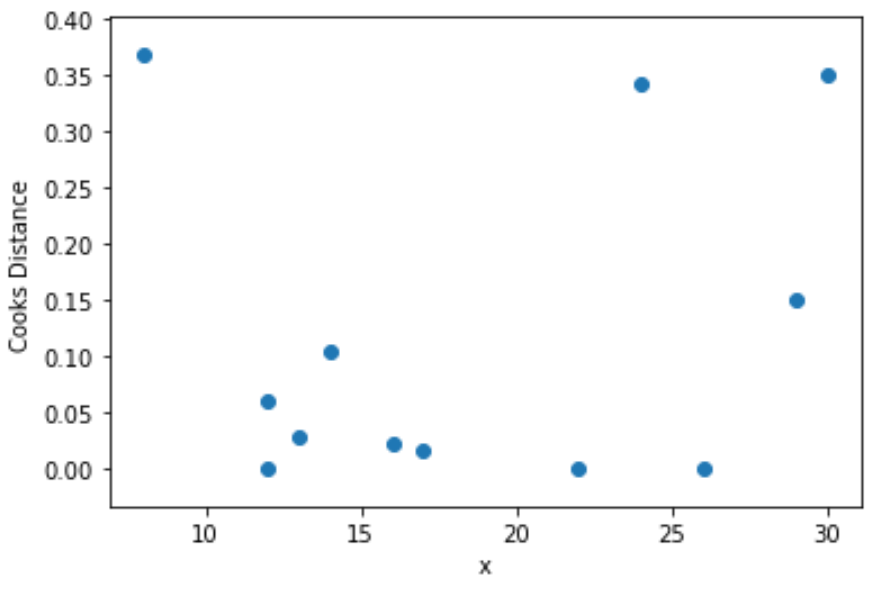
Schritt 4: Visualisieren Sie die Entfernungen des Kochs
Schließlich können wir ein Streudiagramm erstellen, um die Werte der Prädiktorvariablen als Funktion der Cook-Distanz für jede Beobachtung zu visualisieren:
import matplotlib. pyplot as plt
plt. scatter (df.x, cooks[0])
plt. xlabel (' x ')
plt. ylabel (' Cooks Distance ')
plt. show ()
Abschließende Gedanken
Es ist wichtig zu beachten, dass die Cook-Distanz verwendet werden sollte, um potenziell einflussreiche Beobachtungen zu identifizieren . Nur weil eine Beobachtung einflussreich ist, heißt das nicht, dass sie aus dem Datensatz entfernt werden sollte.
Zunächst müssen Sie sicherstellen, dass die Beobachtung nicht auf einen Dateneingabefehler oder ein anderes seltsames Ereignis zurückzuführen ist. Wenn sich herausstellt, dass es sich um einen legitimen Wert handelt, können Sie entscheiden, ob es angemessen ist, ihn zu entfernen, ihn unverändert zu lassen oder ihn einfach durch einen alternativen Wert wie den Median zu ersetzen.