So führen sie eine mehrdimensionale skalierung in r durch (mit beispiel)
In der Statistik ist die mehrdimensionale Skalierung eine Möglichkeit, die Ähnlichkeit von Beobachtungen in einem Datensatz in einem abstrakten kartesischen Raum (normalerweise 2D-Raum) zu visualisieren.
Der einfachste Weg, eine mehrdimensionale Skalierung in R durchzuführen, ist die Verwendung der integrierten Funktion cmdscale() , die die folgende grundlegende Syntax verwendet:
cmdscale(d, eig = FALSE, k = 2, …)
Gold:
- d : Eine Distanzmatrix, die im Allgemeinen von der Funktion dist() berechnet wird.
- eig : ob Eigenwerte zurückgegeben werden sollen oder nicht.
- k : Die Anzahl der Dimensionen, in denen die Daten angezeigt werden sollen. Der Standardwert ist 2 .
Das folgende Beispiel zeigt, wie Sie diese Funktion in der Praxis nutzen können.
Beispiel: Mehrdimensionale Skalierung in R
Angenommen, wir haben den folgenden Datenrahmen in R, der Informationen über verschiedene Basketballspieler enthält:
#create data frame df <- data. frame (points=c(4, 4, 6, 7, 8, 14, 16, 19, 25, 25, 28), assists=c(3, 2, 2, 5, 4, 8, 7, 6, 8, 10, 11), blocks=c(7, 3, 6, 7, 5, 8, 8, 4, 2, 2, 1), rebounds=c(4, 5, 5, 6, 5, 8, 10, 4, 3, 2, 2)) #add row names row. names (df) <- LETTERS[1:11] #view data frame df points assists blocks rebounds A 4 3 7 4 B 4 2 3 5 C 6 2 6 5 D 7 5 7 6 E 8 4 5 5 F 14 8 8 8 G 16 7 8 10 H 19 6 4 4 I 25 8 2 3 D 25 10 2 2 K 28 11 1 2
Mit dem folgenden Code können wir eine mehrdimensionale Skalierung mit der Funktion cmdscale() durchführen und die Ergebnisse im 2D-Raum visualisieren:
#calculate distance matrix
d <- dist(df)
#perform multidimensional scaling
fit <- cmdscale(d, eig= TRUE , k= 2 )
#extract (x, y) coordinates of multidimensional scaling
x <- fit$points[,1]
y <- fit$points[,2]
#create scatterplot
plot(x, y, xlab=" Coordinate 1 ", ylab=" Coordinate 2 ",
main=" Multidimensional Scaling Results ", type=" n ")
#add row names of data frame as labels
text(x, y, labels=row. names (df))
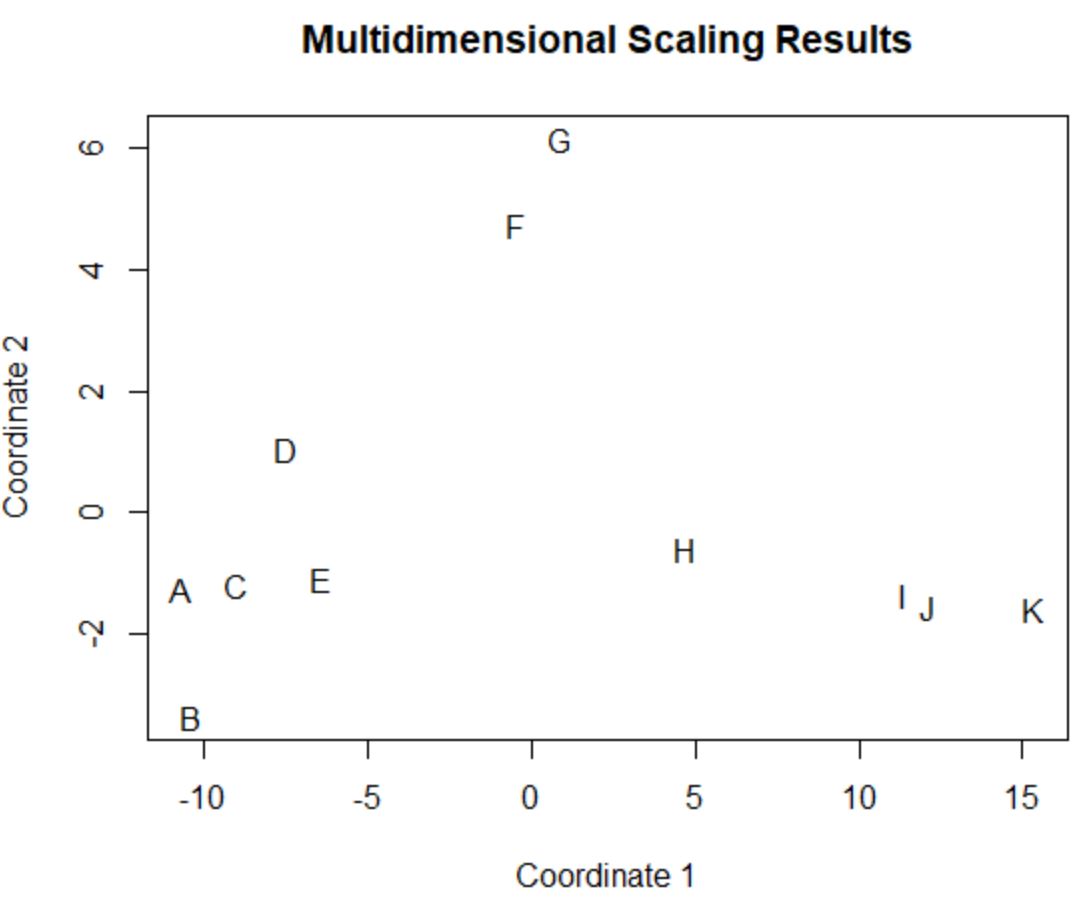
Spieler im ursprünglichen Datenrahmen, die ähnliche Werte in den ursprünglichen vier Spalten (Punkte, Assists, Blocks und Rebounds) haben, liegen im Diagramm nahe beieinander.
Beispielsweise stehen Spieler A und C nahe beieinander. Hier sind ihre Werte aus dem ursprünglichen Datenrahmen:
#view data frame values for players A and C df[rownames(df) %in% c(' A ', ' C '), ] points assists blocks rebounds A 4 3 7 4 C 6 2 6 5
Ihre Werte für Punkte, Assists, Blocks und Rebounds sind alle ziemlich ähnlich, was erklärt, warum sie im 2D-Plot so nahe beieinander liegen.
Betrachten Sie im Gegensatz dazu die Spieler B und K , die in der Handlung weit voneinander entfernt sind.
Wenn wir uns auf ihre Werte in den Originaldaten beziehen, können wir sehen, dass sie ziemlich unterschiedlich sind:
#view data frame values for players B and K df[rownames(df) %in% c(' B ', ' K '), ] points assists blocks rebounds B 4 2 3 5 K 28 11 1 2
Das 2D-Diagramm ist also eine gute Möglichkeit, zu visualisieren, wie ähnlich sich die einzelnen Spieler über alle Variablen im Datenrahmen hinweg sind.
Spieler mit ähnlichen Statistiken werden eng beieinander gruppiert, während Spieler mit sehr unterschiedlichen Statistiken in der Handlung weiter voneinander entfernt sind.
Beachten Sie, dass Sie auch die genauen Koordinaten (x, y) jedes Spielers im Plot extrahieren können, indem Sie fit eingeben. Dabei handelt es sich um den Namen der Variablen, in der wir die Ergebnisse der Funktion cmdscale() gespeichert haben:
#view (x, y) coordinates of points in the plot
fit
[,1] [,2]
A -10.6617577 -1.2511291
B -10.3858237 -3.3450473
C -9.0330408 -1.1968116
D -7.4905743 1.0578445
E -6.4021114 -1.0743669
F -0.4618426 4.7392534
G 0.8850934 6.1460850
H 4.7352436 -0.6004609
I 11.3793381 -1.3563398
J 12.0844168 -1.5494108
K 15.3510585 -1.5696166
Zusätzliche Ressourcen
In den folgenden Tutorials wird erläutert, wie Sie andere häufige Aufgaben in R ausführen:
So normalisieren Sie Daten in R
Anleitung zum Rechenzentrum in R
So entfernen Sie Ausreißer in R