आर में के-मेडोइड्स: चरण-दर-चरण उदाहरण
क्लस्टरिंग एक मशीन लर्निंग तकनीक है जो डेटा सेट के भीतर समूहों या अवलोकनों के समूहों को खोजने का प्रयास करती है।
लक्ष्य ऐसे समूहों को ढूंढना है कि प्रत्येक क्लस्टर के भीतर अवलोकन एक-दूसरे के समान हों, जबकि विभिन्न समूहों में अवलोकन एक-दूसरे से काफी भिन्न हों।
क्लस्टरिंग बिना पर्यवेक्षित शिक्षण का एक रूप है क्योंकि हम केवल प्रतिक्रिया चर के मूल्य की भविष्यवाणी करने के बजाय डेटा सेट के भीतर संरचना खोजने की कोशिश कर रहे हैं।
क्लस्टरिंग का उपयोग अक्सर विपणन में किया जाता है जब व्यवसायों के पास जानकारी तक पहुंच होती है जैसे:
- घरेलू आय
- परिवार का आकार
- घर के मुखिया का पेशा
- निकटतम शहरी क्षेत्र से दूरी
जब यह जानकारी उपलब्ध होती है, तो क्लस्टरिंग का उपयोग उन घरों की पहचान करने के लिए किया जा सकता है जो समान हैं और कुछ उत्पादों को खरीदने या एक निश्चित प्रकार के विज्ञापन पर बेहतर प्रतिक्रिया देने की अधिक संभावना हो सकती है।
क्लस्टरिंग के सबसे सामान्य रूपों में से एक को के-मीन्स क्लस्टरिंग के रूप में जाना जाता है।
दुर्भाग्य से, यह विधि आउटलेर्स से प्रभावित हो सकती है, यही कारण है कि अक्सर इस्तेमाल किया जाने वाला विकल्प के-मेडोइड्स क्लस्टरिंग है।
K-मेडोइड्स क्लस्टरिंग क्या है?
K-मेडोइड्स क्लस्टरिंग एक ऐसी तकनीक है जिसमें हम प्रत्येक अवलोकन को डेटासेट में K क्लस्टरों में से एक में रखते हैं।
अंतिम लक्ष्य K क्लस्टर बनाना है जिसमें प्रत्येक क्लस्टर के भीतर अवलोकन एक-दूसरे के समान होते हैं जबकि विभिन्न समूहों में अवलोकन एक-दूसरे से काफी भिन्न होते हैं।
व्यवहार में, हम K-मीन्स क्लस्टरिंग करने के लिए निम्नलिखित चरणों का उपयोग करते हैं:
1. K के लिए एक मान चुनें.
- सबसे पहले, हमें यह तय करना होगा कि हम डेटा में कितने समूहों की पहचान करना चाहते हैं। अक्सर हमें K के लिए कई अलग-अलग मानों का परीक्षण करने और परिणामों का विश्लेषण करने की आवश्यकता होती है ताकि यह देखा जा सके कि किसी समस्या के लिए कौन से क्लस्टर सबसे अधिक उपयुक्त लगते हैं।
2. 1 से K तक, प्रत्येक अवलोकन को यादृच्छिक रूप से प्रारंभिक क्लस्टर में निर्दिष्ट करें।
3. निम्नलिखित प्रक्रिया तब तक करें जब तक क्लस्टर असाइनमेंट बदलना बंद न हो जाए।
- प्रत्येक K क्लस्टर के लिए, क्लस्टर के गुरुत्वाकर्षण के केंद्र की गणना करें। यह k वें क्लस्टर के अवलोकनों के लिए सुविधाओं के पी माध्यकों का वेक्टर है।
- प्रत्येक अवलोकन को निकटतम केन्द्रक वाले क्लस्टर में निर्दिष्ट करें। यहां, यूक्लिडियन दूरी का उपयोग करके निकटतम को परिभाषित किया गया है।
तकनीकी नोट:
चूँकि k-medoids साधनों के बजाय माध्यिकाओं का उपयोग करके क्लस्टर सेंट्रोइड्स की गणना करता है, यह k-मीन्स की तुलना में आउटलेर्स के लिए अधिक मजबूत होता है।
व्यवहार में, यदि डेटा सेट में कोई चरम आउटलेर नहीं हैं, तो के-मीन्स और के-मेडोइड्स समान परिणाम देंगे।
आर में के-मेडोइड्स क्लस्टरिंग
निम्नलिखित ट्यूटोरियल आर में के-मेडोइड्स क्लस्टरिंग कैसे करें इसका चरण-दर-चरण उदाहरण प्रदान करता है।
चरण 1: आवश्यक पैकेज लोड करें
सबसे पहले, हम आर में के-मेडोइड्स क्लस्टरिंग के लिए कई उपयोगी कार्यों वाले दो पैकेज लोड करेंगे।
library (factoextra) library (cluster)
चरण 2: डेटा लोड करें और तैयार करें
इस उदाहरण के लिए, हम आर में निर्मित यूएसएरेस्ट्स डेटासेट का उपयोग करेंगे, जिसमें 1973 में प्रत्येक अमेरिकी राज्य में हत्या , हमले और बलात्कार के लिए प्रति 100,000 लोगों पर गिरफ्तारियों की संख्या, साथ ही प्रत्येक राज्य की शहरी आबादी का प्रतिशत शामिल है। क्षेत्र. , अर्बनपॉप ।
निम्नलिखित कोड दिखाता है कि निम्नलिखित कैसे करें:
- यूएसएअरेस्ट डेटासेट लोड करें
- लुप्त मानों वाली सभी पंक्तियाँ हटाएँ
- डेटासेट में प्रत्येक चर को 0 के माध्य और 1 के मानक विचलन के साथ स्केल करें
#load data df <-USArrests #remove rows with missing values df <- na. omitted (df) #scale each variable to have a mean of 0 and sd of 1 df <- scale(df) #view first six rows of dataset head(df) Murder Assault UrbanPop Rape Alabama 1.24256408 0.7828393 -0.5209066 -0.003416473 Alaska 0.50786248 1.1068225 -1.2117642 2.484202941 Arizona 0.07163341 1.4788032 0.9989801 1.042878388 Arkansas 0.23234938 0.2308680 -1.0735927 -0.184916602 California 0.27826823 1.2628144 1.7589234 2.067820292 Colorado 0.02571456 0.3988593 0.8608085 1.864967207
चरण 3: समूहों की इष्टतम संख्या ज्ञात करें
आर में के-मेडॉइड क्लस्टरिंग करने के लिए, हम pam() फ़ंक्शन का उपयोग कर सकते हैं, जिसका अर्थ है “मध्यस्थों के आसपास विभाजन” और निम्नलिखित सिंटैक्स का उपयोग करता है:
pam(डेटा, k, मीट्रिक = “यूक्लिडियन”, स्टैंड = गलत)
सोना:
- डेटा: डेटासेट का नाम.
- k: समूहों की संख्या।
- मीट्रिक: दूरी की गणना करने के लिए उपयोग की जाने वाली मीट्रिक। डिफ़ॉल्ट यूक्लिडियन है लेकिन आप मैनहट्टन भी निर्दिष्ट कर सकते हैं।
- स्टैंड: डेटासेट में प्रत्येक वेरिएबल को सामान्यीकृत करना है या नहीं। मूल मूल्य गलत है।
चूँकि हम पहले से नहीं जानते कि समूहों की कौन सी संख्या इष्टतम है, हम दो अलग-अलग ग्राफ़ बनाएंगे जो हमें निर्णय लेने में मदद कर सकते हैं:
1. वर्गों के योग के सापेक्ष समूहों की संख्या
सबसे पहले, हम वर्गों की कुल संख्या बनाम समूहों की संख्या का एक प्लॉट बनाने के लिए fviz_nbclust() फ़ंक्शन का उपयोग करेंगे:
fviz_nbclust(df, pam, method = “ wss ”)
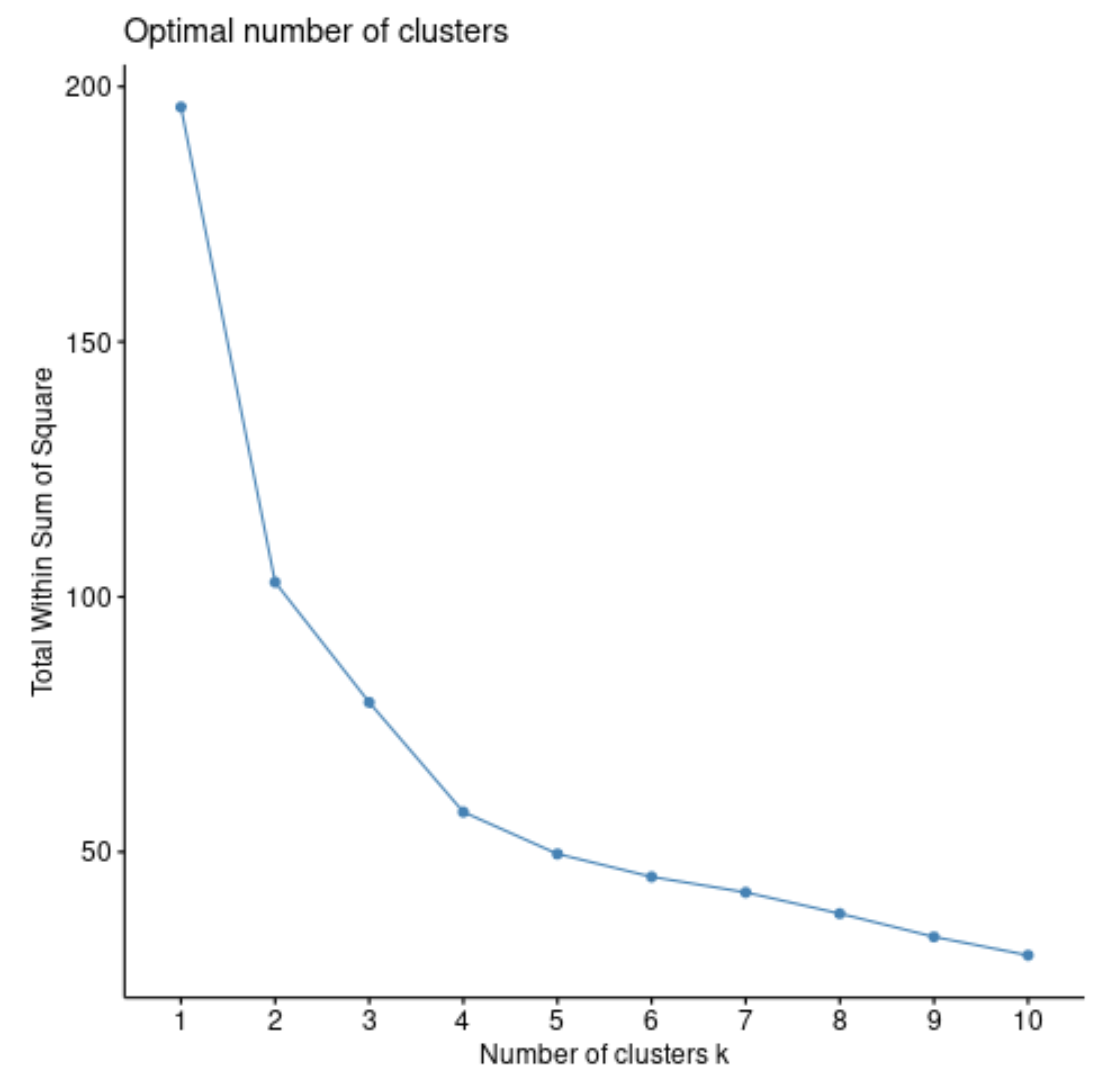
जैसे-जैसे हम समूहों की संख्या बढ़ाते हैं, वर्गों का योग आम तौर पर हमेशा बढ़ता जाएगा। इसलिए जब हम इस प्रकार का प्लॉट बनाते हैं, तो हम एक “घुटने” की तलाश में होते हैं जहां वर्गों का योग “झुकना” या समतल होना शुरू हो जाता है।
कथानक का वक्रता बिंदु आम तौर पर समूहों की इष्टतम संख्या से मेल खाता है। इस आंकड़े से परे, ओवरफिटिंग होने की संभावना है।
इस ग्राफ़ के लिए, ऐसा प्रतीत होता है कि k = 4 समूहों पर एक छोटा सा मोड़ या “मोड़” है।
2. समूहों की संख्या बनाम अंतराल आँकड़े
समूहों की इष्टतम संख्या निर्धारित करने का एक अन्य तरीका विचलन सांख्यिकी नामक एक मीट्रिक का उपयोग करना है, जो क्लस्टरिंग के बिना वितरण के लिए उनके अपेक्षित मूल्यों के साथ k के विभिन्न मूल्यों के लिए कुल इंट्रा-क्लस्टर भिन्नता की तुलना करता है।
हम क्लस्टर पैकेज से clusGap() फ़ंक्शन का उपयोग करके क्लस्टर की प्रत्येक संख्या के लिए अंतराल आंकड़ों की गणना कर सकते हैं और साथ ही fviz_gap_stat() फ़ंक्शन का उपयोग करके क्लस्टर बनाम अंतराल आंकड़ों की एक साजिश की गणना कर सकते हैं:
#calculate gap statistic based on number of clusters gap_stat <- clusGap(df, FUN = pam, K.max = 10, #max clusters to consider B = 50) #total bootstrapped iterations #plot number of clusters vs. gap statistic fviz_gap_stat(gap_stat)
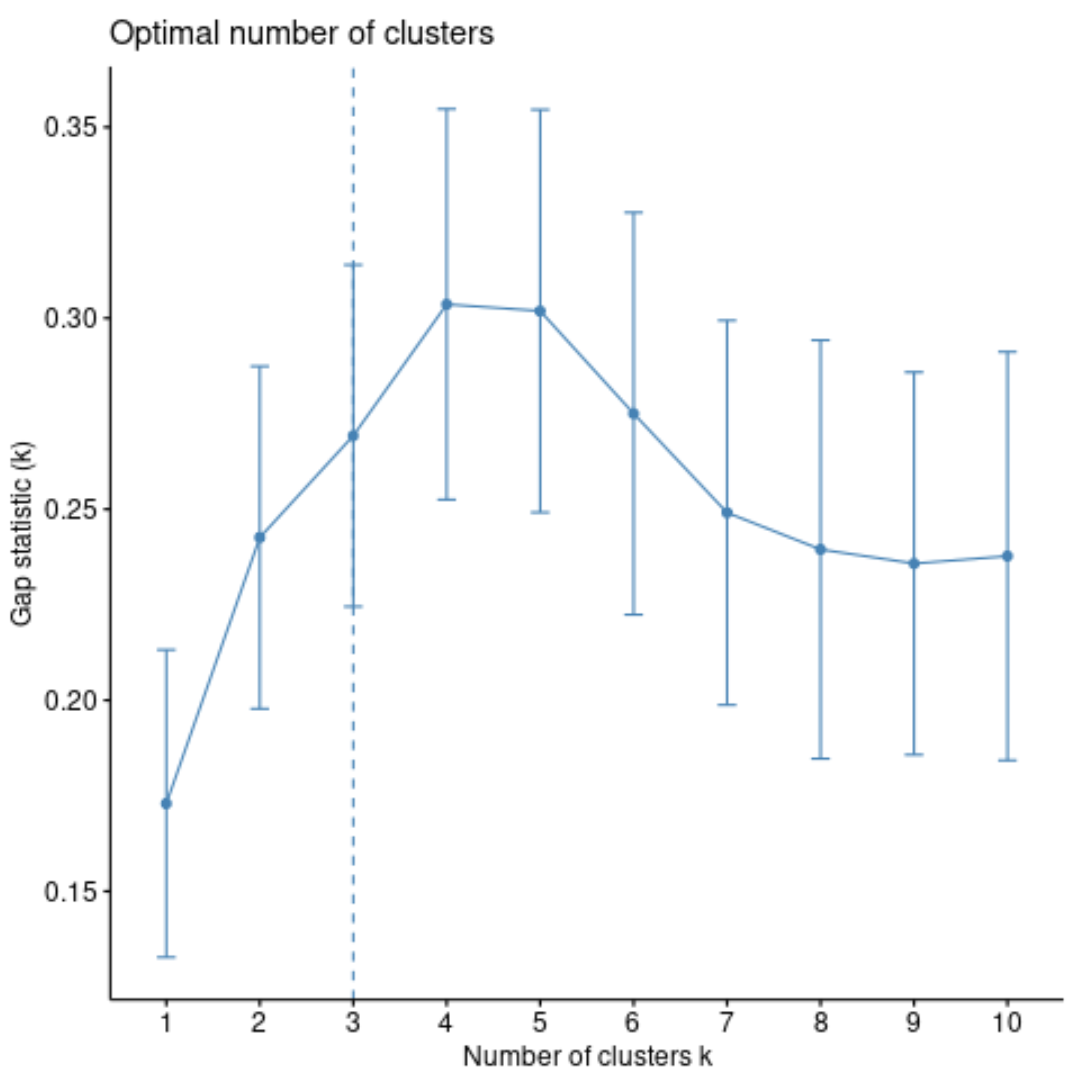
ग्राफ़ से, हम देख सकते हैं कि अंतराल आँकड़ा k = 4 समूहों पर सबसे अधिक है, जो कि हमारे द्वारा पहले इस्तेमाल की गई कोहनी विधि से मेल खाता है।
चरण 4: ऑप्टिमल K के साथ K-मेडोइड्स क्लस्टरिंग करें
अंत में, हम 4 में से k के लिए इष्टतम मान का उपयोग करके डेटासेट पर k-medoids क्लस्टरिंग कर सकते हैं:
#make this example reproducible set.seed(1) #perform k-medoids clustering with k = 4 clusters kmed <- pam(df, k = 4) #view results kmed ID Murder Assault UrbanPop Rape Alabama 1 1.2425641 0.7828393 -0.5209066 -0.003416473 Michigan 22 0.9900104 1.0108275 0.5844655 1.480613993 Oklahoma 36 -0.2727580 -0.2371077 0.1699510 -0.131534211 New Hampshire 29 -1.3059321 -1.3650491 -0.6590781 -1.252564419 Vector clustering: Alabama Alaska Arizona Arkansas California 1 2 2 1 2 Colorado Connecticut Delaware Florida Georgia 2 3 3 2 1 Hawaii Idaho Illinois Indiana Iowa 3 4 2 3 4 Kansas Kentucky Louisiana Maine Maryland 3 3 1 4 2 Massachusetts Michigan Minnesota Mississippi Missouri 3 2 4 1 3 Montana Nebraska Nevada New Hampshire New Jersey 3 3 2 4 3 New Mexico New York North Carolina North Dakota Ohio 2 2 1 4 3 Oklahoma Oregon Pennsylvania Rhode Island South Carolina 3 3 3 3 1 South Dakota Tennessee Texas Utah Vermont 4 1 2 3 4 Virginia Washington West Virginia Wisconsin Wyoming 3 3 4 4 3 Objective function: build swap 1.035116 1.027102 Available components: [1] "medoids" "id.med" "clustering" "objective" "isolation" [6] "clusinfo" "silinfo" "diss" "call" "data"
ध्यान दें कि सभी चार क्लस्टर सेंट्रोइड डेटासेट में वास्तविक अवलोकन हैं। आउटपुट के शीर्ष के पास, हम देख सकते हैं कि चार केन्द्रक निम्नलिखित अवस्थाएँ हैं:
- अलाबामा
- मिशिगन
- ओकलाहोमा
- न्यू हैम्पशायर
हम एक स्कैटरप्लॉट पर क्लस्टर की कल्पना कर सकते हैं जो fivz_cluster() फ़ंक्शन का उपयोग करके अक्षों पर पहले दो प्रमुख घटकों को प्रदर्शित करता है:
#plot results of final k-medoids model
fviz_cluster(kmed, data = df)
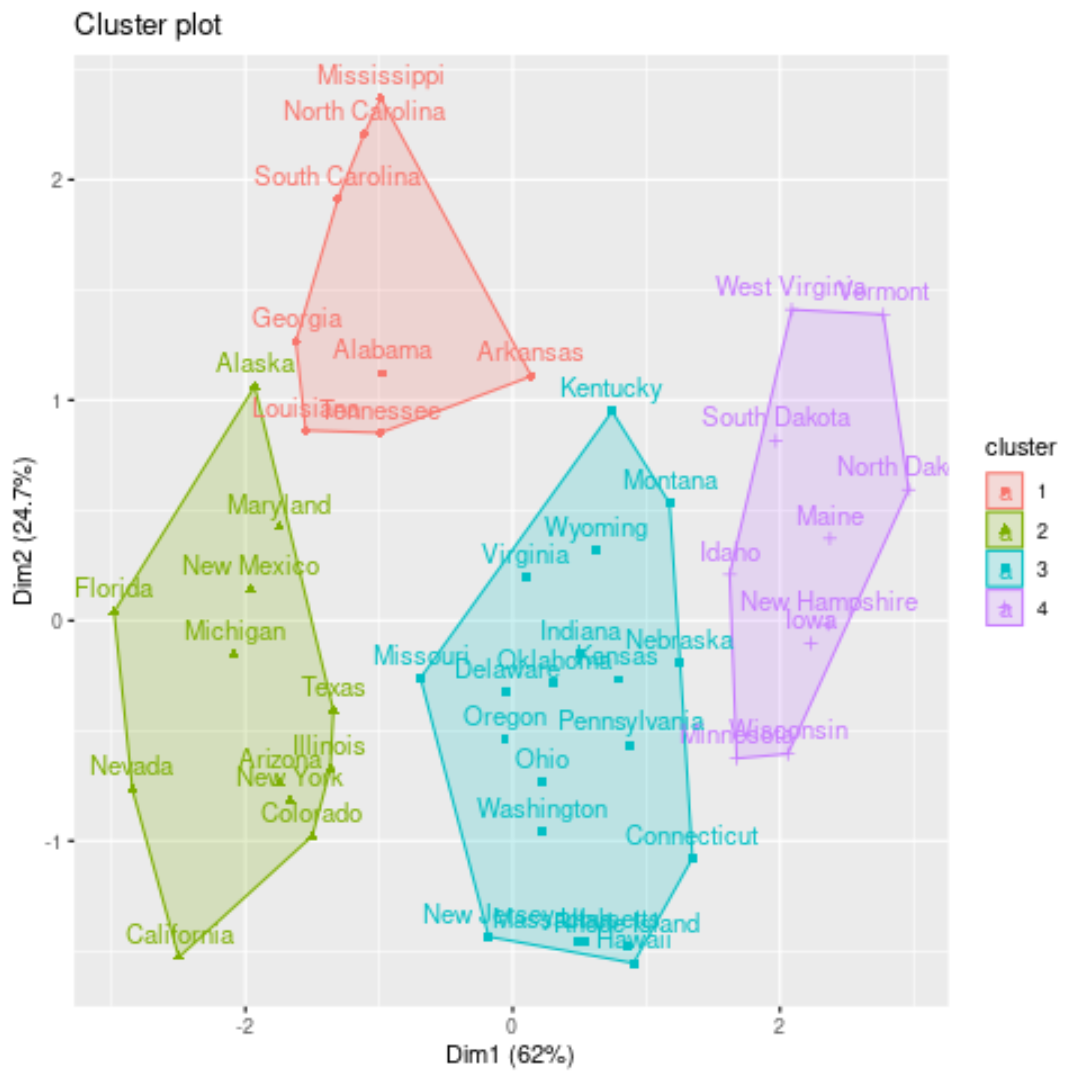
हम प्रत्येक राज्य के क्लस्टर असाइनमेंट को मूल डेटासेट में भी जोड़ सकते हैं:
#add cluster assignment to original data
final_data <- cbind(USArrests, cluster = kmed$cluster)
#view final data
head(final_data)
Murder Assault UrbanPop Rape cluster
Alabama 13.2 236 58 21.2 1
Alaska 10.0 263 48 44.5 2
Arizona 8.1 294 80 31.0 2
Arkansas 8.8 190 50 19.5 1
California 9.0 276 91 40.6 2
Colorado 7.9 204 78 38.7 2
आप इस उदाहरण में प्रयुक्त पूरा आर कोड यहां पा सकते हैं।