पायथन में बहुआयामी स्केलिंग कैसे करें
आंकड़ों में, बहुआयामी स्केलिंग एक अमूर्त कार्टेशियन स्पेस (आमतौर पर 2 डी स्पेस) में डेटा सेट में अवलोकनों की समानता को देखने का एक तरीका है।
पायथन में बहुआयामी स्केलिंग करने का सबसे आसान तरीका sklearn.manifold सबमॉड्यूल के MDS() फ़ंक्शन का उपयोग करना है।
निम्नलिखित उदाहरण दिखाता है कि व्यवहार में इस फ़ंक्शन का उपयोग कैसे करें।
उदाहरण: पायथन में बहुआयामी स्केलिंग
मान लीजिए कि हमारे पास निम्नलिखित पांडा डेटाफ़्रेम है जिसमें विभिन्न बास्केटबॉल खिलाड़ियों के बारे में जानकारी है:
import pandas as pd #create DataFrane df = pd. DataFrame ({' player ': ['A', 'B', 'C', 'D', 'E', 'F', 'G', 'H', 'I', 'J', 'K '], ' points ': [4, 4, 6, 7, 8, 14, 16, 19, 25, 25, 28], ' assists ': [3, 2, 2, 5, 4, 8, 7, 6, 8, 10, 11], ' blocks ': [7, 3, 6, 7, 5, 8, 8, 4, 2, 2, 1], ' rebounds ': [4, 5, 5, 6, 5, 8, 10, 4, 3, 2, 2]}) #set player column as index column df = df. set_index (' player ') #view Dataframe print (df) points assists blocks rebounds player A 4 3 7 4 B 4 2 3 5 C 6 2 6 5 D 7 5 7 6 E 8 4 5 5 F 14 8 8 8 G 16 7 8 10 H 19 6 4 4 I 25 8 2 3 D 25 10 2 2 K 28 11 1 2
हम sklearn.manifold मॉड्यूल के MDS() फ़ंक्शन के साथ बहुआयामी स्केलिंग करने के लिए निम्नलिखित कोड का उपयोग कर सकते हैं:
from sklearn. manifold import MDS
#perform multi-dimensional scaling
mds = MDS(random_state= 0 )
scaled_df = mds. fit_transform (df)
#view results of multi-dimensional scaling
print (scaled_df)
[[ 7.43654469 8.10247222]
[4.13193821 10.27360901]
[5.20534681 7.46919526]
[6.22323046 4.45148627]
[3.74110999 5.25591459]
[3.69073384 -2.88017811]
[3.89092087 -5.19100988]
[ -3.68593169 -3.0821144 ]
[ -9.13631889 -6.81016012]
[ -8.97898385 -8.50414387]
[-12.51859044 -9.08507097]]
मूल डेटाफ़्रेम की प्रत्येक पंक्ति को (x, y) निर्देशांक में घटा दिया गया है।
हम इन निर्देशांकों को 2डी स्पेस में देखने के लिए निम्नलिखित कोड का उपयोग कर सकते हैं:
import matplotlib.pyplot as plt #create scatterplot plt. scatter (scaled_df[:,0], scaled_df[:,1]) #add axis labels plt. xlabel (' Coordinate 1 ') plt. ylabel (' Coordinate 2 ') #add lables to each point for i, txt in enumerate( df.index ): plt. annotate (txt, (scaled_df[:,0][i]+.3, scaled_df[:,1][i])) #display scatterplot plt. show ()
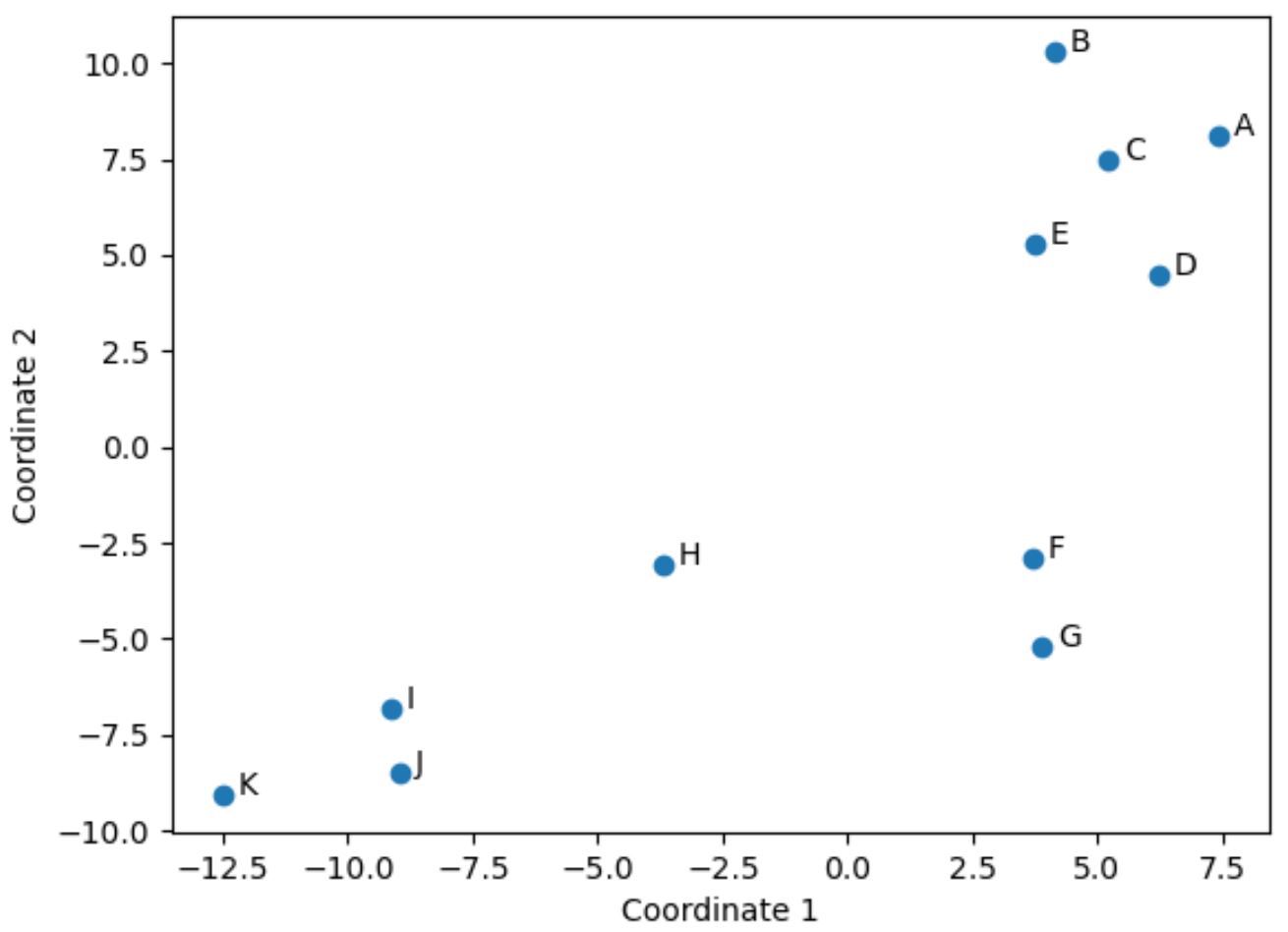
मूल डेटाफ़्रेम में खिलाड़ी जिनके मूल चार कॉलम (अंक, सहायता, ब्लॉक और रिबाउंड) में समान मान हैं, प्लॉट में एक दूसरे के करीब हैं।
उदाहरण के लिए, खिलाड़ी F और G एक दूसरे के करीब हैं। मूल डेटाफ़्रेम से उनके मान यहां दिए गए हैं:
#select rows with index labels 'F' and 'G'
df. loc [[' F ',' G ']]
points assists blocks rebounds
player
F 14 8 8 8
G 16 7 8 10
अंक, सहायता, ब्लॉक और रिबाउंड के लिए उनके मूल्य काफी समान हैं, जो बताता है कि वे 2डी प्लॉट में एक-दूसरे के इतने करीब क्यों हैं।
इसके विपरीत, खिलाड़ी बी और के पर विचार करें जो कथानक में बहुत दूर हैं।
यदि हम मूल डेटाफ़्रेम में उनके मूल्यों का उल्लेख करते हैं, तो हम देख सकते हैं कि वे काफी भिन्न हैं:
#select rows with index labels 'B' and 'K'
df. loc [[' B ',' K ']]
points assists blocks rebounds
player
B 4 2 3 5
K 28 11 1 2
इसलिए 2डी प्लॉट यह कल्पना करने का एक अच्छा तरीका है कि डेटाफ़्रेम में सभी वेरिएबल्स में प्रत्येक खिलाड़ी कितना समान है।
समान आँकड़े वाले खिलाड़ियों को एक साथ समूहीकृत किया जाता है जबकि बहुत भिन्न आँकड़ों वाले खिलाड़ी कथानक में एक-दूसरे से दूर होते हैं।
अतिरिक्त संसाधन
निम्नलिखित ट्यूटोरियल बताते हैं कि पायथन में अन्य सामान्य कार्य कैसे करें:
पायथन में डेटा को सामान्य कैसे करें
पायथन में आउटलेर्स कैसे हटाएं
पायथन में सामान्यता का परीक्षण कैसे करें