Come eseguire il bootstrap in r (con esempi)
Il bootstrap è un metodo che può essere utilizzato per stimare l’errore standard di qualsiasi statistica e produrre un intervallo di confidenza per la statistica.
Il processo di base per il bootstrap è il seguente:
- Prendi k campioni replicati con sostituzione da un dato set di dati.
- Per ciascun campione, calcolare la statistica di interesse.
- Ciò fornisce k stime diverse per una determinata statistica, che puoi quindi utilizzare per calcolare l’errore standard della statistica e creare un intervallo di confidenza per la statistica.
Possiamo eseguire il bootstrap in R utilizzando le seguenti funzioni dalla libreria bootstrap :
1. Genera campioni bootstrap.
boot(dati, statistiche, R, …)
Oro:
- dati: un vettore, una matrice o un blocco di dati
- statistica: una funzione che produce la/e statistica/e da avviare
- A: Numero di ripetizioni di bootstrap
2. Generare un intervallo di confidenza bootstrap.
boot.ci(oggetto di avvio, conf, tipo)
Oro:
- bootobject: un oggetto restituito dalla funzione boot()
- conf: l’intervallo di confidenza da calcolare. Il valore predefinito è 0,95
- type: tipo di intervallo di confidenza da calcolare. Le opzioni includono ‘standard’, ‘basic’, ‘stud’, ‘perc’, ‘bca’ e ‘all’ – il valore predefinito è ‘all’
I seguenti esempi mostrano come utilizzare queste funzioni nella pratica.
Esempio 1: bootstrap di una singola statistica
Il codice seguente mostra come calcolare l’errore standard per l’ R quadrato di un modello di regressione lineare semplice:
set.seed(0) library (boot) #define function to calculate R-squared rsq_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (summary(fit)$r.square) #return R-squared of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=rsq_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = rsq_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 0.7183433 0.002164339 0.06513426
Dai risultati possiamo vedere:
- L’R quadrato stimato per questo modello di regressione è 0,7183433 .
- L’errore standard per questa stima è 0,06513426 .
Possiamo anche visualizzare rapidamente la distribuzione dei campioni sottoposti a bootstrap:
plot(reps)
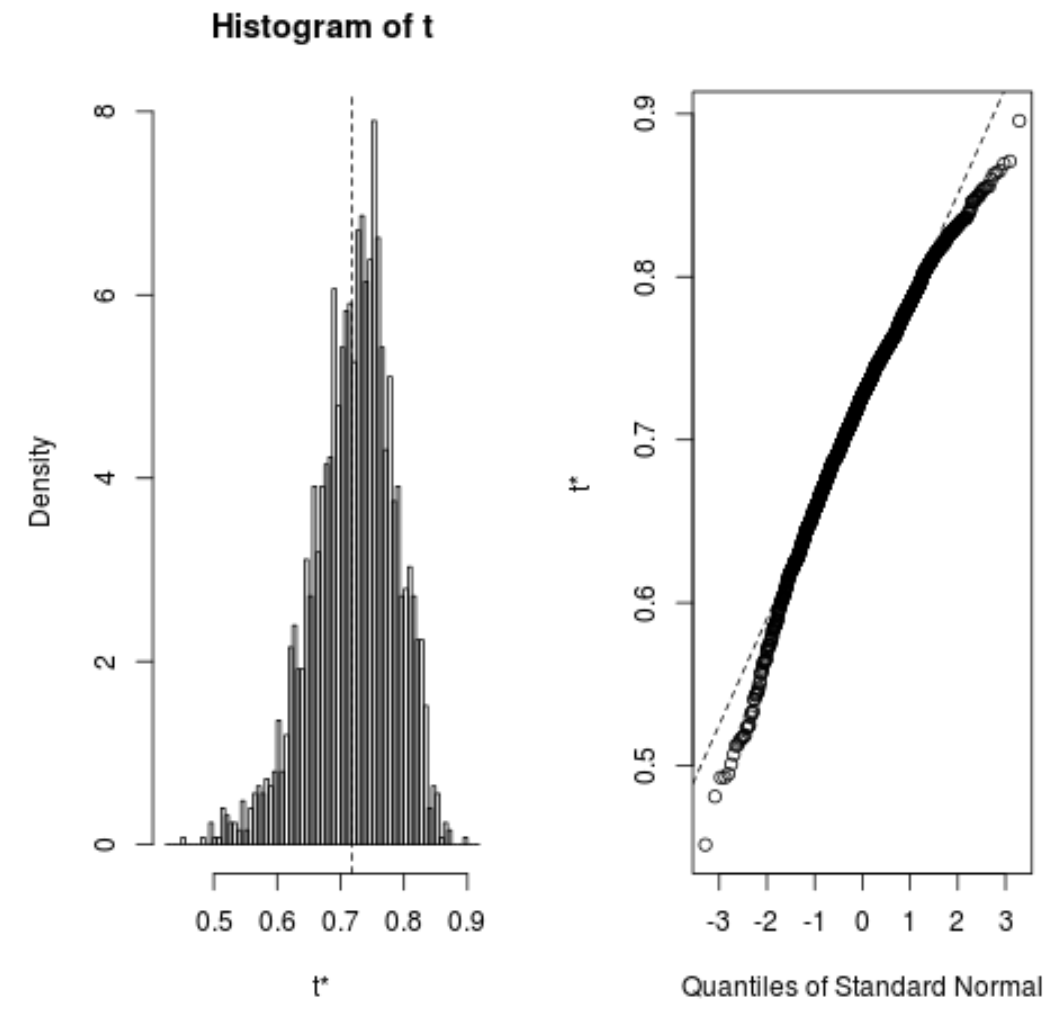
Possiamo anche utilizzare il seguente codice per calcolare l’intervallo di confidenza al 95% per l’R quadrato stimato del modello:
#calculate adjusted bootstrap percentile (BCa) interval boot.ci(reps, type=" bca ") CALL: boot.ci(boot.out = reps, type = "bca") Intervals: Level BCa 95% (0.5350, 0.8188) Calculations and Intervals on Original Scale
Dal risultato, possiamo vedere che l’intervallo di confidenza bootstrap al 95% per i valori R quadrati reali è (0,5350, 0,8188).
Esempio 2: bootstrap di più statistiche
Il codice seguente mostra come calcolare l’errore standard per ciascun coefficiente in un modello di regressione lineare multipla:
set.seed(0) library (boot) #define function to calculate fitted regression coefficients coef_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (coef(fit)) #return coefficient estimates of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=coef_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = coef_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 29.59985476 -5.058601e-02 1.49354577 t2* -0.04121512 6.549384e-05 0.00527082
Dai risultati possiamo vedere:
- Il coefficiente stimato per l’intercetta del modello è 29,59985476 e l’errore standard di questa stima è 1,49354577 .
- Il coefficiente stimato per la variabile predittiva disp nel modello è -0,04121512 e l’errore standard di questa stima è 0,00527082 .
Possiamo anche visualizzare rapidamente la distribuzione dei campioni sottoposti a bootstrap:
plot(reps, index=1) #intercept of model plot(reps, index=2) #disp predictor variable
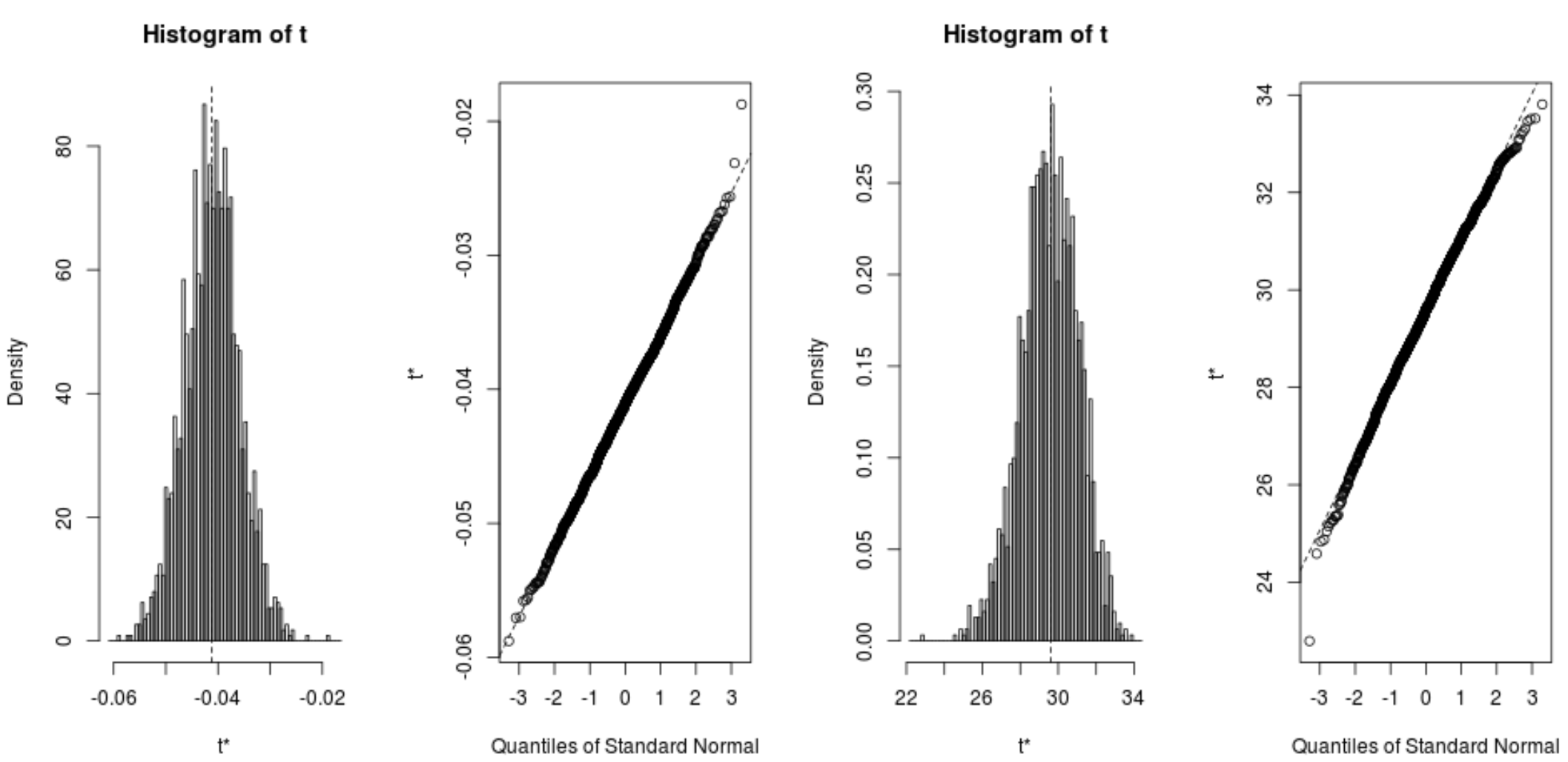
Possiamo anche utilizzare il seguente codice per calcolare gli intervalli di confidenza al 95% per ciascun coefficiente:
#calculate adjusted bootstrap percentile (BCa) intervals boot.ci(reps, type=" bca ", index=1) #intercept of model boot.ci(reps, type=" bca ", index=2) #disp predictor variable CALL: boot.ci(boot.out = reps, type = "bca", index = 1) Intervals: Level BCa 95% (26.78, 32.66) Calculations and Intervals on Original Scale BOOTSTRAP CONFIDENCE INTERVAL CALCULATIONS Based on 2000 bootstrap replicates CALL: boot.ci(boot.out = reps, type = "bca", index = 2) Intervals: Level BCa 95% (-0.0520, -0.0312) Calculations and Intervals on Original Scale
Dai risultati, possiamo vedere che gli intervalli di confidenza bootstrap al 95% per i coefficienti del modello sono i seguenti:
- IC per intercettazione: (26.78, 32.66)
- CI per visualizzazione : (-.0520, -.0312)
Risorse addizionali
Come eseguire una regressione lineare semplice in R
Come eseguire la regressione lineare multipla in R
Introduzione agli intervalli di confidenza