Come creare un grafico residuo in ggplot2 (con esempio)
I grafici dei residui vengono utilizzati per valutare se i residui di un modello di regressione sono distribuiti normalmente e se presentano o meno eteroschedasticità .
Per creare una trama residua in ggplot2, puoi utilizzare la seguente sintassi di base:
library (ggplot2) ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 )
L’esempio seguente mostra come utilizzare questa sintassi nella pratica.
Esempio: creazione di un grafico residuo in ggplot2
Per questo esempio, utilizzeremo il set di dati mtcars integrato in R:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
Innanzitutto, adatteremo un modello di regressione utilizzando mpg come variabile di risposta e qsec come variabile predittrice:
#fit regression model
model <- lm(mpg ~ qsec, data=mtcars)
Successivamente, utilizzeremo la seguente sintassi per creare un grafico residuo in ggplot2:
library (ggplot2) #create residual plot ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 )
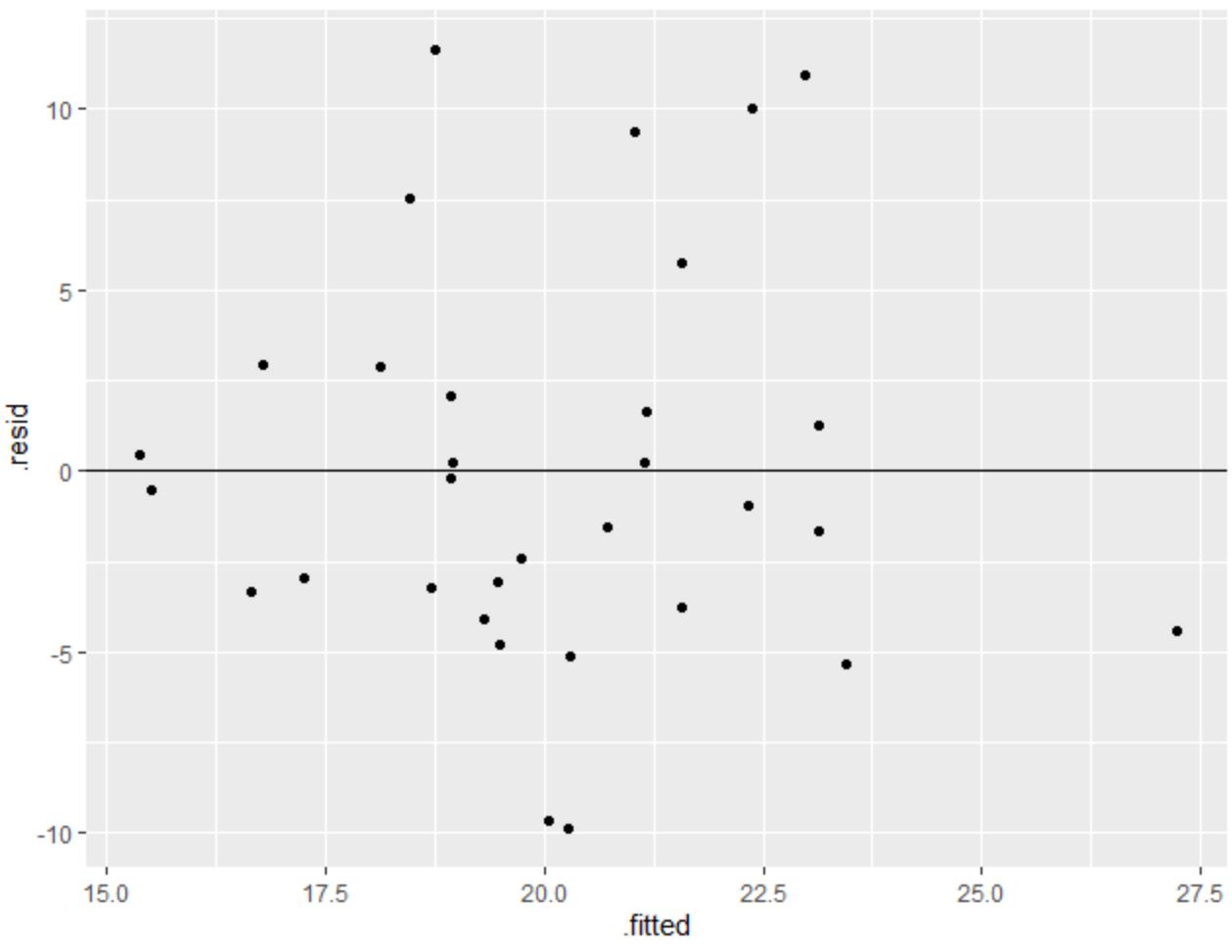
L’asse x mostra i valori adattati e l’asse y mostra i residui.
I residui sembrano essere sparsi in modo casuale attorno allo zero senza uno schema chiaro, indicando che l’ipotesi di omoschedasticità è soddisfatta.
In altre parole, i coefficienti del modello di regressione dovrebbero essere affidabili e non è necessario eseguire alcuna trasformazione sui dati.
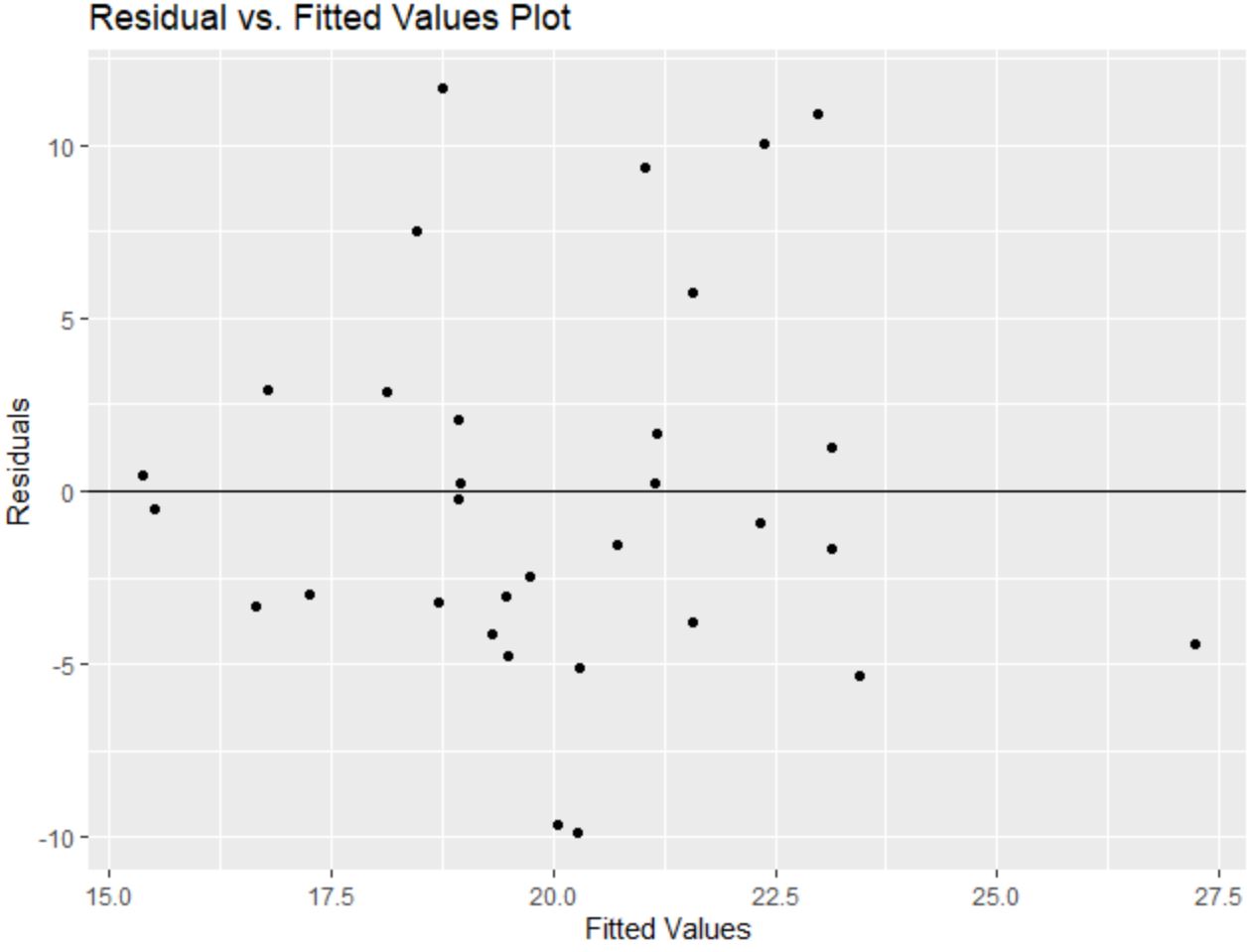
Tieni inoltre presente che potremmo utilizzare la funzione labs() per aggiungere un titolo e le etichette degli assi al grafico dei residui:
library (ggplot2) #create residual plot with title and axis labels ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 ) + labs(title=' Residual vs. Fitted Values Plot ', x=' Fitted Values ', y=' Residuals ')
Risorse addizionali
I seguenti tutorial spiegano come eseguire altre attività comuni in R:
Come calcolare i residui standardizzati in R
Come calcolare i residui studentizzati in R
Come creare un istogramma dei residui in R