Come eseguire un test di mancanza di adattamento in r (passo dopo passo)
Un test di mancanza di adattamento viene utilizzato per determinare se un modello di regressione completo fornisce o meno un adattamento significativamente migliore a un set di dati rispetto a una versione ridotta del modello.
Ad esempio, supponiamo di voler utilizzare il numero di ore di studio per prevedere i punteggi degli esami per gli studenti di un determinato college. Possiamo decidere di adattare i seguenti due modelli di regressione:
Modello completo: punteggio = β 0 + B 1 (ore) + B 2 (ore) 2
Modello ridotto: punteggio = β 0 + B 1 (ore)
Il seguente esempio dettagliato mostra come eseguire un test di adattamento per mancanza in R per determinare se il modello completo fornisce un adattamento significativamente migliore rispetto al modello ridotto.
Passaggio 1: crea e visualizza un set di dati
Innanzitutto, utilizzeremo il seguente codice per creare un set di dati contenente il numero di ore studiate e i punteggi degli esami ottenuti per 50 studenti:
#make this example reproducible set. seeds (1) #create dataset df <- data. frame (hours = runif (50, 5, 15), score=50) df$score = df$score + df$hours^3/150 + df$hours* runif (50, 1, 2) #view first six rows of data head(df) hours score 1 7.655087 64.30191 2 8.721239 70.65430 3 10.728534 73.66114 4 14.082078 86.14630 5 7.016819 59.81595 6 13.983897 83.60510
Successivamente, creeremo un grafico a dispersione per visualizzare la relazione tra ore e punteggio:
#load ggplot2 visualization package library (ggplot2) #create scatterplot ggplot(df, aes (x=hours, y=score)) + geom_point()
Passaggio 2: adattare due modelli diversi al set di dati
Successivamente, adatteremo due diversi modelli di regressione al set di dati:
#fit full model full <- lm(score ~ poly (hours,2), data=df) #fit reduced model reduced <- lm(score ~ hours, data=df)
Passaggio 3: eseguire un test di idoneità
Successivamente, utilizzeremo il comando anova() per eseguire un test di adattamento tra i due modelli:
#lack of fit test
anova(full, reduced)
Analysis of Variance Table
Model 1: score ~ poly(hours, 2)
Model 2: score ~ hours
Res.Df RSS Df Sum of Sq F Pr(>F)
1 47 368.48
2 48 451.22 -1 -82.744 10.554 0.002144 **
---
Significant. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
La statistica del test F risulta essere 10,554 e il corrispondente valore p è 0,002144 . Poiché questo valore p è inferiore a 0,05, possiamo rifiutare l’ipotesi nulla del test e concludere che il modello completo fornisce un adattamento migliore, statisticamente significativo, rispetto al modello ridotto.
Passaggio 4: Visualizza il modello finale
Infine, possiamo visualizzare il modello finale (il modello completo) rispetto al set di dati originale:
ggplot(df, aes (x=hours, y=score)) +
geom_point() +
stat_smooth(method=' lm ', formula = y ~ poly (x,2), size = 1) +
xlab(' Hours Studied ') +
ylab(' Score ')
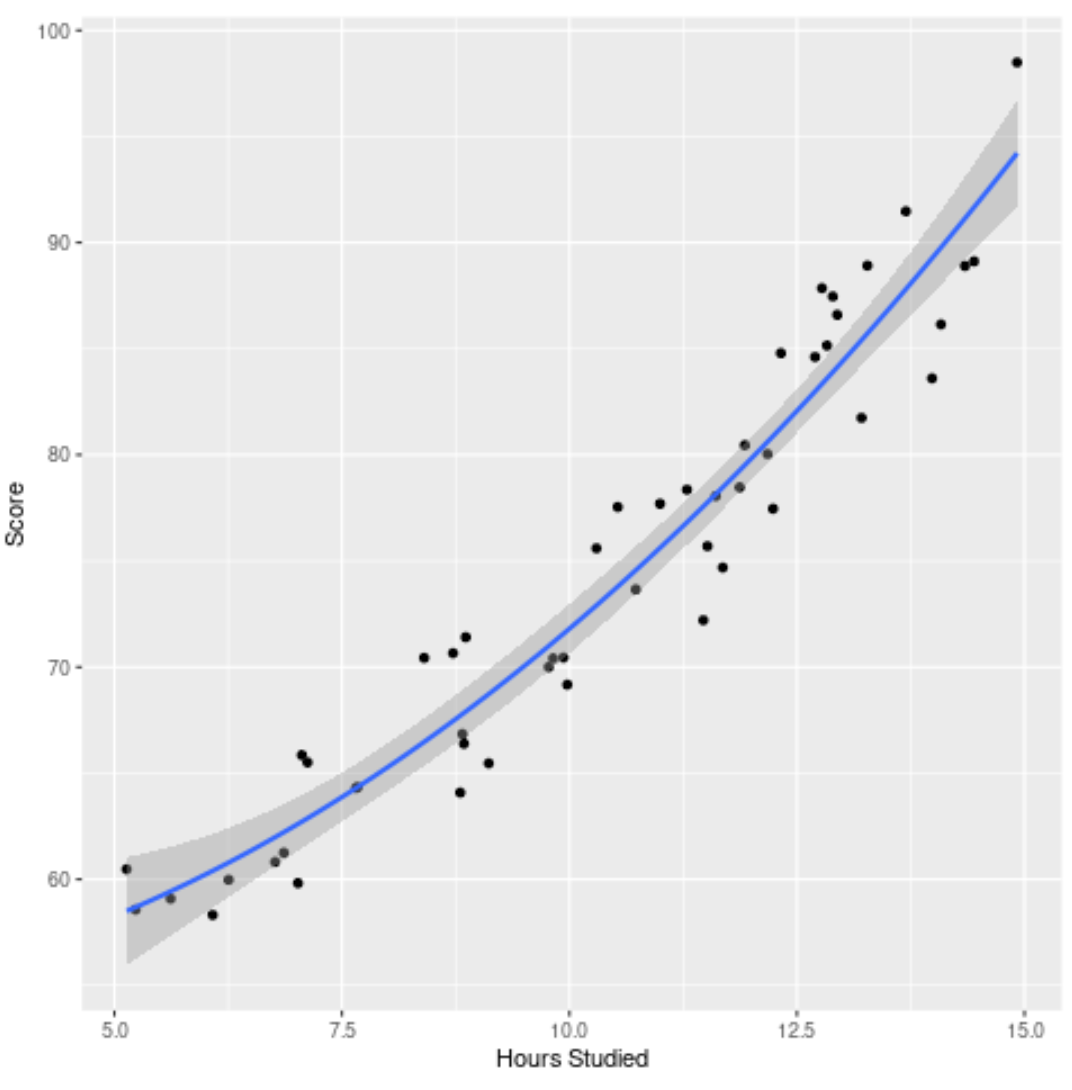
Possiamo vedere che la curva del modello si adatta abbastanza bene ai dati.
Risorse addizionali
Come eseguire una regressione lineare semplice in R
Come eseguire la regressione lineare multipla in R
Come eseguire la regressione polinomiale in R