Test di brown-forsythe in r: esempio passo passo
Un’ANOVA unidirezionale viene utilizzata per determinare se esiste o meno una differenza significativa tra le medie di tre o più gruppi indipendenti.
Uno dei presupposti di un’ANOVA unidirezionale è che le varianze delle popolazioni da cui vengono estratti i campioni siano uguali.
Uno dei modi più comuni per verificarlo è utilizzare un test di Brown-Forsythe , che è un test statistico che utilizza le seguenti ipotesi :
- H 0 : le varianze tra le popolazioni sono uguali.
- H A : Le differenze tra le popolazioni non sono uguali.
Se il valore p del test è inferiore a un certo livello di significatività (ad esempio α = 0,05), allora rifiutiamo l’ipotesi nulla e concludiamo che le varianze non sono uguali tra popolazioni diverse.
Questo tutorial fornisce un esempio passo passo di come eseguire un test di Brown-Forsythe in R.
Passaggio 1: inserisci i dati
Supponiamo di voler sapere se tre diversi programmi di allenamento portano o meno a diversi livelli di perdita di peso.
Per testarlo, reclutiamo 90 persone e ne assegniamo casualmente 30 a utilizzare ciascun programma. Quindi misuriamo la perdita di peso di ogni persona dopo un mese.
Il seguente set di dati contiene informazioni sulla quantità di peso perso con ciascun programma:
#make this example reproducible set.seed(0) #create data frame data <- data.frame(program = as . factor ( rep (c(" A ", " B ", " C "), each = 30)), weight_loss = c( runif (30, 0, 3), runif (30, 0, 5), runif (30, 1, 7))) #view first six rows of data frame head(data) # program weight_loss #1 A 2.6900916 #2 A 0.7965260 #3 A 1.1163717 #4 A 1.7185601 #5 A 2.7246234 #6 A 0.6050458
Passaggio 2: riepilogare e visualizzare i dati
Prima di eseguire un test di Brown-Forsythe, possiamo creare box plot per visualizzare la varianza nella perdita di peso per ciascun gruppo:
boxplot(weight_loss ~ program, data = data)
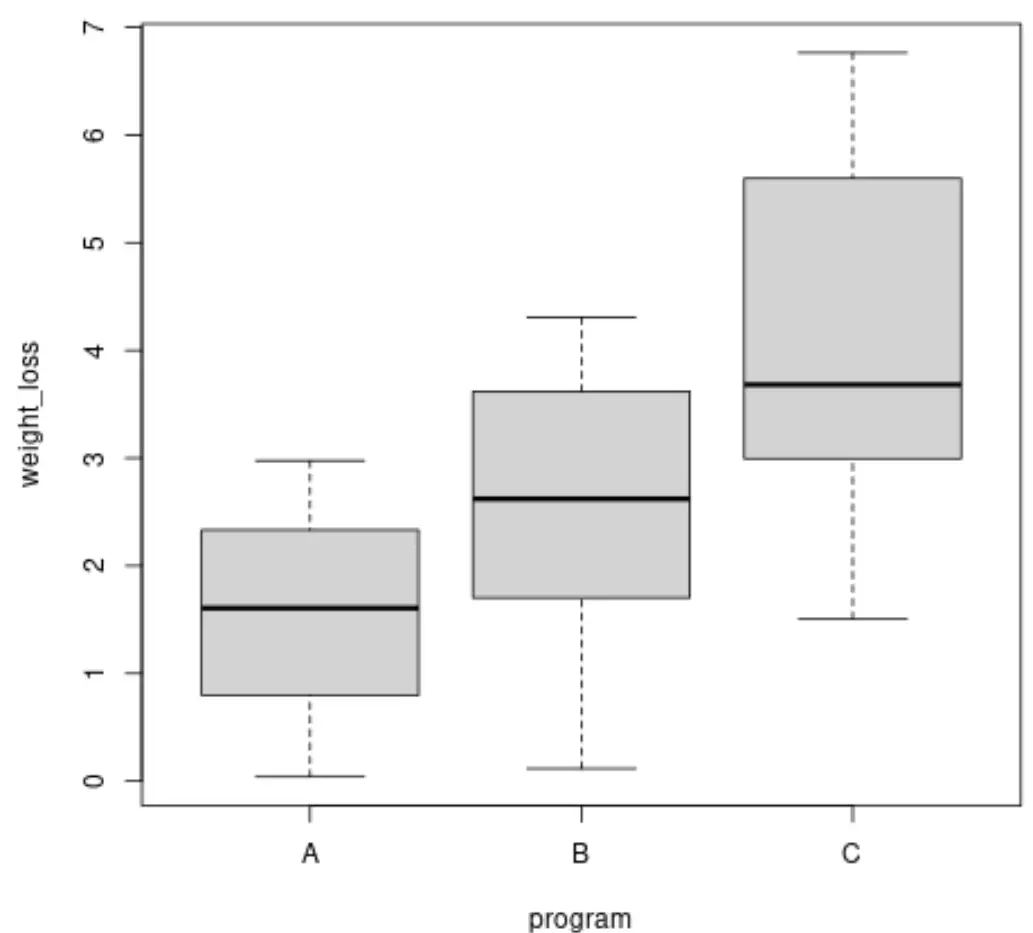
Possiamo anche calcolare la varianza della perdita di peso in ciascun gruppo:
#load dplyr package library (dplyr) #calculate variance of weight loss by group data %>% group_by (program) %>% summarize (var=var(weight_loss)) # A tibble: 3 x 2 program var 1 A 0.819 2 B 1.53 3 C 2.46
Possiamo vedere che le varianze tra i gruppi differiscono, ma per determinare se queste differenze sono statisticamente significative , possiamo eseguire il test di Brown-Forsythe.
Passaggio 3: eseguire il test di Brown-Forsythe
Per eseguire un test Brown-Forsythe in R, possiamo utilizzare la funzione bf.test() dal pacchetto onewaytests :
#load onewaytests package library (onewaytests) #perform Brown-Forsythe test bf.test(weight_loss ~ program, data = data) Brown-Forsythe Test (alpha = 0.05) -------------------------------------------------- ----------- data: weight_loss and program statistic: 30.83304 num df: 2 name df: 74.0272 p.value: 1.816529e-10 Result: Difference is statistically significant. -------------------------------------------------- -----------
Il p-value del test risulta essere inferiore a 0,000 e, come indica il risultato, le differenze nelle varianze tra i tre gruppi sono statisticamente significative.
Prossimi passi
Se non sei in grado di rifiutare l’ipotesi nulla del test di Brown-Forsythe, puoi eseguire un’ANOVA unidirezionale sui dati.
Tuttavia, se si rifiuta l’ipotesi nulla, significa che l’ipotesi di uguaglianza delle varianze è violata. In questo caso hai due opzioni:
1. Eseguire comunque un’ANOVA unidirezionale.
Si scopre che un’ANOVA unidirezionale è in realtà robusta rispetto a varianze disuguali purché la varianza maggiore non sia maggiore di 4 volte la varianza minima.
Nel passaggio 2 dell’esempio precedente, abbiamo scoperto che la varianza più piccola era 0,819 e la varianza più grande era 2,46. Quindi il rapporto tra la varianza più grande e quella più piccola è 2,46 / 0,819 = 3,003 .
Poiché questo valore è inferiore a 4, potremmo semplicemente eseguire l’ANOVA unidirezionale.
2. Eseguire un test Kruskal-Wallis
Se il rapporto tra la varianza più grande e la varianza più piccola è maggiore di 4, si può scegliere invece di eseguire un test di Kruskal-Wallis . Questo è considerato l’equivalente non parametrico dell’ANOVA unidirezionale.
Puoi trovare un esempio passo passo di un test Kruskal-Wallis in R qui .