R で不適合テストを実行する方法 (ステップバイステップ)
不適合テストは、完全な回帰モデルがモデルの縮小バージョンよりもデータセットに対して大幅に良好な適合を提供するかどうかを判断するために使用されます。
たとえば、学習時間数を使用して、特定の大学の学生の試験の得点を予測したいとします。次の 2 つの回帰モデルを適用することを決定できます。
完全なモデル:スコア = β 0 + B 1 (時間) + B 2 (時間) 2
縮小モデル:スコア = β 0 + B 1 (時間)
次のステップバイステップの例は、R で不適合テストを実行して、完全なモデルが縮小モデルよりも大幅に優れた適合を提供するかどうかを判断する方法を示しています。
ステップ 1: データセットを作成して視覚化する
まず、次のコードを使用して、50 人の学生の学習時間数と獲得した試験のスコアを含むデータセットを作成します。
#make this example reproducible set. seeds (1) #create dataset df <- data. frame (hours = runif (50, 5, 15), score=50) df$score = df$score + df$hours^3/150 + df$hours* runif (50, 1, 2) #view first six rows of data head(df) hours score 1 7.655087 64.30191 2 8.721239 70.65430 3 10.728534 73.66114 4 14.082078 86.14630 5 7.016819 59.81595 6 13.983897 83.60510
次に、散布図を作成して、時間とスコアの関係を視覚化します。
#load ggplot2 visualization package library (ggplot2) #create scatterplot ggplot(df, aes (x=hours, y=score)) + geom_point()
ステップ 2: 2 つの異なるモデルをデータセットに適合させる
次に、2 つの異なる回帰モデルをデータセットに適合させます。
#fit full model full <- lm(score ~ poly (hours,2), data=df) #fit reduced model reduced <- lm(score ~ hours, data=df)
ステップ 3: 不適合テストを実行する
次に、 anova()コマンドを使用して、2 つのモデル間の不適合テストを実行します。
#lack of fit test
anova(full, reduced)
Analysis of Variance Table
Model 1: score ~ poly(hours, 2)
Model 2: score ~ hours
Res.Df RSS Df Sum of Sq F Pr(>F)
1 47 368.48
2 48 451.22 -1 -82.744 10.554 0.002144 **
---
Significant. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
F 検定統計量は10.554で、対応する p 値は0.002144であることがわかります。この p 値は 0.05 未満であるため、検定の帰無仮説を棄却し、完全なモデルが縮小モデルよりも統計的に有意に良好な適合を提供すると結論付けることができます。
ステップ 4: 最終モデルを視覚化する
最後に、元のデータセットに対して最終モデル (完全なモデル) を視覚化できます。
ggplot(df, aes (x=hours, y=score)) +
geom_point() +
stat_smooth(method=' lm ', formula = y ~ poly (x,2), size = 1) +
xlab(' Hours Studied ') +
ylab(' Score ')
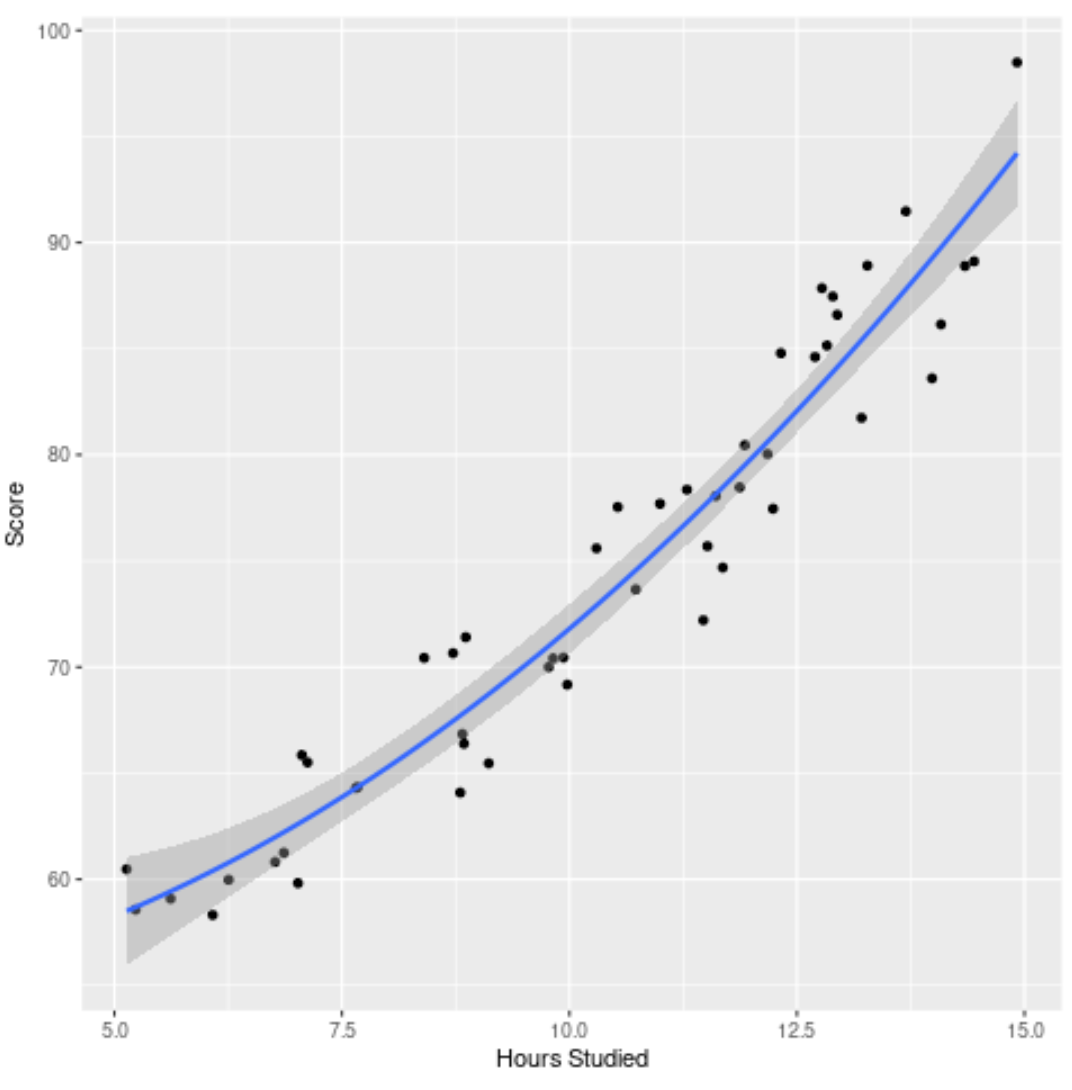
モデル曲線がデータに非常によく適合していることがわかります。