R の部分最小二乗法 (ステップバイステップ)
機械学習で遭遇する最も一般的な問題の 1 つは、 多重共線性です。これは、データセット内の 2 つ以上の予測子変数の相関性が高い場合に発生します。
これが起こると、モデルはトレーニング データ セットにうまく適合できるかもしれませんが、トレーニング データ セットに過剰適合するため、これまでに見たことのない新しいデータ セットではパフォーマンスが低下する可能性があります。トレーニングセット。
この問題を回避する 1 つの方法は、次のように機能する 部分最小二乗法と呼ばれる方法を使用することです。
- 予測変数と応答変数を標準化します。
- 応答変数と予測子変数の両方における有意な量の変動を説明する、 p個の元の予測子変数のM 個の線形結合 (「PLS 成分」と呼ばれる) を計算します。
- 最小二乗法を使用して、PLS コンポーネントを予測子として使用して線形回帰モデルを近似します。
- k 分割交差検証を使用して、モデルに保持する PLS コンポーネントの最適な数を見つけます。
このチュートリアルでは、R で部分最小二乗法を実行する方法の段階的な例を示します。
ステップ 1: 必要なパッケージをロードする
R で部分最小二乗計算を実行する最も簡単な方法は、 plsパッケージの関数を使用することです。
#install pls package (if not already installed) install.packages(" pls ") load pls package library(pls)
ステップ 2: 部分最小二乗モデルを当てはめる
この例では、さまざまなタイプの車のデータが含まれるmtcarsという組み込みの R データセットを使用します。
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
この例では、 応答変数としてhp を使用し、予測変数として次の変数を使用して、部分最小二乗 (PLS) モデルを近似します。
- mpg
- 画面
- たわごと
- 重さ
- qsec
次のコードは、PLS モデルをこのデータに適合させる方法を示しています。次の引数に注意してください。
- scale=TRUE : これは、データセット内の各変数が平均 0、標準偏差 1 になるようにスケーリングする必要があることを R に指示します。これにより、異なる単位で測定された場合に、予測子変数がモデルに過度の影響を与えることがなくなります。
- validation=”CV” : これは R にk 分割相互検証を使用してモデルのパフォーマンスを評価するように指示します。これはデフォルトで k=10 のフォールドを使用することに注意してください。また、代わりに「LOOCV」を指定して、Leave-One-Out 相互検証を実行できることにも注意してください。
#make this example reproducible set.seed(1) #fit PCR model model <- plsr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
ステップ 3: PLS コンポーネントの数を選択する
モデルを当てはめたら、保持する PLS コンポーネントの数を決定する必要があります。
これを行うには、k-cross 検証によって計算されたテスト二乗平均平方根誤差 (テスト RMSE) を確認するだけです。
#view summary of model fitting
summary(model)
Data:
Y dimension: 32 1
Fit method: kernelpls
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comp 2 comps 3 comps 4 comps 5 comps
CV 69.66 40.57 35.48 36.22 36.74 36.67
adjCV 69.66 40.41 35.12 35.80 36.27 36.20
TRAINING: % variance explained
1 comp 2 comps 3 comps 4 comps 5 comps
X 68.66 89.27 95.82 97.94 100.00
hp 71.84 81.74 82.00 82.02 82.03
結果には 2 つの興味深いテーブルがあります。
1. 検証: RMSEP
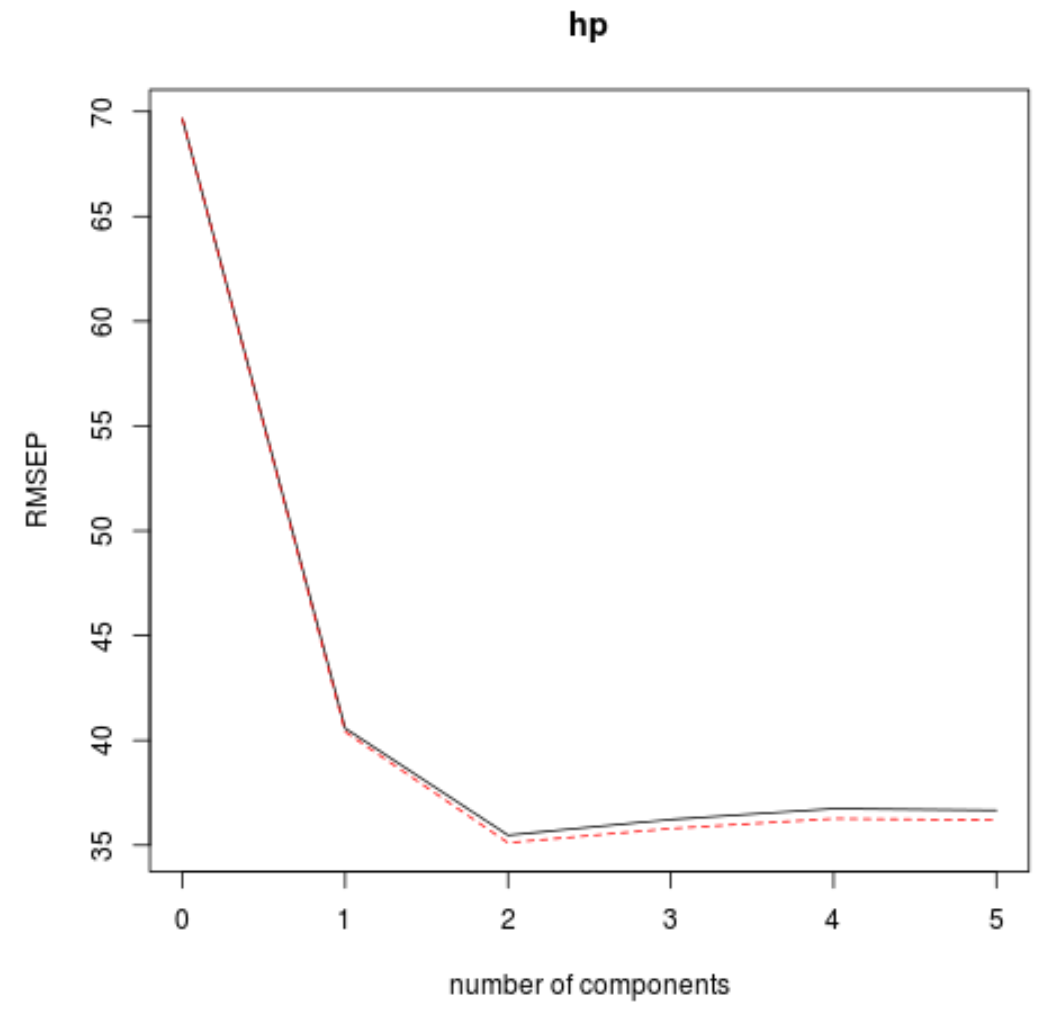
この表は、k 分割相互検証によって計算された RMSE テストを示しています。次のことがわかります。
- モデル内の元の項のみを使用した場合、テストの RMSE は69.66になります。
- 最初の PLS コンポーネントを追加すると、RMSE テストは40.57 に低下します。
- 2 番目の PLS コンポーネントを追加すると、RMSE テストは35.48 に低下します。
PLS コンポーネントを追加すると、実際にテストの RMSE が増加することがわかります。したがって、最終モデルでは PLS コンポーネントを 2 つだけ使用するのが最適であると思われます。
2. トレーニング: 分散の % を説明する
この表は、PLS コンポーネントによって説明される応答変数の分散のパーセンテージを示しています。次のことがわかります。
- 最初の PLS コンポーネントのみを使用すると、応答変数の変動の68.66%を説明できます。
- 2 番目の PLS コンポーネントを追加することで、応答変数の変動の89.27%を説明できます。
より多くの PLS コンポーネントを使用することで、より多くの分散を説明できることに注意してください。ただし、3 つ以上の PLS コンポーネントを追加しても、説明される分散のパーセンテージは実際にはそれほど増加しないことがわかります。
validationplot()関数を使用して、PLS コンポーネントの数の関数として RMSE テスト (MSE および R 二乗テストとともに) を視覚化することもできます。
#visualize cross-validation plots validationplot(model) validationplot(model, val.type=" MSEP ") validationplot(model, val.type=" R2 ")
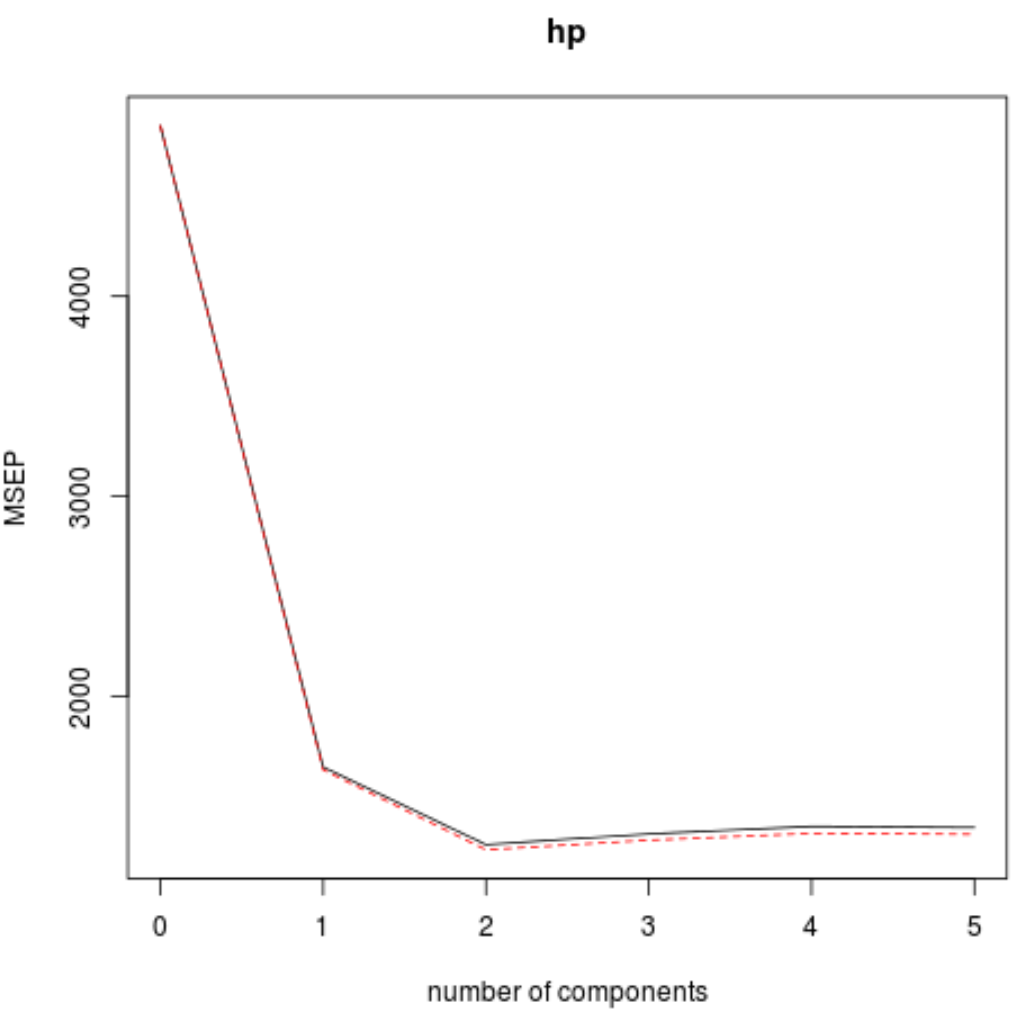
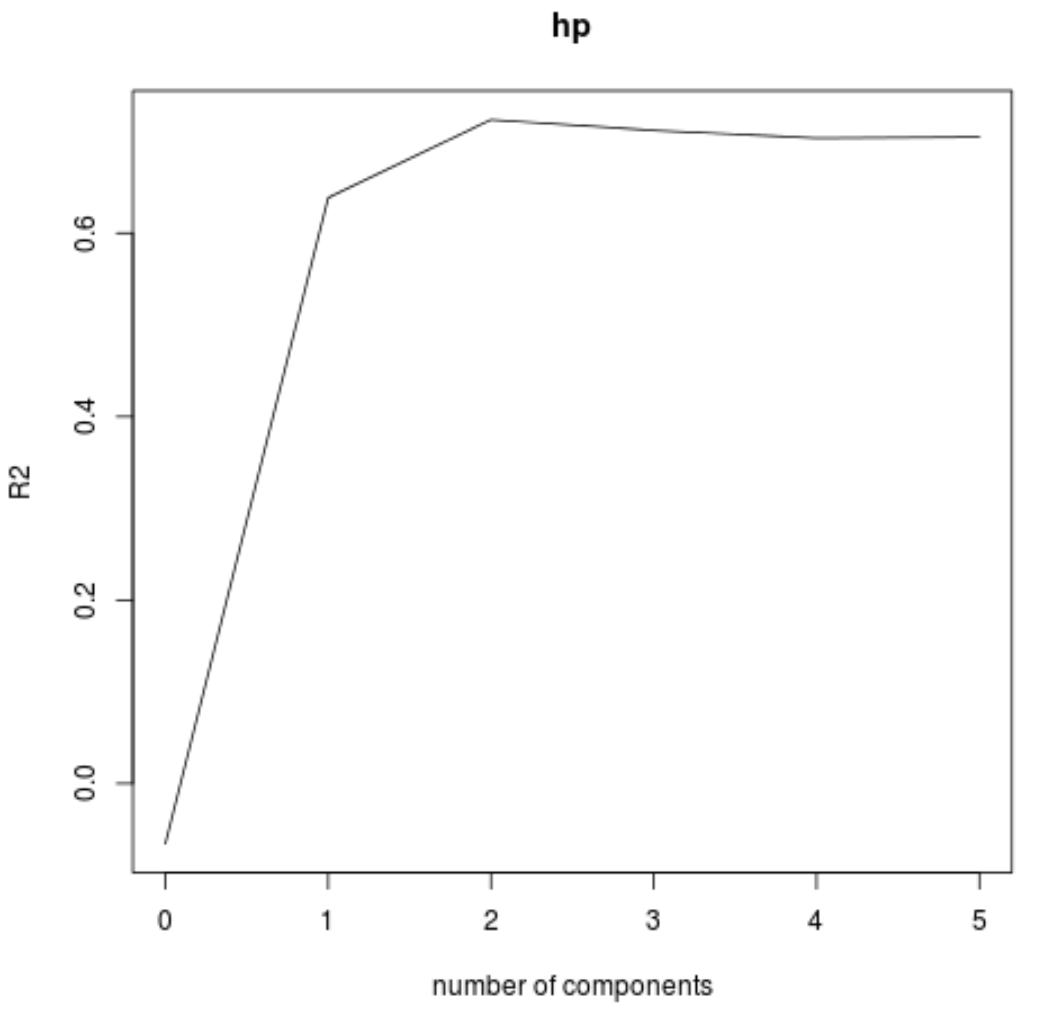
各グラフでは、2 つの PLS コンポーネントを追加することでモデルの適合性が向上しますが、さらに多くの PLS コンポーネントを追加するとモデルの適合性が低下する傾向があることがわかります。
したがって、最適なモデルには最初の 2 つの PLS コンポーネントのみが含まれます。
ステップ 4: 最終モデルを使用して予測を行う
2 つの PLS コンポーネントを含む最終モデルを使用して、新しい観測についての予測を行うことができます。
次のコードは、元のデータセットをトレーニング セットとテスト セットに分割し、2 つの PLS コンポーネントを含む最終モデルを使用してテスト セットで予測を行う方法を示しています。
#define training and testing sets train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")] y_test <- mtcars[26: nrow (mtcars), c("hp")] test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")] #use model to make predictions on a test set model <- plsr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ") pcr_pred <- predict(model, test, ncomp= 2 ) #calculate RMSE sqrt ( mean ((pcr_pred - y_test)^2)) [1] 54.89609
テストの RMSE が54.89609であることがわかります。これは、テスト セットの観測値の予測hp値と観測されたhp値の間の平均偏差です。
2 つの主成分を含む等価主成分回帰モデルでは、テスト RMSE が56.86549になることに注意してください。したがって、このデータセットでは、PLS モデルが PCR モデルよりわずかに優れたパフォーマンスを示しました。
この例での R コードの完全な使用法は、 ここで参照できます。