R でログ ランク テストを実行する方法
ログランク検定は、 2 つのグループ間の生存曲線を比較する最も一般的な方法です。
このテストでは次の仮定を使用します。
H 0 : 2 つのグループ間で生存率に差はありません。
H A : 2 つのグループの間には生存率に差があります。
検定のp 値が一定の有意水準 (例: α = 0.05) を下回っている場合、帰無仮説を棄却し、2 つのグループ間に生存率に差があると言える十分な証拠があると結論付けることができます。
R でログランク テストを実行するには、 survdiff()関数をSurvdiff () 関数を使用します (次の構文を使用します)。
Survdiff(Surv(時間, ステータス) ~ 予測子, データ)
この関数は、カイ二乗検定統計量と対応する p 値を返します。
次の例は、この関数を使用して R でログランク テストを実行する方法を示しています。
例: R でのログ ランク テスト
この例では、 Survivalパッケージの卵巣データセットを使用します。このデータセットには、26 人の患者に関する次の情報が含まれています。
- 卵巣がんと診断された後の生存期間(月単位)
- 生存時間が検閲されたかどうか
- 受けた治療の種類 (rx = 1 または rx = 2)
次のコードは、このデータセットの最初の 6 行を表示する方法を示しています。
library (survival) #view first six rows of dataset head(ovarian) futime fustat age resid.ds rx ecog.ps 1 59 1 72.3315 2 1 1 2 115 1 74.4932 2 1 1 3 156 1 66.4658 2 1 2 4 421 0 53.3644 2 2 1 5,431 1 50.3397 2 1 1 6 448 0 56.4301 1 1 2
次のコードは、ログランク テストを実行して、異なる治療を受けた患者間に生存率の差があるかどうかを判断する方法を示しています。
#perform log rank test
survdiff(Surv(futime, fustat) ~ rx, data=ovarian)
Call:
survdiff(formula = Surv(futime, fustat) ~ rx, data = ovarian)
N Observed Expected (OE)^2/E (OE)^2/V
rx=1 13 7 5.23 0.596 1.06
rx=2 13 5 6.77 0.461 1.06
Chisq= 1.1 on 1 degrees of freedom, p= 0.3
カイ二乗検定統計量は1.1 (自由度 1)、対応する p 値は0.3です。この p 値は 0.05 未満ではないため、帰無仮説を棄却できません。
言い換えれば、2 つの治療法の間に統計的に有意な生存期間の差があると言える十分な証拠はありません。
次の構文を使用して、各グループの生存曲線をプロットすることもできます。
#plot survival curves for each treatment group plot(survfit(Surv(futime, fustat) ~ rx, data = ovarian), xlab = " Time ", ylab = “ Overall survival probability ”)
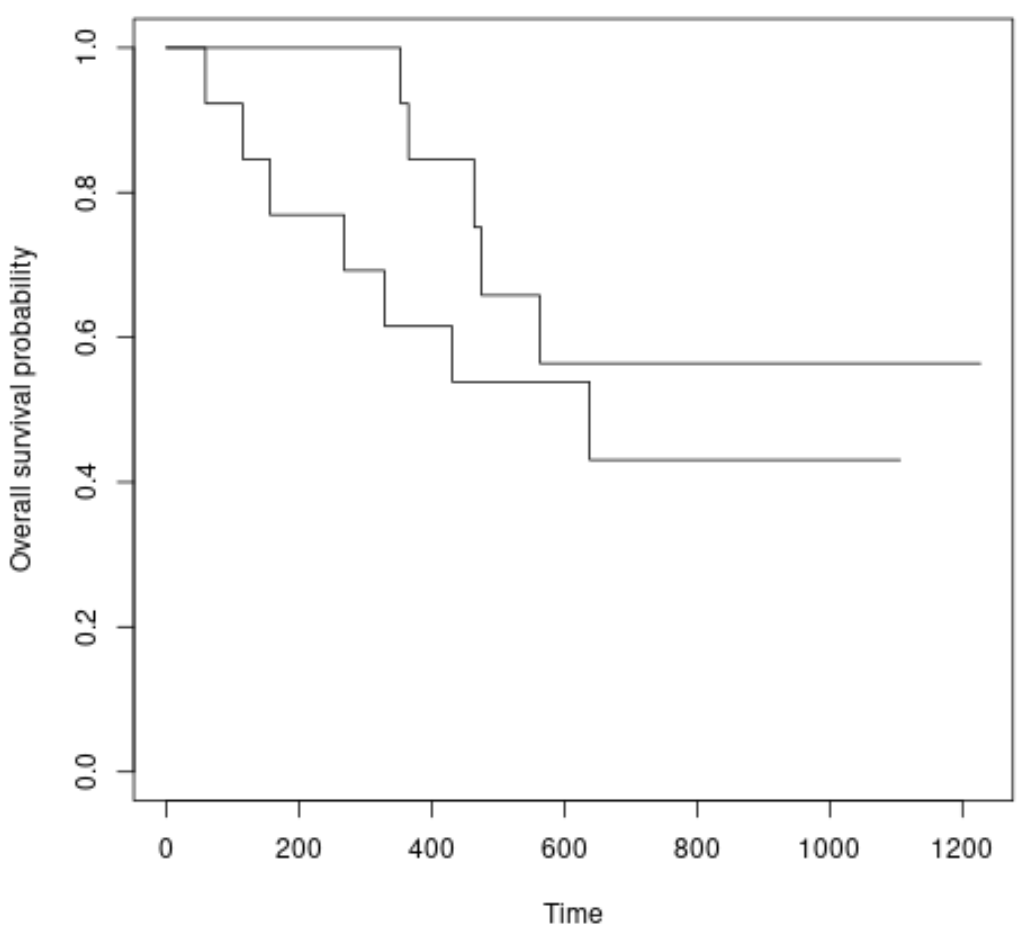
生存曲線がわずかに異なることがわかりますが、ログランク検定では、その差が統計的に有意ではないことがわかりました。