R でブレイとカーティスの相違度を計算する方法
Bray-Curtis の相違度は、 2 つの異なるサイト間の相違度を測定する方法です。
これは、生態学や生物学において、2 つの場所で見られる種の観点から 2 つの場所の違いを定量化するためによく使用されます。
次のように計算されます。
BC ij = 1 – (2*C ij ) / (S i + S j )
金:
- C ij :各サイトで見つかった種の最小値の合計。
- S i :サイトiで計数された検体の総数
- S j :サイトjで計数された検体の総数
ブレイとカーティスの相違度は常に 0 と 1 の間にあります。ここで、
- 0 は、2 つのサイトに非類似性がないことを示します。言い換えれば、各種類の種の数がまったく同じになります。
- 1 は、2 つのサイトが完全に類似していないことを示します。言い換えれば、それらは同じ種類の種を共有しません。
たとえば、植物学者が外出して、2 つの異なる場所で 5 つの異なる植物種 (A、B、C、D、および E) の数を数えたとします。
次の表は、彼女が収集したデータをまとめたものです。
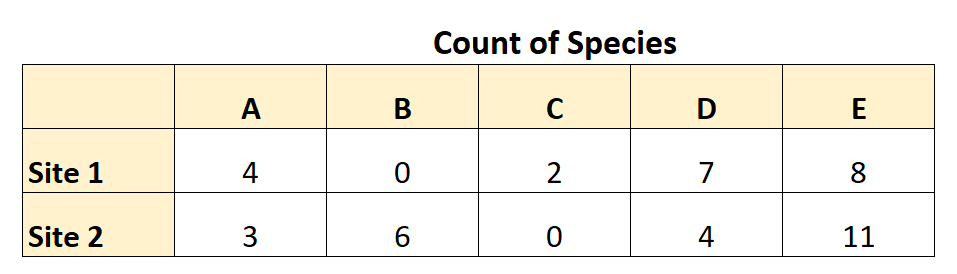
このデータを使用すると、次のようにブレイとカーティスの相違度を計算できます。
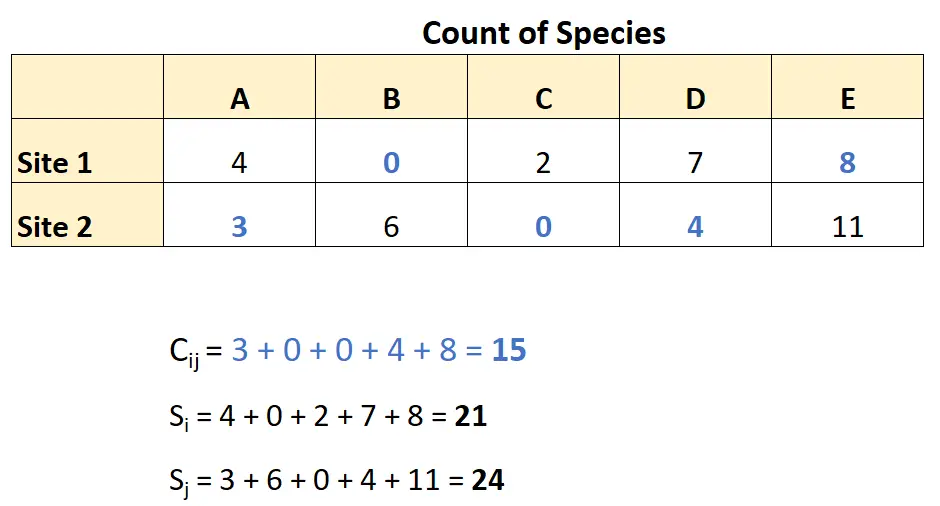
これらの数値を Bray-Curtis の相違度の公式に統合すると、次の結果が得られます。
- BC ij = 1 – (2*C ij ) / (S i + S j )
- BC ij = 1 – (2*15) / (21 + 24)
- BC ij = 0.33
これら 2 つのサイト間の Bray-Curtis の相違度は0.33です。
次の例は、R で Bray-Curtis の相違度を計算する方法を示しています。
例: R でのブレイとカーティスの相違度の計算
まず、データ値を保持するために R で次のデータ フレームを作成しましょう。
#create data frame df <- data. frame (A=c(4, 3), B=c(0, 6), C=c(2, 0), D=c(7, 4), E=c(8, 11)) #view data frame df A B C D E 1 4 0 2 7 8 2 3 6 0 4 11
次のコードを使用して、データ フレーム内の 2 つの行間の Bray-Curtis の非類似度を計算できます。
#calculate Bray–Curtis dissimilarity sum( apply (df, 2, function (x) abs ( max (x)- min (x)))) / sum ( rowSums (df)) [1] 0.3333333
Bray-Curtis は0.33であることが判明しました。
これは、以前に手動で計算した値と一致します。
注: この数式は、データ フレーム内の各行が個別のサイトを表す場合にのみ機能します。
追加リソース
次のチュートリアルでは、R で他の類似性メトリクスを計算する方法を説明します。