R에서 bray-curtis 차이점을 계산하는 방법
Bray-Curtis 차이점은 서로 다른 두 사이트 간의 차이점을 측정하는 방법입니다.
이는 생태학 및 생물학에서 두 지역에서 발견된 종의 측면에서 두 지역 간의 차이를 정량화하기 위해 자주 사용됩니다.
다음과 같이 계산됩니다.
BC ij = 1 – (2*C ij ) / (S i + S j )
금:
- C ij : 각 현장에서 발견된 종의 최저값의 합.
- S i : 사이트 i 에서 계수된 총 검체 수
- S j : 사이트 j 에서 계수된 총 검체 수
Bray-Curtis 차이점은 항상 0과 1 사이입니다.
- 0은 두 사이트에 차이점이 없음을 나타냅니다. 즉, 그들은 각 유형의 종을 정확히 동일한 수로 공유합니다.
- 1은 두 사이트가 완전히 다르다는 것을 나타냅니다. 즉, 동일한 유형의 어떤 종도 공유하지 않습니다.
예를 들어, 식물학자가 두 곳의 다른 장소에 나가서 다섯 가지 식물 종(A, B, C, D, E)의 수를 세었다고 가정해 보겠습니다.
다음 표에는 그녀가 수집한 데이터가 요약되어 있습니다.
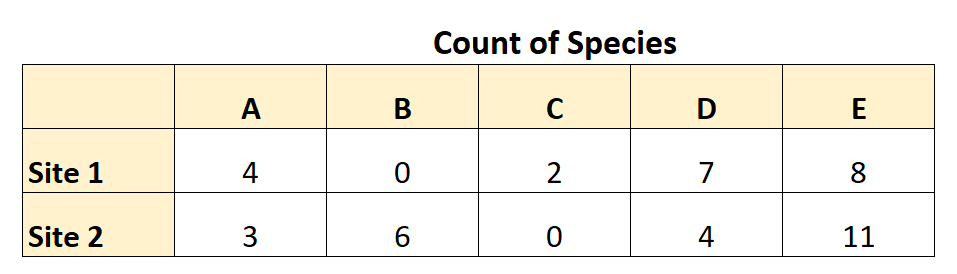
이 데이터를 사용하여 그녀는 Bray-Curtis 상이성을 다음과 같이 계산할 수 있습니다.
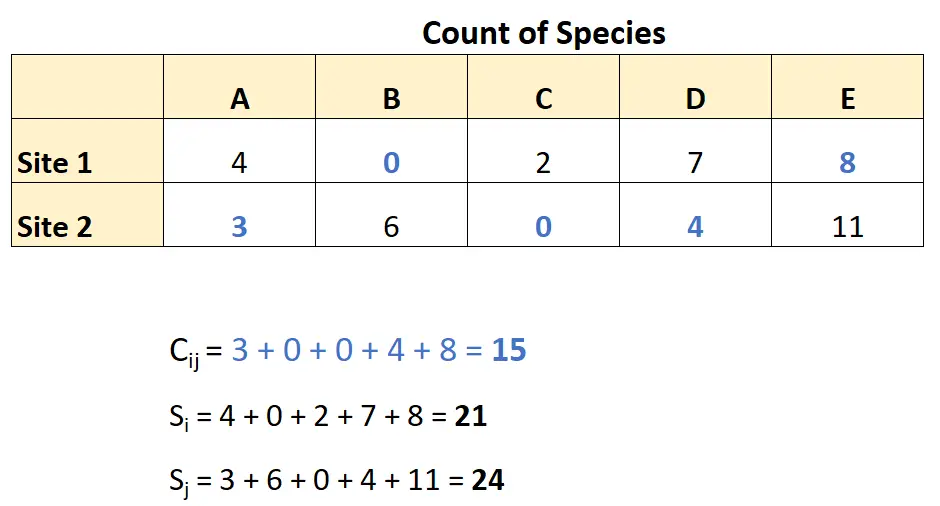
이 숫자를 Bray-Curtis 비유사성 공식에 통합하면 다음을 얻을 수 있습니다.
- BC ij = 1 – (2*C ij ) / (S i + S j )
- BC ij = 1 – (2*15) / (21 + 24)
- 기원전 ij = 0.33
이 두 사이트 간의 Bray-Curtis 차이점은 0.33 입니다.
다음 예는 R에서 Bray-Curtis 비평사성을 계산하는 방법을 보여줍니다.
예: R에서 Bray-Curtis 차이점 계산
먼저, 데이터 값을 보관하기 위해 R에서 다음 데이터 프레임을 생성해 보겠습니다.
#create data frame df <- data. frame (A=c(4, 3), B=c(0, 6), C=c(2, 0), D=c(7, 4), E=c(8, 11)) #view data frame df A B C D E 1 4 0 2 7 8 2 3 6 0 4 11
다음 코드를 사용하여 데이터 프레임의 두 행 사이의 Bray-Curtis 차이점을 계산할 수 있습니다.
#calculate Bray–Curtis dissimilarity sum( apply (df, 2, function (x) abs ( max (x)- min (x)))) / sum ( rowSums (df)) [1] 0.3333333
Bray-Curtis는 0.33 으로 다르게 나타났습니다.
이는 이전에 수동으로 계산한 값과 일치합니다.
참고 : 이 공식은 데이터 프레임의 각 행이 별도의 사이트를 나타내는 경우에만 작동합니다.
추가 리소스
다음 튜토리얼에서는 R에서 다른 유사성 측정항목을 계산하는 방법을 설명합니다.