Python의 주성분 회귀(단계별)
p 개의 예측 변수 세트와 응답 변수가 주어지면 다중 선형 회귀 분석 에서는 RSS(잔차 제곱합)를 최소화하기 위해 최소 제곱이라는 방법을 사용합니다.
RSS = Σ(y i – ŷ i ) 2
금:
- Σ : 합계를 의미하는 그리스 기호
- y i : i번째 관측값에 대한 실제 응답 값
- ŷ i : 다중선형회귀모델을 기반으로 예측된 반응값
그러나 예측변수의 상관관계가 높으면 다중공선성이 문제가 될 수 있습니다. 이로 인해 모델 계수 추정이 신뢰할 수 없게 되고 높은 분산이 나타날 수 있습니다.
이 문제를 피하는 한 가지 방법은 주성분 회귀를 사용하는 것입니다. 이는 원래 p 예측 변수의 M개의 선형 조합(“주성분”이라고 함)을 찾은 다음 최소 제곱을 사용하여 주성분을 예측 변수로 사용하여 선형 회귀 모델을 적합시킵니다.
이 튜토리얼에서는 Python에서 주성분 회귀를 수행하는 방법에 대한 단계별 예를 제공합니다.
1단계: 필요한 패키지 가져오기
먼저 Python에서 주성분 회귀(PCR)를 수행하는 데 필요한 패키지를 가져옵니다.
import numpy as np
import pandas as pd
import matplotlib. pyplot as plt
from sklearn. preprocessing import scale
from sklearn import model_selection
from sklearn. model_selection import RepeatedKFold
from sklearn.model_selection import train_test_split
from sklearn. PCA import decomposition
from sklearn. linear_model import LinearRegression
from sklearn. metrics import mean_squared_error
2단계: 데이터 로드
이 예에서는 33개의 서로 다른 자동차에 대한 정보가 포함된 mtcars 라는 데이터 세트를 사용합니다. hp를 반응 변수로 사용하고 다음 변수를 예측 변수로 사용합니다.
- mpg
- 표시하다
- 똥
- 무게
- q초
다음 코드는 이 데이터세트를 로드하고 표시하는 방법을 보여줍니다.
#define URL where data is located
url = "https://raw.githubusercontent.com/Statorials/Python-Guides/main/mtcars.csv"
#read in data
data_full = pd. read_csv (url)
#select subset of data
data = data_full[["mpg", "disp", "drat", "wt", "qsec", "hp"]]
#view first six rows of data
data[0:6]
mpg disp drat wt qsec hp
0 21.0 160.0 3.90 2.620 16.46 110
1 21.0 160.0 3.90 2.875 17.02 110
2 22.8 108.0 3.85 2.320 18.61 93
3 21.4 258.0 3.08 3.215 19.44 110
4 18.7 360.0 3.15 3.440 17.02 175
5 18.1 225.0 2.76 3.460 20.22 105
3단계: PCR 모델 조정
다음 코드는 PCR 모델을 이 데이터에 맞추는 방법을 보여줍니다. 다음 사항에 유의하세요.
- pca.fit_transform(scale(X)) : 이는 각 예측 변수가 평균 0, 표준 편차 1을 갖도록 스케일링되어야 함을 Python에 지시합니다. 이렇게 하면 예측 변수가 다음과 같은 경우 모델에 너무 많은 영향을 미치지 않도록 합니다. 이런 일이 발생합니다. 다양한 단위로 측정해야 합니다.
- cv = RepeatedKFold() : Python에 k-겹 교차 검증을 사용하여 모델 성능을 평가하도록 지시합니다. 이 예에서는 k = 10 접기를 선택하고 3번 반복합니다.
#define predictor and response variables
X = data[["mpg", "disp", "drat", "wt", "qsec"]]
y = data[["hp"]]
#scale predictor variables
pca = pca()
X_reduced = pca. fit_transform ( scale (X))
#define cross validation method
cv = RepeatedKFold(n_splits= 10 , n_repeats= 3 , random_state= 1 )
regr = LinearRegression()
mse = []
# Calculate MSE with only the intercept
score = -1*model_selection. cross_val_score (regr,
n.p. ones ((len(X_reduced),1)), y, cv=cv,
scoring=' neg_mean_squared_error '). mean ()
mse. append (score)
# Calculate MSE using cross-validation, adding one component at a time
for i in np. arange (1, 6):
score = -1*model_selection. cross_val_score (regr,
X_reduced[:,:i], y, cv=cv, scoring=' neg_mean_squared_error '). mean ()
mse. append (score)
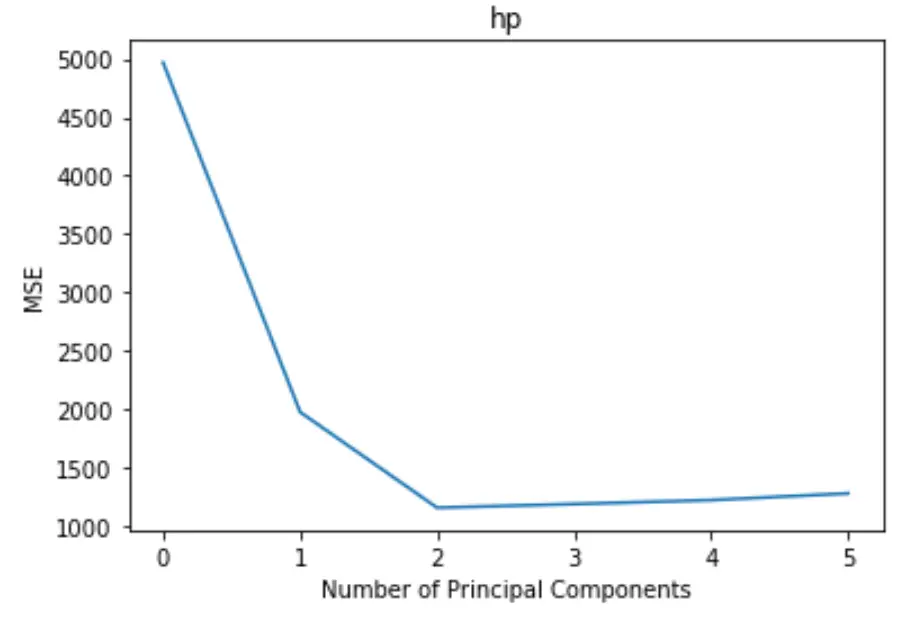
# Plot cross-validation results
plt. plot (mse)
plt. xlabel ('Number of Principal Components')
plt. ylabel ('MSE')
plt. title ('hp')
플롯은 x축을 따라 주성분 수를 표시하고 y축을 따라 MSE(평균 제곱 오차) 테스트를 표시합니다.
그래프에서 두 개의 주성분을 추가하면 테스트의 MSE가 감소하지만 두 개 이상의 주성분을 추가하면 증가하기 시작하는 것을 볼 수 있습니다.
따라서 최적 모델에는 처음 두 개의 주성분만 포함됩니다.
또한 다음 코드를 사용하여 모델에 각 주성분을 추가하여 설명된 응답 변수의 분산 백분율을 계산할 수 있습니다.
n.p. cumsum (np. round (pca. explained_variance_ratio_ , decimals= 4 )* 100 )
array([69.83, 89.35, 95.88, 98.95, 99.99])
우리는 다음을 볼 수 있습니다:
- 첫 번째 주성분만 사용하여 반응 변수 변동의 69.83% 를 설명할 수 있습니다.
- 두 번째 주성분을 추가하면 반응 변수 변동의 89.35% 를 설명할 수 있습니다.
더 많은 주성분을 사용하여 더 많은 분산을 설명할 수 있지만, 두 개 이상의 주성분을 추가해도 실제로 설명된 분산의 비율이 크게 증가하지 않는다는 것을 알 수 있습니다.
4단계: 최종 모델을 사용하여 예측
최종 2주성분 PCR 모델을 사용하여 새로운 관찰에 대한 예측을 할 수 있습니다.
다음 코드는 원본 데이터 세트를 훈련 세트와 테스트 세트로 분할하고 두 가지 주요 구성 요소가 있는 PCR 모델을 사용하여 테스트 세트에 대해 예측하는 방법을 보여줍니다.
#split the dataset into training (70%) and testing (30%) sets
X_train,X_test,y_train,y_test = train_test_split (X,y,test_size= 0.3 , random_state= 0 )
#scale the training and testing data
X_reduced_train = pca. fit_transform ( scale (X_train))
X_reduced_test = pca. transform ( scale (X_test))[:,:1]
#train PCR model on training data
regr = LinearRegression()
reg. fit (X_reduced_train[:,:1], y_train)
#calculate RMSE
pred = regr. predict (X_reduced_test)
n.p. sqrt ( mean_squared_error (y_test, pred))
40.2096
RMSE 테스트 결과는 40.2096 인 것으로 나타났습니다. 이는 테스트 세트 관찰에 대해 예측된 HP 값과 관찰된 HP 값 사이의 평균 편차입니다.
이 예제에 사용된 전체 Python 코드는 여기에서 찾을 수 있습니다.