R의 k-medoids: 단계별 예
클러스터링은 데이터 세트 내에서 관찰 그룹 또는 클러스터를 찾으려고 시도하는 기계 학습 기술입니다.
목표는 각 군집 내의 관측값이 서로 매우 유사한 반면, 다른 군집의 관측값은 서로 상당히 다른 군집을 찾는 것입니다.
클러스터링은 비지도 학습 의 한 형태입니다 . 응답 변수 의 값을 예측하는 것이 아니라 단순히 데이터 세트 내에서 구조를 찾으려고 하기 때문입니다.
클러스터링은 기업이 다음과 같은 정보에 액세스할 수 있는 경우 마케팅에 자주 사용됩니다.
- 가구 소득
- 가구 규모
- 세대주 직업
- 가장 가까운 도시 지역까지의 거리
이 정보를 사용할 수 있으면 클러스터링을 사용하여 유사하고 특정 제품을 구매할 가능성이 높거나 특정 유형의 광고에 더 잘 반응할 수 있는 가구를 식별할 수 있습니다.
클러스터링의 가장 일반적인 형태 중 하나는 k-평균 클러스터링 으로 알려져 있습니다.
불행하게도 이 방법은 이상값의 영향을 받을 수 있으므로 k-medoids 클러스터링이 자주 사용되는 대안입니다.
K-Medoids 클러스터링이란 무엇입니까?
K-medoids 클러스터링은 데이터세트의 각 관측값을 K 클러스터 중 하나에 배치하는 기술입니다.
최종 목표는 각 클러스터 내의 관측치가 서로 매우 유사한 반면 다른 클러스터의 관측치는 서로 상당히 다른 K 개의 클러스터를 갖는 것입니다.
실제로 K-평균 클러스터링을 수행하기 위해 다음 단계를 사용합니다.
1. K 값을 선택합니다.
- 먼저, 데이터에서 식별하려는 클러스터 수를 결정해야 합니다. 종종 우리는 K 에 대해 여러 가지 다른 값을 테스트하고 결과를 분석하여 주어진 문제에 가장 적합한 클러스터 수를 확인해야 합니다.
2. 각 관측치를 1부터 K 까지 초기 클러스터에 무작위로 할당합니다.
3. 클러스터 할당 변경이 중지될 때까지 다음 절차를 수행합니다.
- K 개의 클러스터 각각에 대해 클러스터의 무게 중심을 계산합니다. 이는 k 번째 클러스터의 관측값에 대한 특징의 p개의 중앙값 으로 구성된 벡터입니다.
- 중심이 가장 가까운 클러스터에 각 관측치를 할당합니다. 여기서 가장 가까운 것은 유클리드 거리를 사용하여 정의됩니다.
기술 노트:
k-medoids는 평균이 아닌 중앙값을 사용하여 군집 중심을 계산하므로 k-평균보다 이상값에 더 강력한 경향이 있습니다.
실제로 데이터 세트에 극단적인 이상값이 없으면 k-평균과 k-medoids는 유사한 결과를 생성합니다.
R의 K-Medoids 클러스터링
다음 튜토리얼에서는 R에서 k-medoids 클러스터링을 수행하는 방법에 대한 단계별 예를 제공합니다.
1단계: 필요한 패키지 로드
먼저 R의 k-medoids 클러스터링에 대한 몇 가지 유용한 기능이 포함된 두 개의 패키지를 로드합니다.
library (factoextra) library (cluster)
2단계: 데이터 로드 및 준비
이 예에서는 R에 내장된 USArrests 데이터세트를 사용합니다. 여기에는 1973년 미국 각 주에서 살인 , 폭행 , 강간 으로 체포된 건수와 도시에 거주하는 각 주의 인구 비율이 포함되어 있습니다. 지역. , 어반팝 .
다음 코드는 다음 작업을 수행하는 방법을 보여줍니다.
- USArrests 데이터 세트 로드
- 누락된 값이 있는 모든 행을 제거합니다.
- 평균이 0이고 표준편차가 1이 되도록 데이터 세트의 각 변수를 조정합니다.
#load data df <-USArrests #remove rows with missing values df <- na. omitted (df) #scale each variable to have a mean of 0 and sd of 1 df <- scale(df) #view first six rows of dataset head(df) Murder Assault UrbanPop Rape Alabama 1.24256408 0.7828393 -0.5209066 -0.003416473 Alaska 0.50786248 1.1068225 -1.2117642 2.484202941 Arizona 0.07163341 1.4788032 0.9989801 1.042878388 Arkansas 0.23234938 0.2308680 -1.0735927 -0.184916602 California 0.27826823 1.2628144 1.7589234 2.067820292 Colorado 0.02571456 0.3988593 0.8608085 1.864967207
3단계: 최적의 클러스터 수 찾기
R에서 k-medoid 클러스터링을 수행하려면 “중앙값을 중심으로 분할”을 의미하고 다음 구문을 사용하는 pam() 함수를 사용할 수 있습니다.
pam(data, k, 메트릭 = “유클리드”, 스탠드 = FALSE)
금:
- 데이터: 데이터 세트의 이름입니다.
- k: 클러스터 수입니다.
- 미터법: 거리를 계산하는 데 사용할 미터법입니다. 기본값은 Euclidean 이지만 manhattan 을 지정할 수도 있습니다.
- 스탠드: 데이터 세트의 각 변수를 정규화할지 여부입니다. 기본값은 false입니다.
최적의 클러스터 수를 미리 알 수 없으므로 결정하는 데 도움이 될 수 있는 두 가지 그래프를 만듭니다.
1. 전체 제곱합에 대한 클러스터 수
먼저 fviz_nbclust() 함수를 사용하여 클러스터 수와 제곱합의 합계에 대한 플롯을 만듭니다.
fviz_nbclust(df, pam, method = “ wss ”)
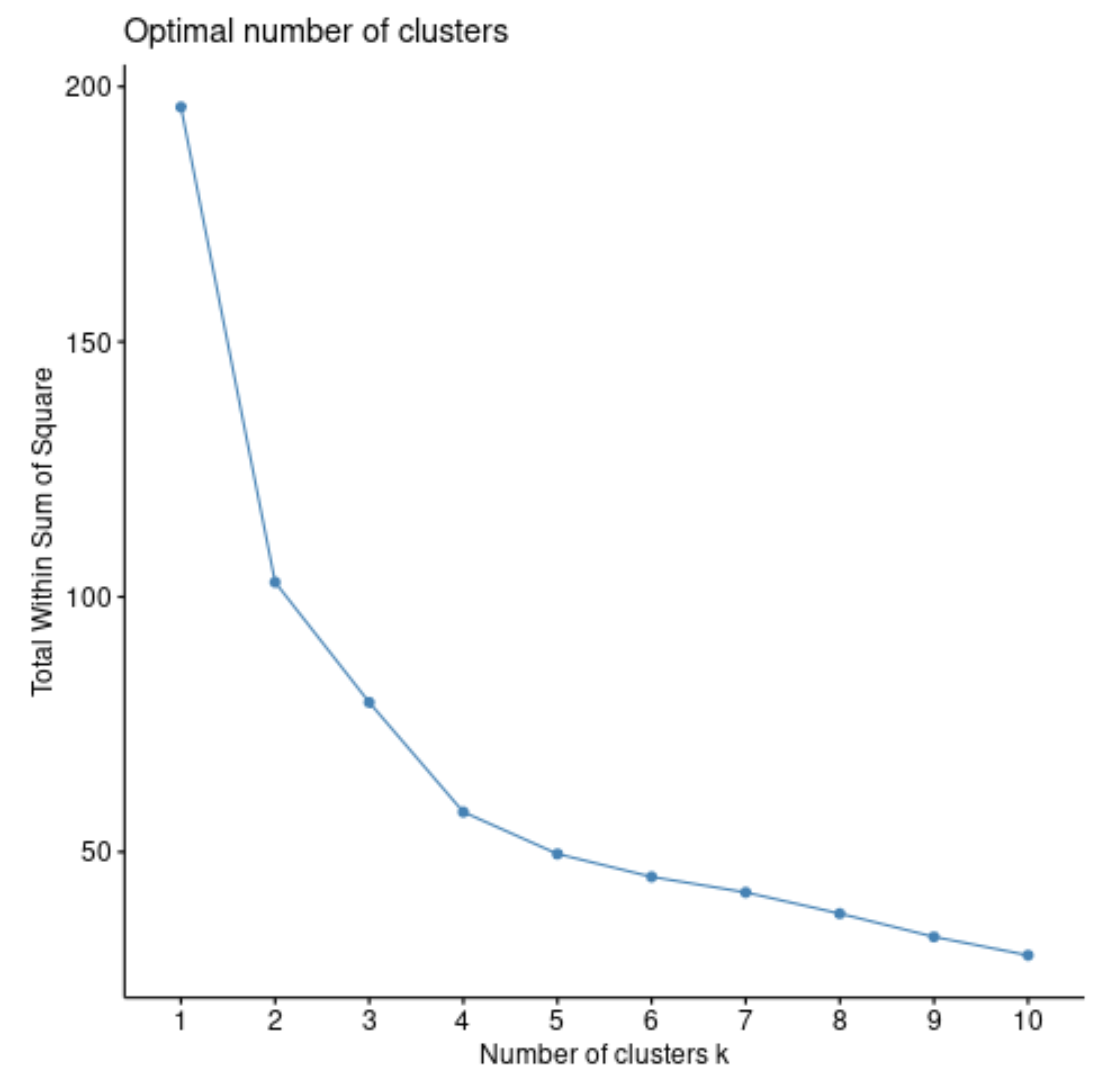
제곱합의 합계는 일반적으로 클러스터 수를 늘리면 항상 증가합니다. 따라서 이러한 유형의 플롯을 만들 때 제곱합이 “구부러지거나” 수평을 이루기 시작하는 “무릎”을 찾습니다.
플롯의 곡률 지점은 일반적으로 최적의 클러스터 수에 해당합니다. 이 수치를 넘어서면 과적합이 발생할 가능성이 높습니다.
이 그래프의 경우 k = 4개 클러스터에 작은 꼬임 또는 “굴곡”이 있는 것으로 나타납니다.
2. 군집 수와 격차 통계
최적의 클러스터 수를 결정하는 또 다른 방법은 다양한 k 값에 대한 전체 클러스터 내 변동을 클러스터링이 없는 분포에 대한 예상 값과 비교하는 편차 통계 라는 측정항목을 사용하는 것입니다.
클러스터 패키지의 clusGap() 함수를 사용하여 각 클러스터 수에 대한 간격 통계를 계산할 수 있을 뿐만 아니라 fviz_gap_stat() 함수를 사용하여 클러스터 대 간격 통계의 플롯을 계산할 수 있습니다.
#calculate gap statistic based on number of clusters gap_stat <- clusGap(df, FUN = pam, K.max = 10, #max clusters to consider B = 50) #total bootstrapped iterations #plot number of clusters vs. gap statistic fviz_gap_stat(gap_stat)
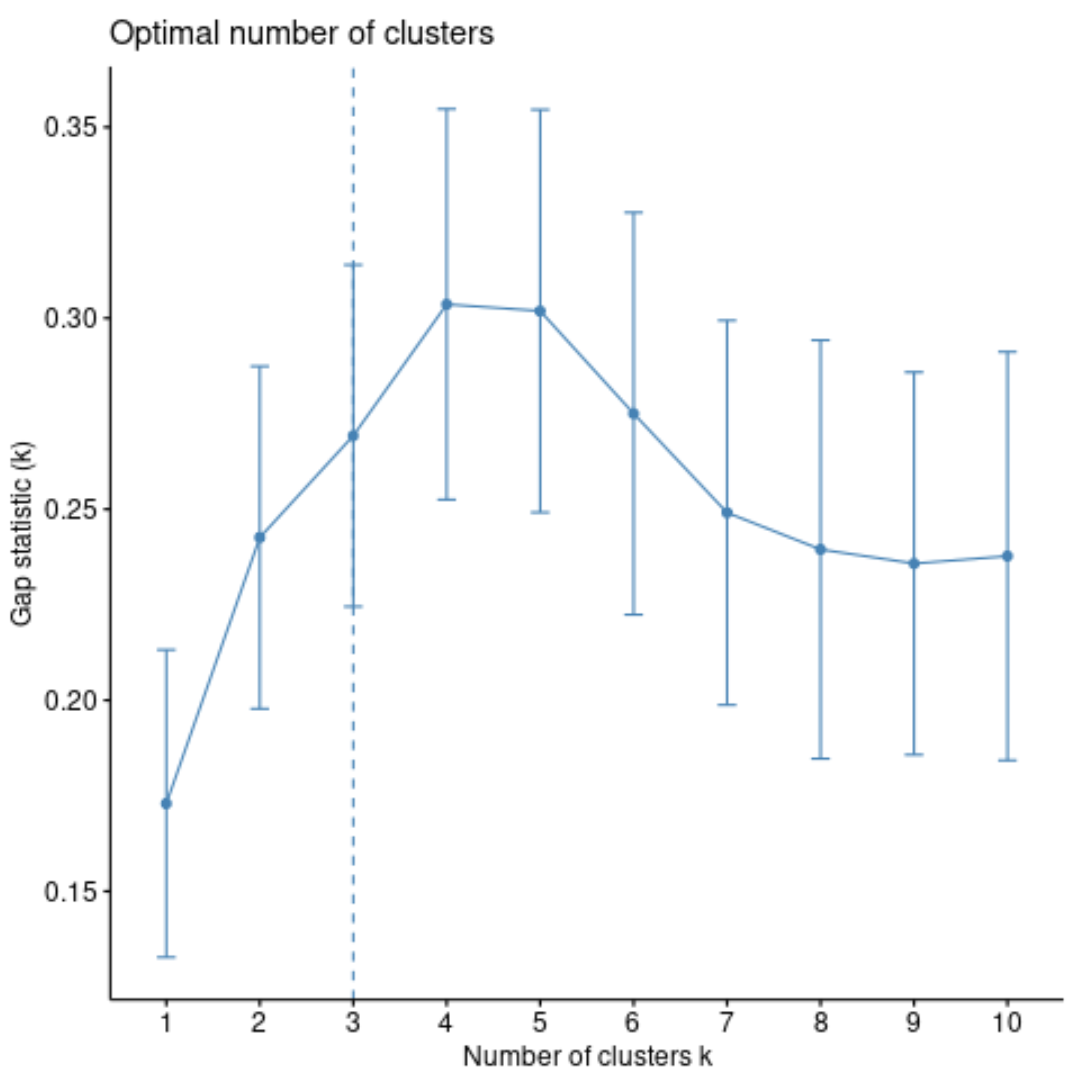
그래프에서 간격 통계는 k = 4 클러스터에서 가장 높다는 것을 알 수 있으며 이는 이전에 사용한 팔꿈치 방법에 해당합니다.
4단계: Optimal K를 사용하여 K-Medoids 클러스터링 수행
마지막으로 k -4에 대한 최적 값을 사용하여 데이터 세트에서 k-medoids 클러스터링을 수행할 수 있습니다.
#make this example reproducible set.seed(1) #perform k-medoids clustering with k = 4 clusters kmed <- pam(df, k = 4) #view results kmed ID Murder Assault UrbanPop Rape Alabama 1 1.2425641 0.7828393 -0.5209066 -0.003416473 Michigan 22 0.9900104 1.0108275 0.5844655 1.480613993 Oklahoma 36 -0.2727580 -0.2371077 0.1699510 -0.131534211 New Hampshire 29 -1.3059321 -1.3650491 -0.6590781 -1.252564419 Vector clustering: Alabama Alaska Arizona Arkansas California 1 2 2 1 2 Colorado Connecticut Delaware Florida Georgia 2 3 3 2 1 Hawaii Idaho Illinois Indiana Iowa 3 4 2 3 4 Kansas Kentucky Louisiana Maine Maryland 3 3 1 4 2 Massachusetts Michigan Minnesota Mississippi Missouri 3 2 4 1 3 Montana Nebraska Nevada New Hampshire New Jersey 3 3 2 4 3 New Mexico New York North Carolina North Dakota Ohio 2 2 1 4 3 Oklahoma Oregon Pennsylvania Rhode Island South Carolina 3 3 3 3 1 South Dakota Tennessee Texas Utah Vermont 4 1 2 3 4 Virginia Washington West Virginia Wisconsin Wyoming 3 3 4 4 3 Objective function: build swap 1.035116 1.027102 Available components: [1] "medoids" "id.med" "clustering" "objective" "isolation" [6] "clusinfo" "silinfo" "diss" "call" "data"
4개의 클러스터 중심은 모두 데이터 세트의 실제 관측치입니다. 출력 상단 근처에서 4개의 중심이 다음과 같은 상태임을 확인할 수 있습니다.
- 앨라배마
- 미시간
- 오클라호마
- 뉴햄프셔
fivz_cluster() 함수를 사용하여 축에 처음 두 개의 주성분을 표시하는 산점도에서 클러스터를 시각화할 수 있습니다.
#plot results of final k-medoids model
fviz_cluster(kmed, data = df)
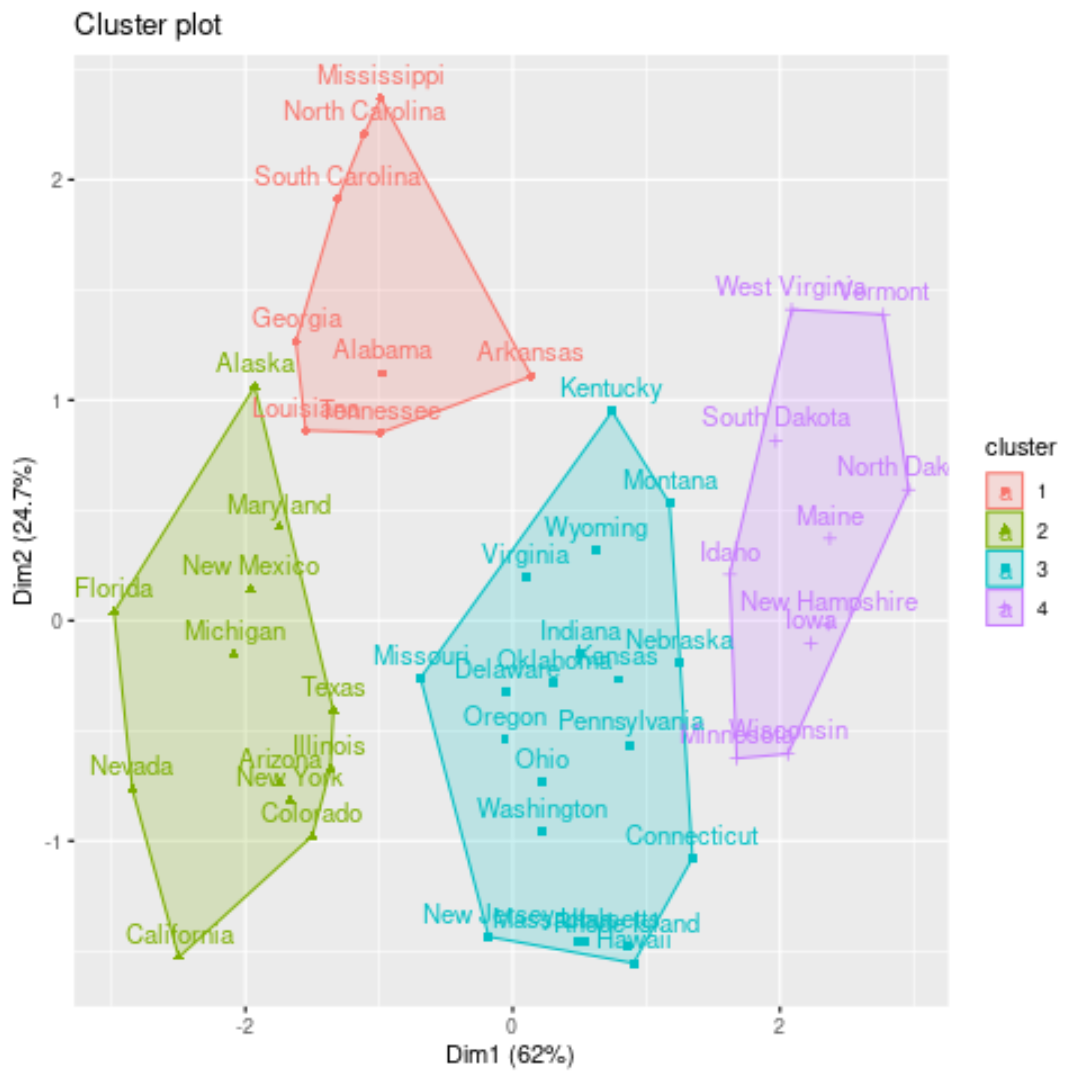
각 주의 클러스터 할당을 원본 데이터세트에 추가할 수도 있습니다.
#add cluster assignment to original data
final_data <- cbind(USArrests, cluster = kmed$cluster)
#view final data
head(final_data)
Murder Assault UrbanPop Rape cluster
Alabama 13.2 236 58 21.2 1
Alaska 10.0 263 48 44.5 2
Arizona 8.1 294 80 31.0 2
Arkansas 8.8 190 50 19.5 1
California 9.0 276 91 40.6 2
Colorado 7.9 204 78 38.7 2
이 예제에 사용된 전체 R 코드는 여기에서 찾을 수 있습니다.