R에서 로그 순위 테스트를 수행하는 방법
로그 순위 테스트는 두 그룹 간의 생존 곡선을 비교하는 가장 일반적인 방법입니다.
이 테스트에서는 다음과 같은 가정을 사용합니다.
H 0 : 두 그룹간 생존율에는 차이가 없다.
H A : 두 그룹의 생존율에는 차이가 있습니다 .
검정의 p-값이 특정 유의 수준(예: α = 0.05)보다 낮으면 귀무 가설을 기각하고 두 그룹 간의 생존율에 차이가 있다고 말할 수 있는 충분한 증거가 있다는 결론을 내릴 수 있습니다.
R에서 로그 순위 테스트를 수행하려면 다음 구문을 사용하는 생존 패키지의 survdiff() 함수를 사용할 수 있습니다.
survdiff(Surv(시간, 상태) ~ 예측변수, 데이터)
이 함수는 카이제곱 검정 통계량과 해당 p-값을 반환합니다.
다음 예에서는 이 함수를 사용하여 R에서 로그 순위 테스트를 수행하는 방법을 보여줍니다.
예: R의 로그 순위 테스트
이 예에서는 생존 패키지의 난소 데이터 세트를 사용합니다. 이 데이터 세트에는 26명의 환자에 대한 다음 정보가 포함되어 있습니다.
- 난소암 진단 후 생존기간(개월)
- 생존시간 검열 여부
- 받은 치료 유형(rx = 1 또는 rx = 2)
다음 코드는 이 데이터세트의 처음 6개 행을 표시하는 방법을 보여줍니다.
library (survival) #view first six rows of dataset head(ovarian) futime fustat age resid.ds rx ecog.ps 1 59 1 72.3315 2 1 1 2 115 1 74.4932 2 1 1 3 156 1 66.4658 2 1 2 4 421 0 53.3644 2 2 1 5,431 1 50.3397 2 1 1 6 448 0 56.4301 1 1 2
다음 코드는 서로 다른 치료를 받은 환자 간의 생존율에 차이가 있는지 확인하기 위해 로그 순위 테스트를 수행하는 방법을 보여줍니다.
#perform log rank test
survdiff(Surv(futime, fustat) ~ rx, data=ovarian)
Call:
survdiff(formula = Surv(futime, fustat) ~ rx, data = ovarian)
N Observed Expected (OE)^2/E (OE)^2/V
rx=1 13 7 5.23 0.596 1.06
rx=2 13 5 6.77 0.461 1.06
Chisq= 1.1 on 1 degrees of freedom, p= 0.3
카이제곱 검정 통계량은 자유도가 1인 1.1 이고 해당 p-값은 0.3 입니다. 이 p-값은 0.05 이상이므로 귀무가설을 기각할 수 없습니다.
즉, 두 치료법 사이에 통계적으로 유의한 생존율 차이가 있다고 말할 만큼 충분한 근거가 없습니다.
다음 구문을 사용하여 각 그룹의 생존 곡선을 그릴 수도 있습니다.
#plot survival curves for each treatment group plot(survfit(Surv(futime, fustat) ~ rx, data = ovarian), xlab = " Time ", ylab = “ Overall survival probability ”)
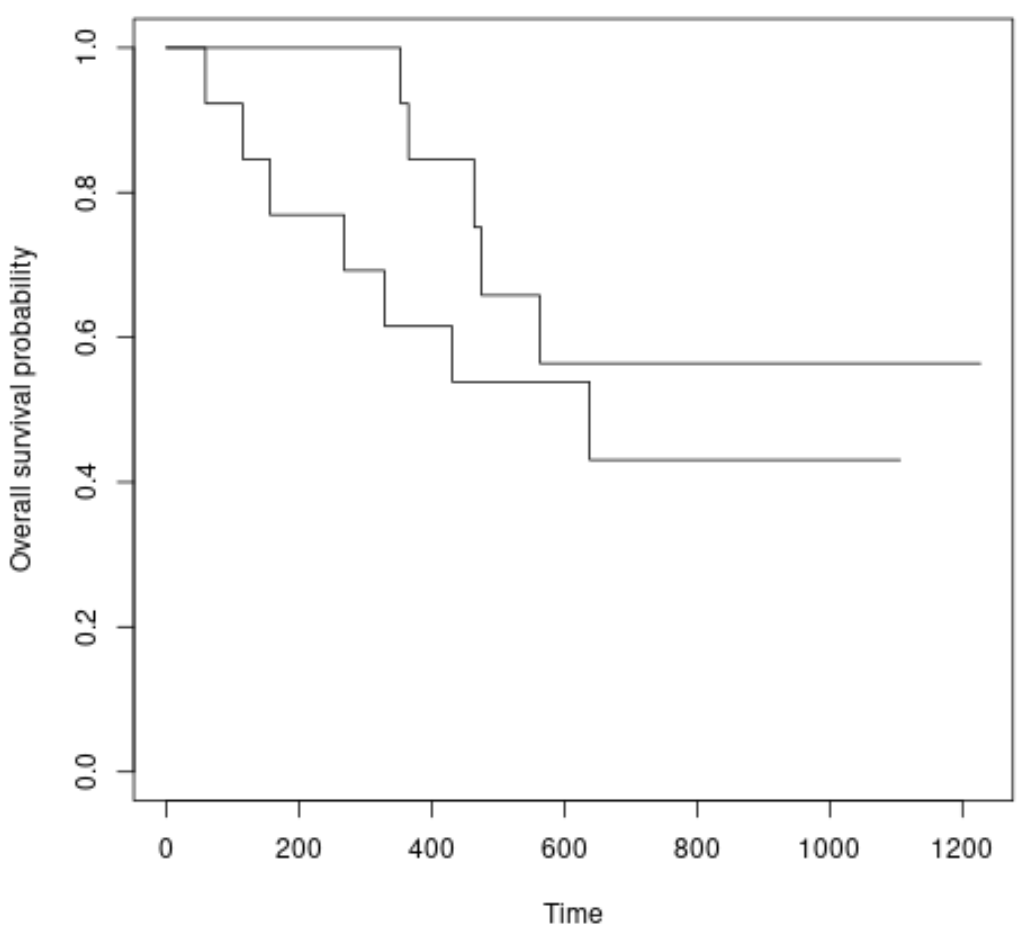
생존 곡선이 약간씩 다른 것을 볼 수 있지만, 로그 순위 테스트를 통해 그 차이가 통계적으로 유의하지 않은 것으로 나타났습니다.