R의 주성분 회귀(단계별)
p 개의 예측 변수 세트와 응답 변수가 주어지면 다중 선형 회귀 분석 에서는 RSS(잔차 제곱합)를 최소화하기 위해 최소 제곱이라는 방법을 사용합니다.
RSS = Σ(y i – ŷ i ) 2
금:
- Σ : 합계를 의미하는 그리스 기호
- y i : i번째 관측값에 대한 실제 응답 값
- ŷ i : 다중선형회귀모델을 기반으로 예측된 반응값
그러나 예측변수의 상관관계가 높으면 다중공선성이 문제가 될 수 있습니다. 이로 인해 모델 계수 추정이 신뢰할 수 없게 되고 높은 분산이 나타날 수 있습니다.
이 문제를 피하는 한 가지 방법은 주성분 회귀를 사용하는 것입니다. 이는 원래 p 예측 변수의 M개의 선형 조합(“주성분”이라고 함)을 찾은 다음 최소 제곱을 사용하여 주성분을 예측 변수로 사용하여 선형 회귀 모델을 적합시킵니다.
이 튜토리얼에서는 R에서 주성분 회귀를 수행하는 방법에 대한 단계별 예를 제공합니다.
1단계: 필요한 패키지 로드
R에서 주성분 회귀를 수행하는 가장 쉬운 방법은 pls 패키지의 함수를 사용하는 것입니다.
#install pls package (if not already installed) install.packages(" pls ") load pls package library(pls)
2단계: PCR 모델 조정
이 예에서는 다양한 유형의 자동차에 대한 데이터가 포함된 mtcars 라는 내장 R 데이터 세트를 사용합니다.
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
이 예에서는 hp를 반응 변수 로 사용하고 다음 변수를 예측 변수로 사용하여 주성분 회귀(PCR) 모델을 적합합니다.
- mpg
- 표시하다
- 똥
- 무게
- q초
다음 코드는 PCR 모델을 이 데이터에 맞추는 방법을 보여줍니다. 다음 인수에 유의하세요.
- scale=TRUE : 이는 각 예측 변수가 평균 0, 표준 편차 1을 갖도록 스케일링되어야 함을 R에 지시합니다. 이렇게 하면 예측 변수가 다른 단위로 측정되는 경우 모델에 너무 많은 영향을 미치지 않도록 합니다. .
- 검증=”CV” : R에게 k-겹 교차 검증을 사용하여 모델 성능을 평가하도록 지시합니다. 이는 기본적으로 k=10 접기를 사용한다는 점에 유의하세요. 또한 Leave-One-Out 교차 검증을 수행하는 대신 “LOOCV”를 지정할 수도 있습니다.
#make this example reproducible set.seed(1) #fit PCR model model <- pcr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
3단계: 주요 구성 요소 수 선택
모델을 조정한 후에는 유지할 가치가 있는 주성분 수를 결정해야 합니다.
이를 수행하려면 k-교차 검증으로 계산된 테스트 평균 제곱근 오차(테스트 RMSE)를 살펴보세요.
#view summary of model fitting
summary(model)
Data:
Y dimension: 32 1
Fit method: svdpc
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comp 2 comps 3 comps 4 comps 5 comps
CV 69.66 44.56 35.64 35.83 36.23 36.67
adjCV 69.66 44.44 35.27 35.43 35.80 36.20
TRAINING: % variance explained
1 comp 2 comps 3 comps 4 comps 5 comps
X 69.83 89.35 95.88 98.96 100.00
hp 62.38 81.31 81.96 81.98 82.03
결과에는 두 가지 흥미로운 테이블이 있습니다.
1. 검증: RMSEP
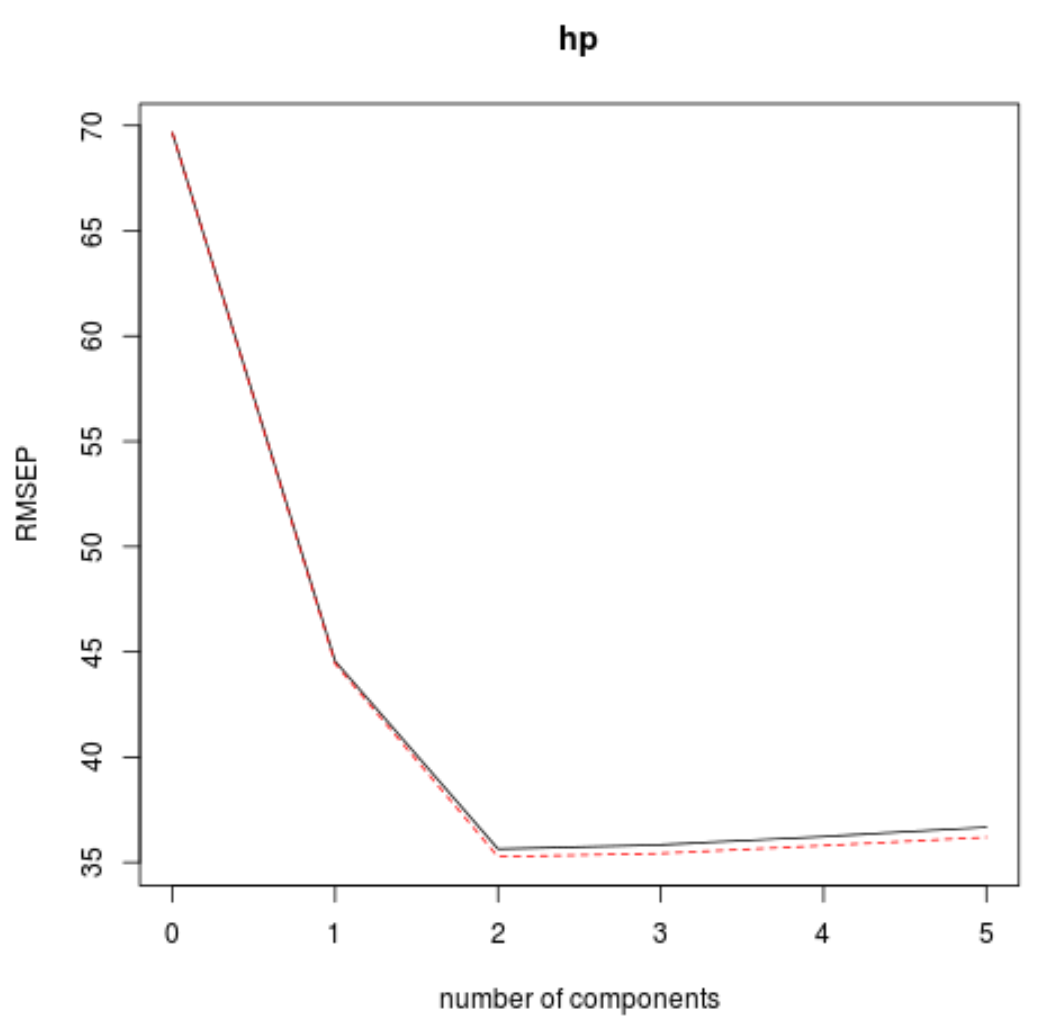
이 표는 k-겹 교차 검증으로 계산된 RMSE 테스트를 보여줍니다. 우리는 다음을 볼 수 있습니다:
- 모델에서 원래 항만 사용하는 경우 테스트의 RMSE는 69.66 입니다.
- 첫 번째 주성분을 추가하면 RMSE 테스트는 44.56으로 떨어집니다.
- 두 번째 주성분을 추가하면 RMSE 테스트는 35.64로 떨어집니다.
추가 주성분을 추가하면 실제로 테스트의 RMSE가 증가하는 것을 볼 수 있습니다. 따라서 최종 모델에서는 두 개의 주성분만 사용하는 것이 최적인 것으로 보입니다.
2. 훈련: 설명된 분산 %
이 표는 주성분에 의해 설명되는 반응 변수의 분산 비율을 보여줍니다. 우리는 다음을 볼 수 있습니다:
- 첫 번째 주성분만 사용하여 반응 변수 변동의 69.83% 를 설명할 수 있습니다.
- 두 번째 주성분을 추가하면 반응 변수 변동의 89.35% 를 설명할 수 있습니다.
더 많은 주성분을 사용하여 더 많은 분산을 설명할 수 있지만, 두 개 이상의 주성분을 추가해도 실제로 설명된 분산의 비율이 크게 증가하지 않는다는 것을 알 수 있습니다.
또한 검증 플롯() 함수를 사용하여 RMSE 테스트(MSE 및 R-제곱 테스트와 함께)를 주성분 수의 함수로 시각화할 수도 있습니다.
#visualize cross-validation plots
validationplot(model)
validationplot(model, val.type="MSEP")
validationplot(model, val.type="R2")
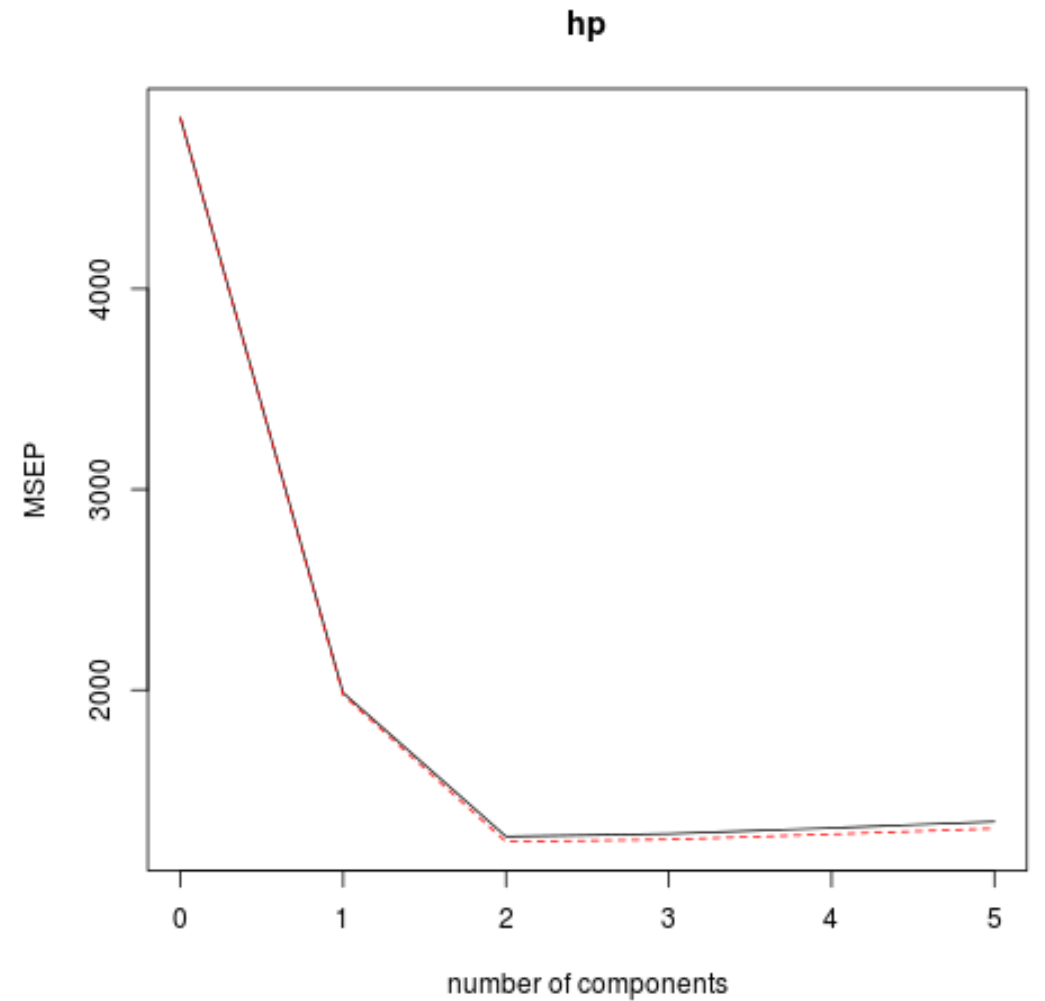
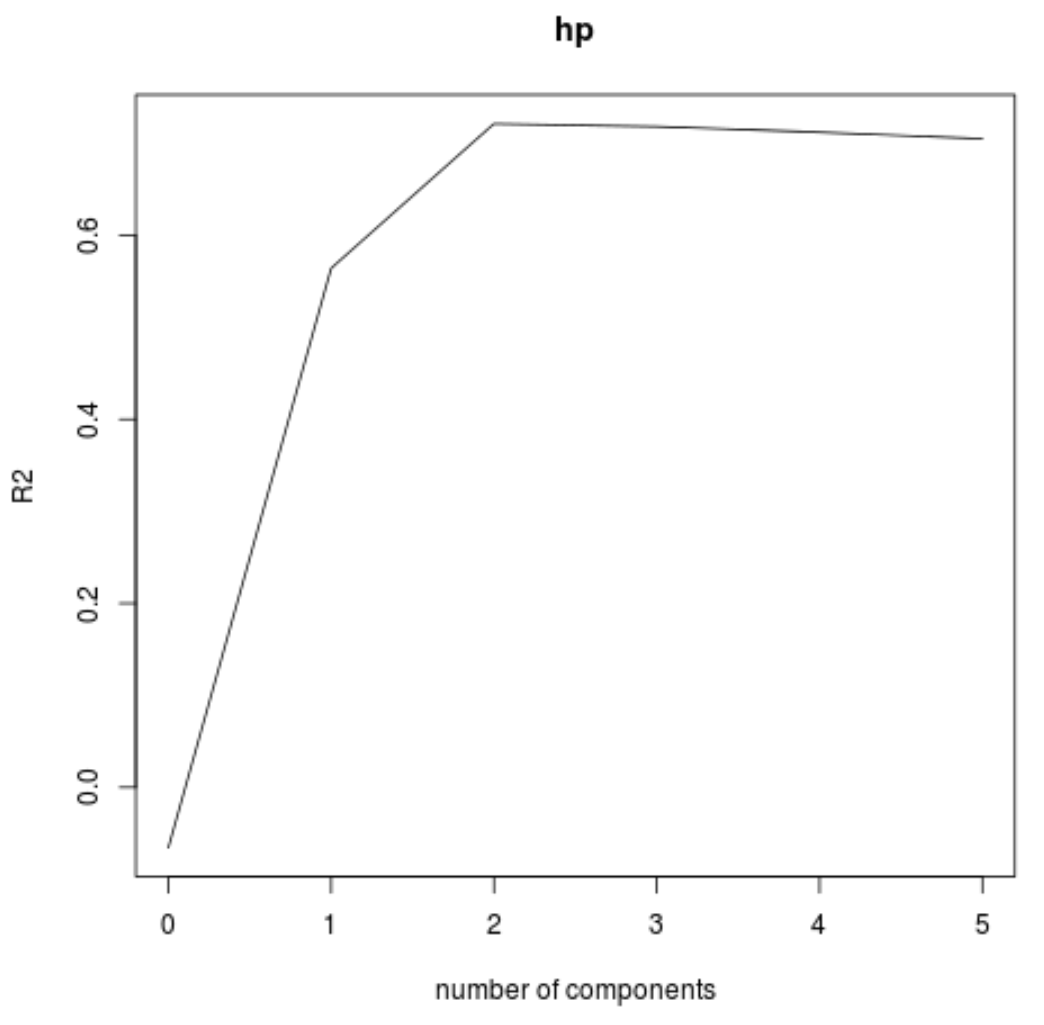
각 그래프에서 두 개의 주성분을 추가하면 모형 적합도가 향상되지만 더 많은 주성분을 추가하면 성능이 저하되는 경향이 있음을 알 수 있습니다.
따라서 최적 모델에는 처음 두 개의 주성분만 포함됩니다.
4단계: 최종 모델을 사용하여 예측
최종 2주성분 PCR 모델을 사용하여 새로운 관찰에 대한 예측을 할 수 있습니다.
다음 코드는 원본 데이터 세트를 훈련 세트와 테스트 세트로 분할하고 두 가지 주요 구성 요소가 있는 PCR 모델을 사용하여 테스트 세트에 대해 예측하는 방법을 보여줍니다.
#define training and testing sets train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")] y_test <- mtcars[26: nrow (mtcars), c("hp")] test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")] #use model to make predictions on a test set model <- pcr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ") pcr_pred <- predict(model, test, ncomp= 2 ) #calculate RMSE sqrt ( mean ((pcr_pred - y_test)^2)) [1] 56.86549
테스트의 RMSE는 56.86549 로 나타났습니다. 이는 테스트 세트 관찰에 대해 예측된 HP 값과 관찰된 HP 값 사이의 평균 편차입니다.
이 예제에서 R 코드의 전체 사용법은 여기에서 확인할 수 있습니다.