Hoe te bootstrapping in r (met voorbeelden)
Bootstrapping is een methode die kan worden gebruikt om de standaardfout van een statistiek te schatten en een betrouwbaarheidsinterval voor de statistiek te genereren.
Het basisproces voor bootstrapping is als volgt:
- Neem k replicaatmonsters met vervanging uit een gegeven dataset.
- Bereken voor elk monster de betreffende statistiek.
- Dit levert k verschillende schattingen op voor een bepaalde statistiek, die u vervolgens kunt gebruiken om de standaardfout van de statistiek te berekenen en een betrouwbaarheidsinterval voor de statistiek te creëren.
We kunnen in R opstarten met behulp van de volgende functies uit de bootstrapbibliotheek :
1. Genereer bootstrap-voorbeelden.
boot(gegevens, statistieken, R, …)
Goud:
- gegevens: een vector, matrix of blok gegevens
- statistiek: een functie die de te starten statistiek(en) produceert
- A: Aantal bootstrap-herhalingen
2. Genereer een bootstrap-betrouwbaarheidsinterval.
boot.ci(opstartobject, conf, type)
Goud:
- bootobject: een object dat wordt geretourneerd door de functie boot().
- conf: het betrouwbaarheidsinterval dat moet worden berekend. De standaardwaarde is 0,95
- type: Type betrouwbaarheidsinterval dat moet worden berekend. Opties omvatten ’standaard‘, ‚basic‘, ’stud‘, ‚perc‘, ‚bca‘ en ‚all‘ – standaard is ‚all‘
De volgende voorbeelden laten zien hoe u deze functies in de praktijk kunt gebruiken.
Voorbeeld 1: start een enkele statistiek
De volgende code laat zien hoe u de standaardfout voor het R-kwadraat van een eenvoudig lineair regressiemodel kunt berekenen:
set.seed(0) library (boot) #define function to calculate R-squared rsq_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (summary(fit)$r.square) #return R-squared of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=rsq_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = rsq_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 0.7183433 0.002164339 0.06513426
Uit de resultaten kunnen we zien:
- De geschatte R-kwadraat voor dit regressiemodel is 0,7183433 .
- De standaardfout voor deze schatting is 0,06513426 .
We kunnen ook snel de distributie van bootstrapped samples visualiseren:
plot(reps)
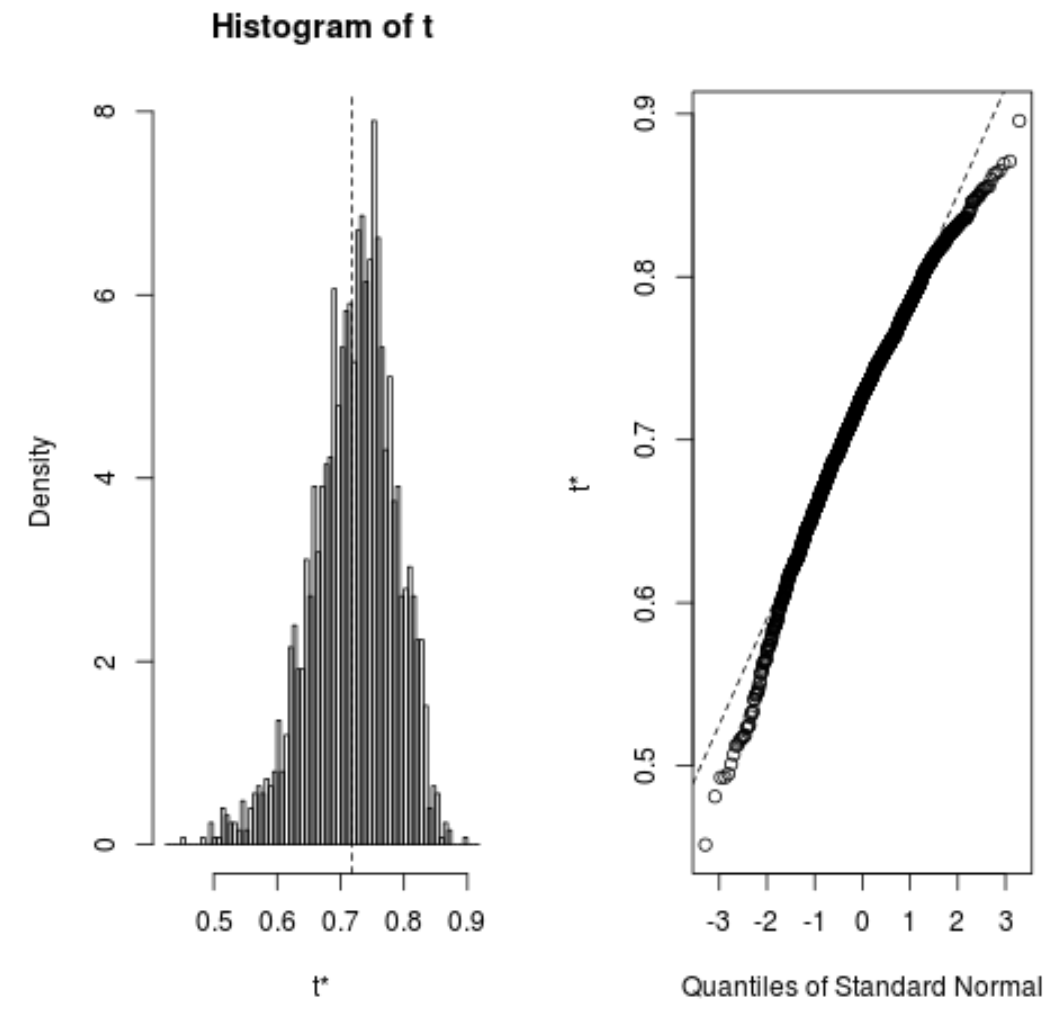
We kunnen ook de volgende code gebruiken om het 95% betrouwbaarheidsinterval voor het geschatte R-kwadraat van het model te berekenen:
#calculate adjusted bootstrap percentile (BCa) interval boot.ci(reps, type=" bca ") CALL: boot.ci(boot.out = reps, type = "bca") Intervals: Level BCa 95% (0.5350, 0.8188) Calculations and Intervals on Original Scale
Uit het resultaat kunnen we zien dat het bootstrapped-betrouwbaarheidsinterval van 95% voor de echte R-kwadraatwaarden (0,5350, 0,8188) is.
Voorbeeld 2: meerdere statistieken opstarten
De volgende code laat zien hoe u de standaardfout voor elke coëfficiënt in een meervoudig lineair regressiemodel kunt berekenen:
set.seed(0) library (boot) #define function to calculate fitted regression coefficients coef_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (coef(fit)) #return coefficient estimates of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=coef_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = coef_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 29.59985476 -5.058601e-02 1.49354577 t2* -0.04121512 6.549384e-05 0.00527082
Uit de resultaten kunnen we zien:
- De geschatte coëfficiënt voor het modelonderschepping is 29,59985476 en de standaardfout van deze schatting is 1,49354577 .
- De geschatte coëfficiënt voor de voorspellende variabele disp in het model is -0,04121512 en de standaardfout van deze schatting is 0,00527082 .
We kunnen ook snel de distributie van bootstrapped samples visualiseren:
plot(reps, index=1) #intercept of model plot(reps, index=2) #disp predictor variable
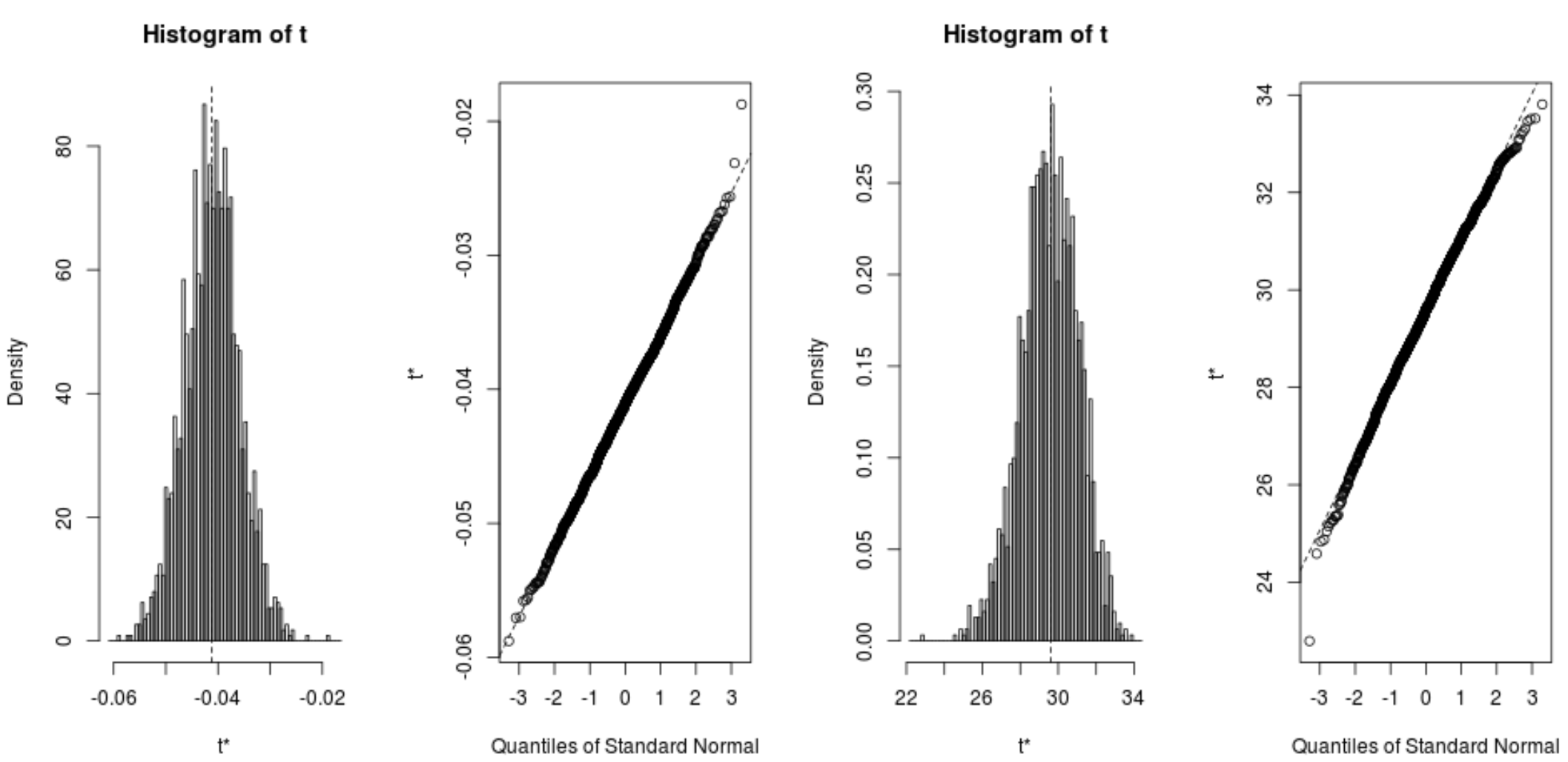
We kunnen ook de volgende code gebruiken om de 95%-betrouwbaarheidsintervallen voor elke coëfficiënt te berekenen:
#calculate adjusted bootstrap percentile (BCa) intervals boot.ci(reps, type=" bca ", index=1) #intercept of model boot.ci(reps, type=" bca ", index=2) #disp predictor variable CALL: boot.ci(boot.out = reps, type = "bca", index = 1) Intervals: Level BCa 95% (26.78, 32.66) Calculations and Intervals on Original Scale BOOTSTRAP CONFIDENCE INTERVAL CALCULATIONS Based on 2000 bootstrap replicates CALL: boot.ci(boot.out = reps, type = "bca", index = 2) Intervals: Level BCa 95% (-0.0520, -0.0312) Calculations and Intervals on Original Scale
Uit de resultaten kunnen we zien dat de bootstrapped 95%-betrouwbaarheidsintervallen voor de modelcoëfficiënten als volgt zijn:
- IC voor onderschepping: (26.78, 32.66)
- CI voor weergave : (-.0520, -.0312)
Aanvullende bronnen
Hoe eenvoudige lineaire regressie uit te voeren in R
Hoe meervoudige lineaire regressie uit te voeren in R
Inleiding tot betrouwbaarheidsintervallen