K-medoids in r: stapsgewijs voorbeeld
Clustering is een machine learning-techniek die probeert groepen of clusters van observaties binnen een dataset te vinden.
Het doel is om clusters zo te vinden dat waarnemingen binnen elke cluster behoorlijk op elkaar lijken, terwijl waarnemingen in verschillende clusters behoorlijk van elkaar verschillen.
Clustering is een vorm van leren zonder toezicht , omdat we eenvoudigweg structuur proberen te vinden binnen een dataset in plaats van de waarde van een responsvariabele te voorspellen.
Clustering wordt vaak gebruikt in marketing wanneer bedrijven toegang hebben tot informatie zoals:
- Huishoudelijk inkomen
- Grootte van het huishouden
- Beroep hoofd huishouden
- Afstand tot het dichtstbijzijnde stedelijke gebied
Wanneer deze informatie beschikbaar is, kan clustering worden gebruikt om huishoudens te identificeren die vergelijkbaar zijn en waarbij de kans groter is dat ze bepaalde producten kopen of beter reageren op een bepaald soort reclame.
Een van de meest voorkomende vormen van clustering staat bekend als k-means clustering .
Helaas kan deze methode worden beïnvloed door uitschieters, daarom is k-medoids clustering een vaak gebruikt alternatief.
Wat is K-Medoids-clustering?
K-medoids-clustering is een techniek waarbij we elke waarneming in een dataset in een van de K- clusters plaatsen.
Het einddoel is om K- clusters te hebben waarin waarnemingen binnen elke cluster behoorlijk op elkaar lijken, terwijl waarnemingen in verschillende clusters behoorlijk van elkaar verschillen.
In de praktijk gebruiken we de volgende stappen om K-means clustering uit te voeren:
1. Kies een waarde voor K.
- Eerst moeten we beslissen hoeveel clusters we in de gegevens willen identificeren. Vaak hoeven we eenvoudigweg verschillende waarden voor K te testen en de resultaten te analyseren om te zien welk aantal clusters het meest logisch lijkt voor een bepaald probleem.
2. Wijs elke waarneming willekeurig toe aan een eerste cluster, van 1 tot K.
3. Voer de volgende procedure uit totdat de clustertoewijzingen niet meer veranderen.
- Bereken voor elk van de K- clusters het zwaartepunt van het cluster. Dit is de vector van de p- medianen van de kenmerken voor de waarnemingen van de k -de cluster.
- Wijs elke waarneming toe aan het cluster met het dichtstbijzijnde zwaartepunt. Hier wordt de dichtstbijzijnde gedefinieerd met behulp van de Euclidische afstand .
Technische notitie:
Omdat k-medoids clusterzwaartepunten berekent met behulp van medianen in plaats van gemiddelden, is het doorgaans robuuster voor uitschieters dan k-gemiddelden.
In de praktijk zullen k-means en k-medoids vergelijkbare resultaten opleveren als er geen extreme uitschieters in de dataset voorkomen.
K-Medoids clusteren in R
De volgende zelfstudie biedt een stapsgewijs voorbeeld van het uitvoeren van k-medoids-clustering in R.
Stap 1: Laad de benodigde pakketten
Eerst zullen we twee pakketten laden die verschillende nuttige functies bevatten voor k-medoids-clustering in R.
library (factoextra) library (cluster)
Stap 2: Gegevens laden en voorbereiden
Voor dit voorbeeld gebruiken we de USArrests- gegevensset die is ingebouwd in R, die het aantal arrestaties per 100.000 mensen in elke Amerikaanse staat in 1973 bevat voor moord , aanranding en verkrachting , evenals het percentage van de bevolking van elke staat dat in stedelijke gebieden woont. gebieden. , UrbanPop .
De volgende code laat zien hoe u het volgende kunt doen:
- Laad USArrests- gegevensset
- Verwijder alle rijen met ontbrekende waarden
- Schaal elke variabele in de dataset zodat deze een gemiddelde van 0 en een standaarddeviatie van 1 heeft
#load data df <-USArrests #remove rows with missing values df <- na. omitted (df) #scale each variable to have a mean of 0 and sd of 1 df <- scale(df) #view first six rows of dataset head(df) Murder Assault UrbanPop Rape Alabama 1.24256408 0.7828393 -0.5209066 -0.003416473 Alaska 0.50786248 1.1068225 -1.2117642 2.484202941 Arizona 0.07163341 1.4788032 0.9989801 1.042878388 Arkansas 0.23234938 0.2308680 -1.0735927 -0.184916602 California 0.27826823 1.2628144 1.7589234 2.067820292 Colorado 0.02571456 0.3988593 0.8608085 1.864967207
Stap 3: Vind het optimale aantal clusters
Om k-medoid-clustering in R uit te voeren, kunnen we de pam()- functie gebruiken, wat staat voor “partitionering rond medianen” en de volgende syntaxis gebruikt:
pam(data, k, metrisch = “Euclidisch”, stand = ONWAAR)
Goud:
- gegevens: naam van de gegevensset.
- k: het aantal clusters.
- metriek: de metriek die moet worden gebruikt om de afstand te berekenen. De standaardwaarde is Euclidisch , maar u kunt ook manhattan opgeven.
- stand: Het al dan niet normaliseren van elke variabele in de dataset. De standaardwaarde is false.
Omdat we van tevoren niet weten welk aantal clusters optimaal is, maken we twee verschillende grafieken die ons kunnen helpen beslissen:
1. Aantal clusters ten opzichte van totaal in som van kwadraten
Eerst gebruiken we de functie fviz_nblust() om een grafiek te maken van het aantal clusters versus het totaal in de som van de kwadraten:
fviz_nbclust(df, pam, method = “ wss ”)
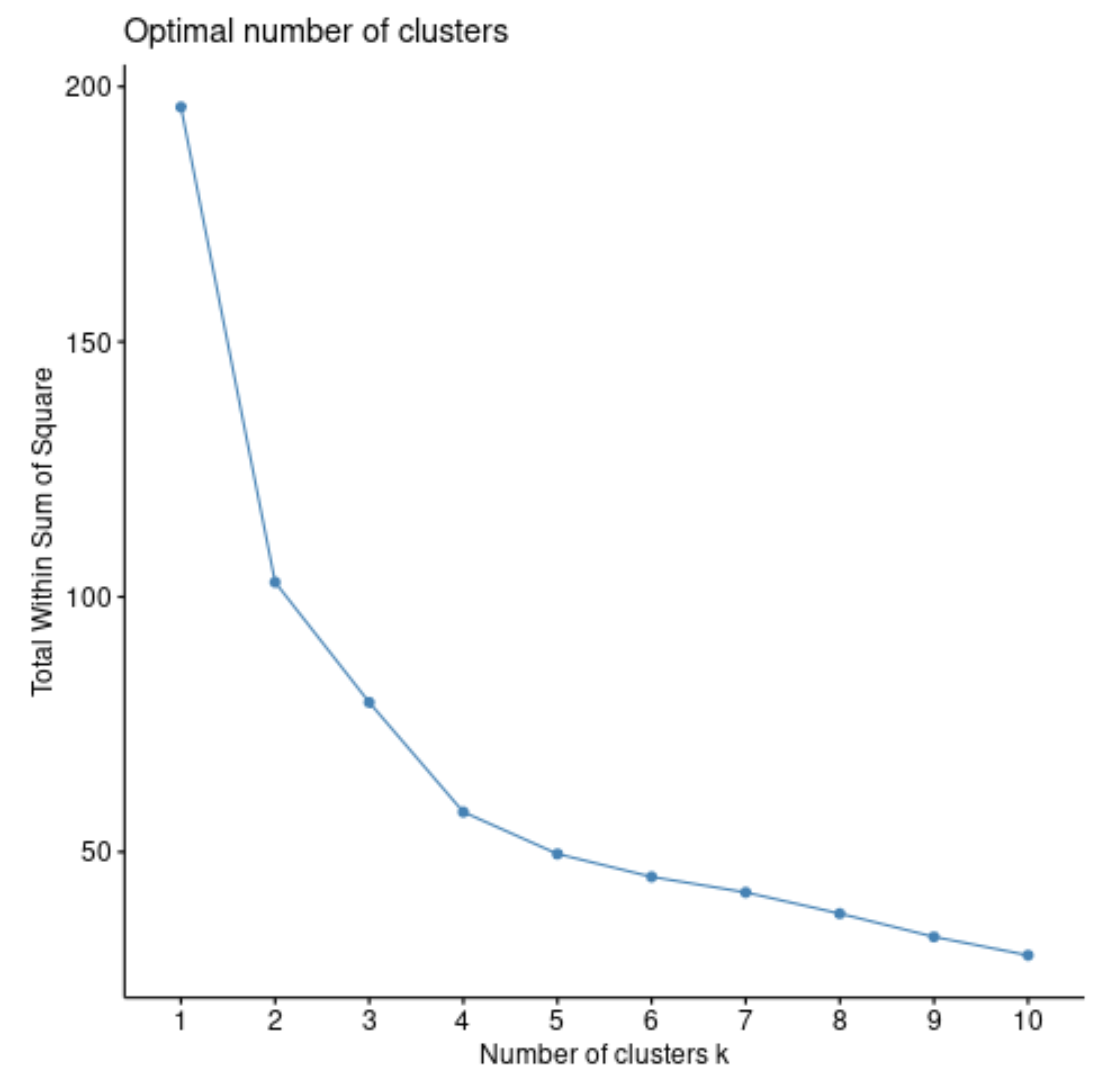
Het totaal in de som der kwadraten zal over het algemeen altijd toenemen naarmate we het aantal clusters vergroten. Dus als we dit type plot maken, zoeken we naar een ‘knie’ waar de som van de kwadraten begint te ‘buigen’ of af te vlakken.
Het krommingspunt van de grafiek komt doorgaans overeen met het optimale aantal clusters. Boven dit cijfer is het waarschijnlijk dat er sprake is van overfitting .
Voor deze grafiek lijkt het erop dat er een kleine knik of „buiging“ is bij k = 4 clusters.
2. Aantal clusters versus gap-statistieken
Een andere manier om het optimale aantal clusters te bepalen is door een metriek te gebruiken die de afwijkingsstatistiek wordt genoemd, die de totale intra-clustervariatie voor verschillende waarden van k vergelijkt met hun verwachte waarden voor een verdeling zonder clustering.
We kunnen de gap-statistiek voor elk aantal clusters berekenen met behulp van de clusGap()- functie uit het clusterpakket , evenals een grafiek van de clusters versus gap-statistieken met behulp van de fviz_gap_stat() functie:
#calculate gap statistic based on number of clusters gap_stat <- clusGap(df, FUN = pam, K.max = 10, #max clusters to consider B = 50) #total bootstrapped iterations #plot number of clusters vs. gap statistic fviz_gap_stat(gap_stat)
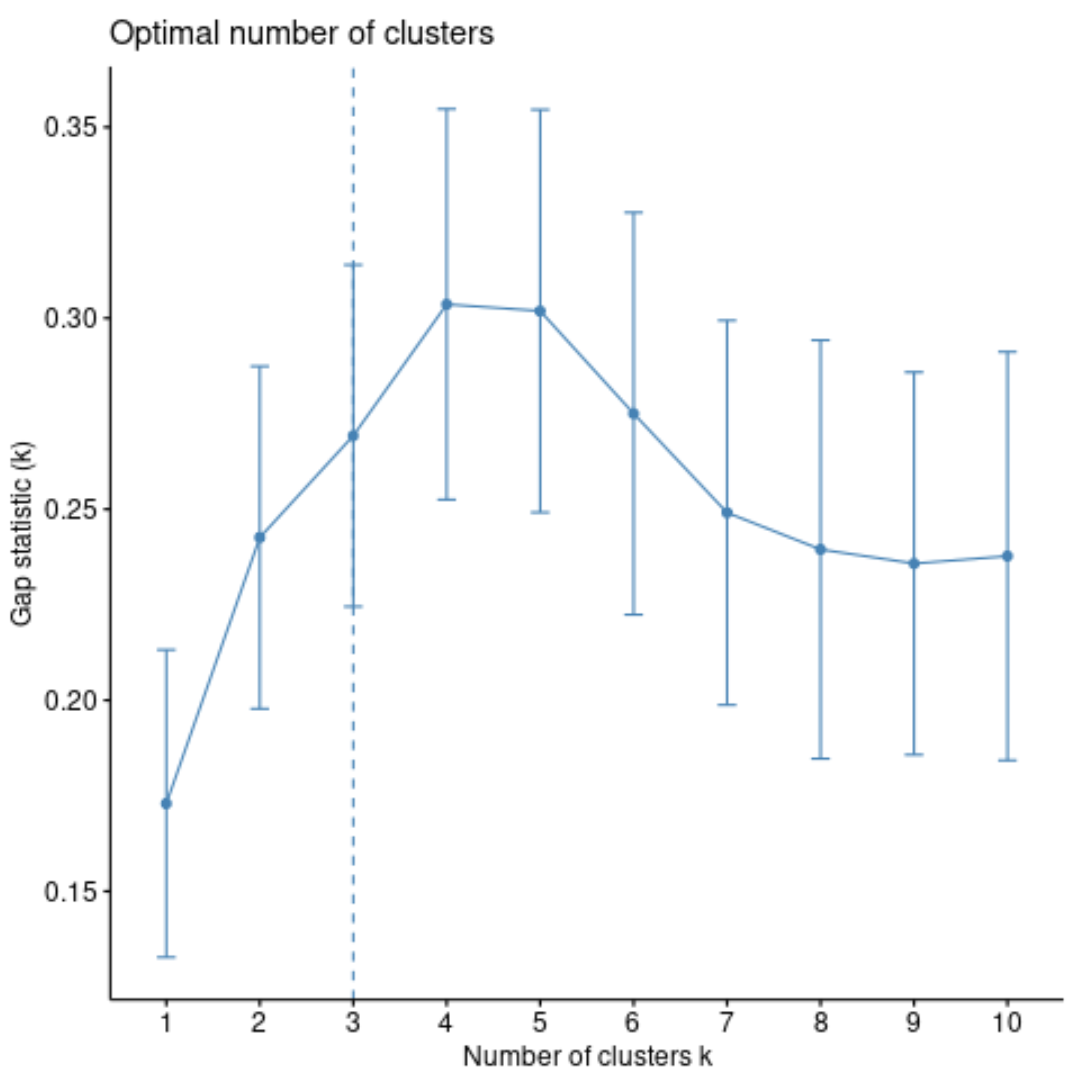
Uit de grafiek kunnen we zien dat de gap-statistiek het hoogst is bij k = 4 clusters, wat overeenkomt met de elleboogmethode die we eerder hebben gebruikt.
Stap 4: Voer K-Medoids-clustering uit met Optimal K
Ten slotte kunnen we k-medoids-clustering uitvoeren op de dataset met behulp van de optimale waarde voor k van 4:
#make this example reproducible set.seed(1) #perform k-medoids clustering with k = 4 clusters kmed <- pam(df, k = 4) #view results kmed ID Murder Assault UrbanPop Rape Alabama 1 1.2425641 0.7828393 -0.5209066 -0.003416473 Michigan 22 0.9900104 1.0108275 0.5844655 1.480613993 Oklahoma 36 -0.2727580 -0.2371077 0.1699510 -0.131534211 New Hampshire 29 -1.3059321 -1.3650491 -0.6590781 -1.252564419 Vector clustering: Alabama Alaska Arizona Arkansas California 1 2 2 1 2 Colorado Connecticut Delaware Florida Georgia 2 3 3 2 1 Hawaii Idaho Illinois Indiana Iowa 3 4 2 3 4 Kansas Kentucky Louisiana Maine Maryland 3 3 1 4 2 Massachusetts Michigan Minnesota Mississippi Missouri 3 2 4 1 3 Montana Nebraska Nevada New Hampshire New Jersey 3 3 2 4 3 New Mexico New York North Carolina North Dakota Ohio 2 2 1 4 3 Oklahoma Oregon Pennsylvania Rhode Island South Carolina 3 3 3 3 1 South Dakota Tennessee Texas Utah Vermont 4 1 2 3 4 Virginia Washington West Virginia Wisconsin Wyoming 3 3 4 4 3 Objective function: build swap 1.035116 1.027102 Available components: [1] "medoids" "id.med" "clustering" "objective" "isolation" [6] "clusinfo" "silinfo" "diss" "call" "data"
Merk op dat alle vier clusterzwaartepunten feitelijke waarnemingen in de dataset zijn. Bovenaan de uitvoer kunnen we zien dat de vier zwaartepunten de volgende toestanden hebben:
- Alabama
- Michigan
- Oklahoma
- New Hampshire
We kunnen de clusters visualiseren op een spreidingsdiagram dat de eerste twee hoofdcomponenten op de assen weergeeft met behulp van de fivz_cluster() functie:
#plot results of final k-medoids model
fviz_cluster(kmed, data = df)
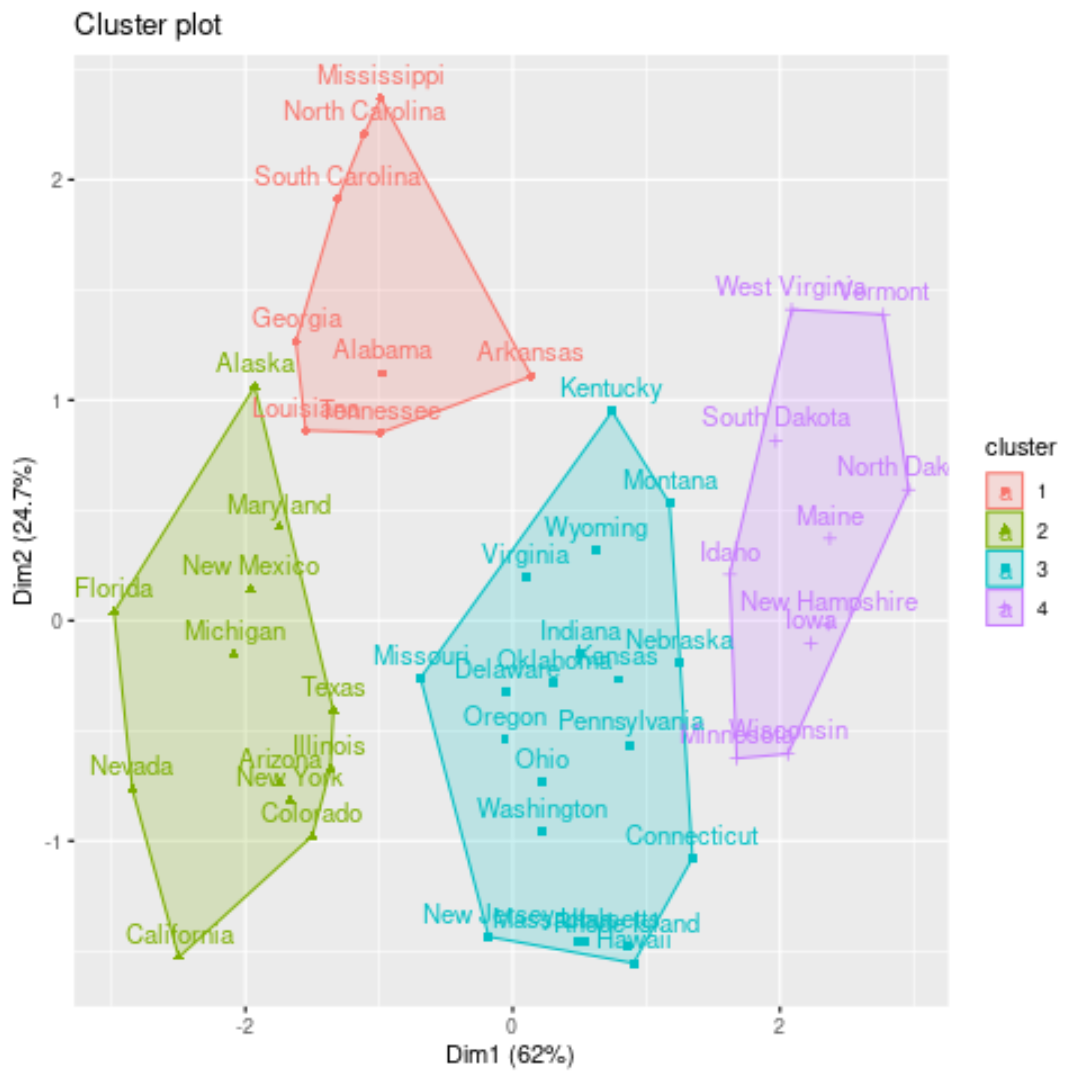
We kunnen ook de clustertoewijzingen van elke staat toevoegen aan de originele dataset:
#add cluster assignment to original data
final_data <- cbind(USArrests, cluster = kmed$cluster)
#view final data
head(final_data)
Murder Assault UrbanPop Rape cluster
Alabama 13.2 236 58 21.2 1
Alaska 10.0 263 48 44.5 2
Arizona 8.1 294 80 31.0 2
Arkansas 8.8 190 50 19.5 1
California 9.0 276 91 40.6 2
Colorado 7.9 204 78 38.7 2
De volledige R-code die in dit voorbeeld wordt gebruikt, vindt u hier .