Hoe multidimensionaal schalen uit te voeren in python
In de statistiek is multidimensionale schaling een manier om de gelijkenis van waarnemingen in een dataset in een abstracte cartesiaanse ruimte (meestal 2D-ruimte) te visualiseren.
De eenvoudigste manier om multidimensionaal schalen in Python uit te voeren, is door de functie MDS() van de submodule sklearn.manifold te gebruiken.
Het volgende voorbeeld laat zien hoe u deze functie in de praktijk kunt gebruiken.
Voorbeeld: multidimensionaal schalen in Python
Stel dat we het volgende panda’s DataFrame hebben dat informatie bevat over verschillende basketbalspelers:
import pandas as pd #create DataFrane df = pd. DataFrame ({' player ': ['A', 'B', 'C', 'D', 'E', 'F', 'G', 'H', 'I', 'J', 'K '], ' points ': [4, 4, 6, 7, 8, 14, 16, 19, 25, 25, 28], ' assists ': [3, 2, 2, 5, 4, 8, 7, 6, 8, 10, 11], ' blocks ': [7, 3, 6, 7, 5, 8, 8, 4, 2, 2, 1], ' rebounds ': [4, 5, 5, 6, 5, 8, 10, 4, 3, 2, 2]}) #set player column as index column df = df. set_index (' player ') #view Dataframe print (df) points assists blocks rebounds player A 4 3 7 4 B 4 2 3 5 C 6 2 6 5 D 7 5 7 6 E 8 4 5 5 F 14 8 8 8 G 16 7 8 10 H 19 6 4 4 I 25 8 2 3 D 25 10 2 2 K 28 11 1 2
We kunnen de volgende code gebruiken om multidimensionaal schalen uit te voeren met de MDS() functie van de sklearn.manifold module:
from sklearn. manifold import MDS
#perform multi-dimensional scaling
mds = MDS(random_state= 0 )
scaled_df = mds. fit_transform (df)
#view results of multi-dimensional scaling
print (scaled_df)
[[ 7.43654469 8.10247222]
[4.13193821 10.27360901]
[5.20534681 7.46919526]
[6.22323046 4.45148627]
[3.74110999 5.25591459]
[3.69073384 -2.88017811]
[3.89092087 -5.19100988]
[ -3.68593169 -3.0821144 ]
[ -9.13631889 -6.81016012]
[ -8.97898385 -8.50414387]
[-12.51859044 -9.08507097]]
Elke rij van het oorspronkelijke DataFrame is teruggebracht tot een (x, y)-coördinaat.
We kunnen de volgende code gebruiken om deze coördinaten in de 2D-ruimte te visualiseren:
import matplotlib.pyplot as plt #create scatterplot plt. scatter (scaled_df[:,0], scaled_df[:,1]) #add axis labels plt. xlabel (' Coordinate 1 ') plt. ylabel (' Coordinate 2 ') #add lables to each point for i, txt in enumerate( df.index ): plt. annotate (txt, (scaled_df[:,0][i]+.3, scaled_df[:,1][i])) #display scatterplot plt. show ()
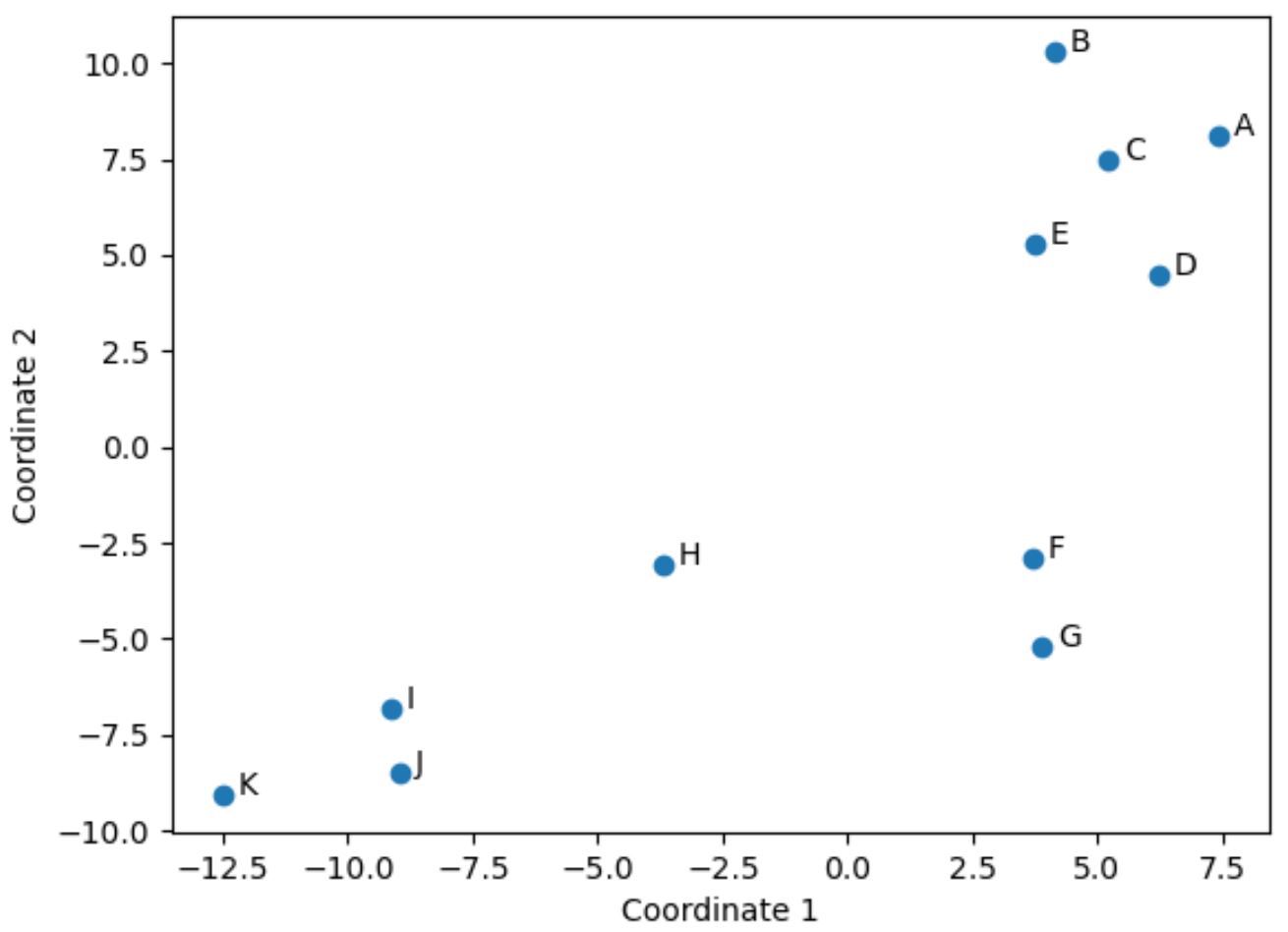
Spelers in het originele DataFrame die vergelijkbare waarden hebben in de oorspronkelijke vier kolommen (punten, assists, blocks en rebounds) staan in de plot dicht bij elkaar.
Spelers F en G zijn bijvoorbeeld gesloten voor elkaar. Hier zijn hun waarden uit het originele DataFrame:
#select rows with index labels 'F' and 'G'
df. loc [[' F ',' G ']]
points assists blocks rebounds
player
F 14 8 8 8
G 16 7 8 10
Hun waarden voor punten, assists, blocks en rebounds zijn allemaal behoorlijk vergelijkbaar, wat verklaart waarom ze zo dicht bij elkaar liggen in de 2D-plot.
Neem daarentegen spelers B en K die ver uit elkaar staan in het plot.
Als we naar hun waarden in het originele DataFrame verwijzen, kunnen we zien dat ze behoorlijk verschillend zijn:
#select rows with index labels 'B' and 'K'
df. loc [[' B ',' K ']]
points assists blocks rebounds
player
B 4 2 3 5
K 28 11 1 2
De 2D-plot is dus een goede manier om te visualiseren hoe vergelijkbaar elke speler is voor alle variabelen in het DataFframe.
Spelers met vergelijkbare statistieken worden dicht bij elkaar gegroepeerd, terwijl spelers met zeer verschillende statistieken verder van elkaar verwijderd zijn in het plot.
Aanvullende bronnen
In de volgende tutorials wordt uitgelegd hoe u andere veelvoorkomende taken in Python kunt uitvoeren:
Hoe gegevens in Python te normaliseren
Hoe uitschieters in Python te verwijderen
Hoe te testen op normaliteit in Python