Een restplot maken in ggplot2 (met voorbeeld)
Residuele plots worden gebruikt om te beoordelen of de residuen van een regressiemodel normaal verdeeld zijn en of ze al dan niet heteroscedasticiteit vertonen.
Om een restplot in ggplot2 te maken, kunt u de volgende basissyntaxis gebruiken:
library (ggplot2) ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 )
Het volgende voorbeeld laat zien hoe u deze syntaxis in de praktijk kunt gebruiken.
Voorbeeld: een restplot maken in ggplot2
Voor dit voorbeeld gebruiken we de mtcars- dataset die in R is ingebouwd:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
Eerst zullen we een regressiemodel passen met mpg als de responsvariabele en qsec als de voorspellende variabele:
#fit regression model
model <- lm(mpg ~ qsec, data=mtcars)
Vervolgens zullen we de volgende syntaxis gebruiken om een restplot in ggplot2 te maken:
library (ggplot2) #create residual plot ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 )
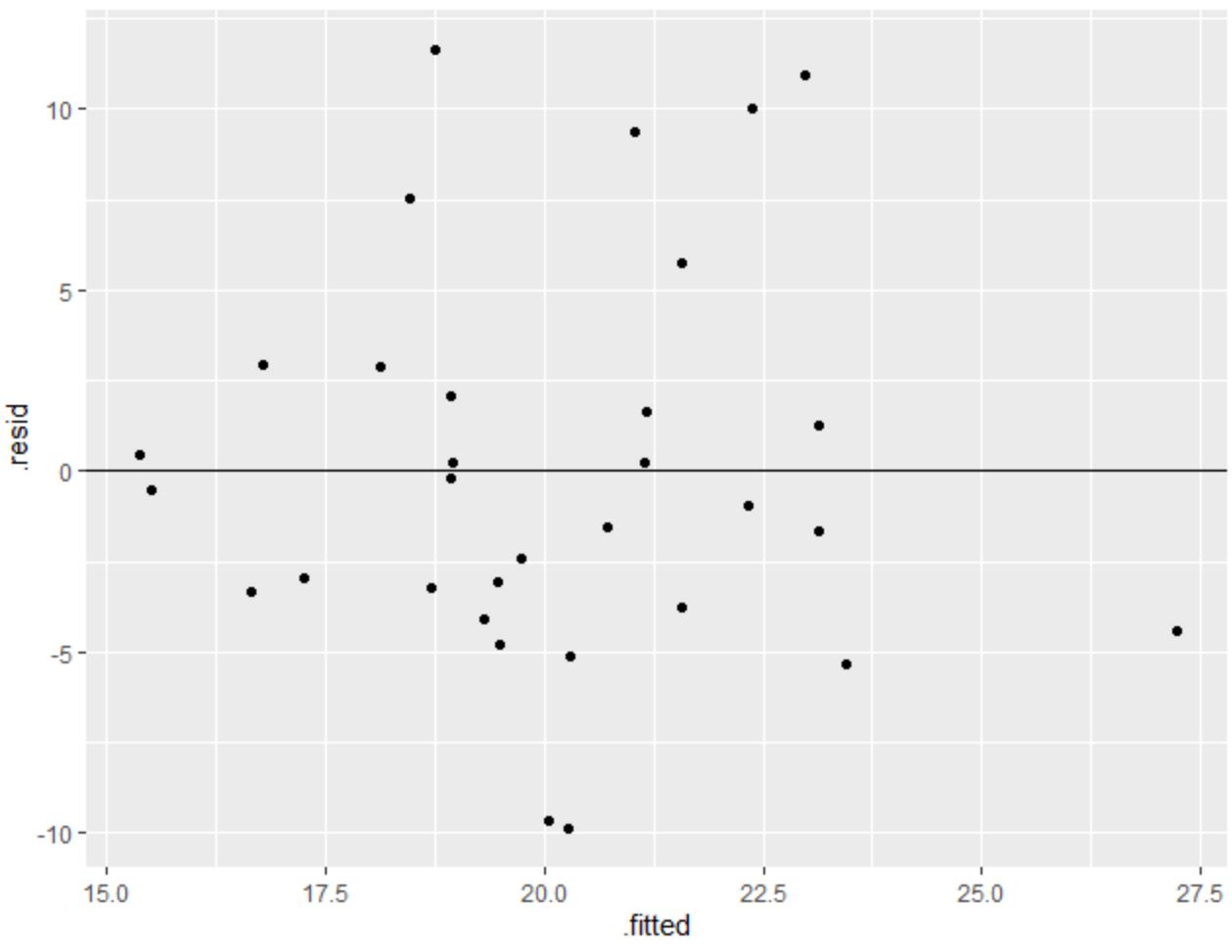
Op de x-as worden de gefitte waarden weergegeven en op de y-as de residuen.
De residuen lijken willekeurig verspreid rond nul te zijn zonder duidelijk patroon, wat aangeeft dat aan de aanname van homoscedasticiteit is voldaan.
Met andere woorden: de coëfficiënten van het regressiemodel moeten betrouwbaar zijn en we hoeven geen enkele transformatie op de gegevens uit te voeren.
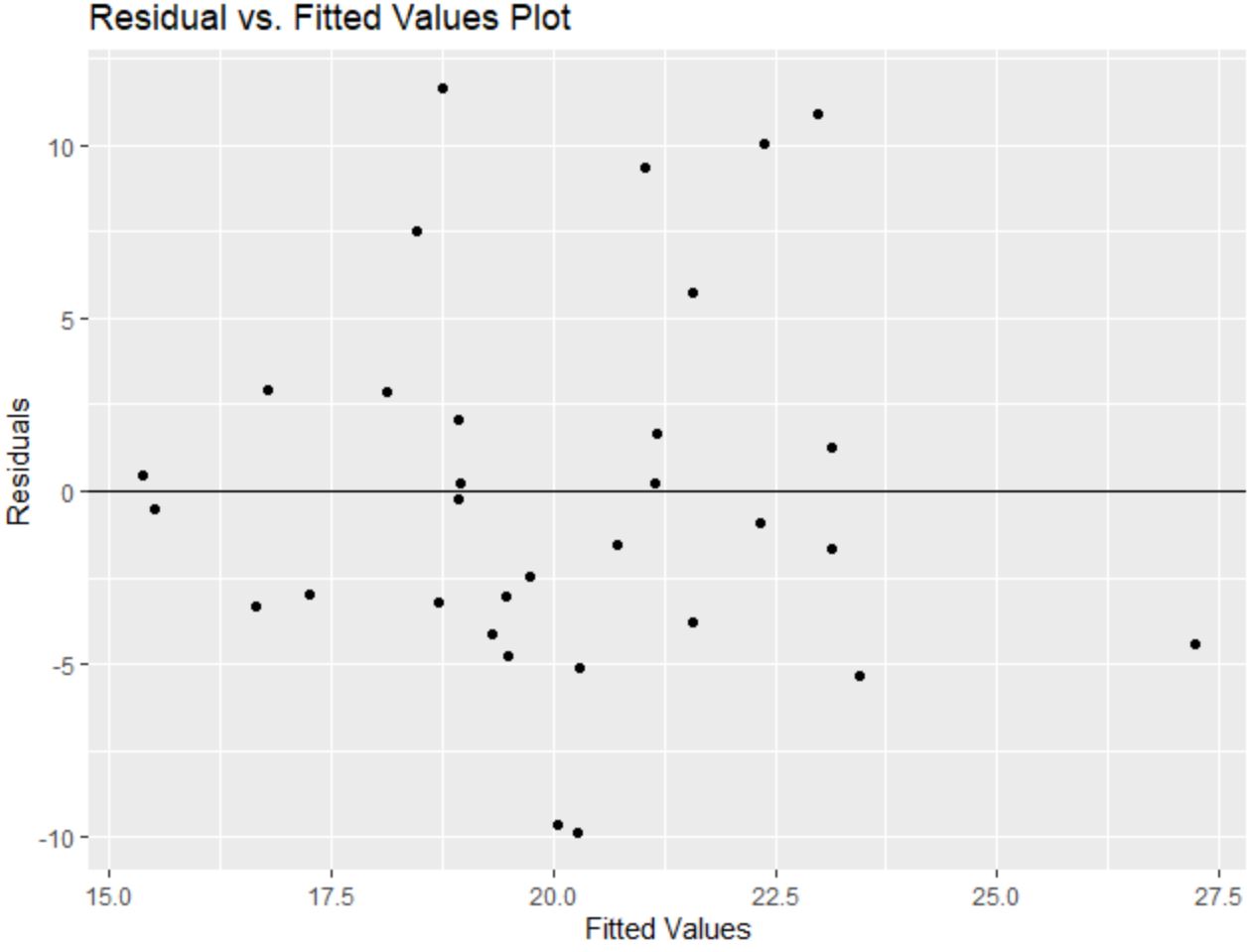
Merk ook op dat we de functie labs() kunnen gebruiken om een titel en aslabels aan de restplot toe te voegen:
library (ggplot2) #create residual plot with title and axis labels ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 ) + labs(title=' Residual vs. Fitted Values Plot ', x=' Fitted Values ', y=' Residuals ')
Aanvullende bronnen
In de volgende tutorials wordt uitgelegd hoe u andere veelvoorkomende taken in R kunt uitvoeren:
Hoe gestandaardiseerde residuen in R te berekenen
Hoe gestudentiseerde residuen in R te berekenen
Hoe maak je een histogram van residuen in R