Jak tworzyć losowe lasy w r (krok po kroku)
Kiedy związek między zbiorem zmiennych predykcyjnych a zmienną odpowiedzi jest bardzo złożony, często używamy metod nieliniowych do modelowania związku między nimi.
Jedną z takich metod jest zbudowanie drzewa decyzyjnego . Jednakże wadą stosowania pojedynczego drzewa decyzyjnego jest to, że charakteryzuje się ono dużą wariancją .
Oznacza to, że jeśli podzielimy zbiór danych na dwie połowy i zastosujemy drzewo decyzyjne do obu połówek, wyniki mogą być bardzo różne.
Jedną z metod, których możemy użyć do zmniejszenia wariancji pojedynczego drzewa decyzyjnego, jest zbudowanie losowego modelu lasu , który działa w następujący sposób:
1. Pobierz próbki bootstrapowe z oryginalnego zbioru danych.
2. Utwórz drzewo decyzyjne dla każdej próbki bootstrap.
- Podczas konstruowania drzewa za każdym razem, gdy rozważany jest podział, tylko losowa próbka m predyktorów jest uważana za kandydatów do podziału z pełnego zestawu p predyktorów. Generalnie wybieramy m równe √p .
3. Uśrednij przewidywania z każdego drzewa, aby otrzymać ostateczny model.
Okazuje się, że losowe lasy zwykle tworzą znacznie dokładniejsze modele niż pojedyncze drzewa decyzyjne, a nawet modele workowe .
W tym samouczku przedstawiono krok po kroku przykład tworzenia losowego modelu lasu dla zbioru danych w języku R.
Krok 1: Załaduj niezbędne pakiety
Najpierw załadujemy pakiety niezbędne dla tego przykładu. W tym prostym przykładzie potrzebujemy tylko jednego pakietu:
library (randomForest)
Krok 2: Dostosuj model losowego lasu
W tym przykładzie użyjemy wbudowanego zbioru danych R o nazwie Air Quality , który zawiera pomiary jakości powietrza w Nowym Jorku w ciągu 153 poszczególnych dni.
#view structure of air quality dataset str(airquality) 'data.frame': 153 obs. of 6 variables: $ Ozone: int 41 36 12 18 NA 28 23 19 8 NA ... $Solar.R: int 190 118 149 313 NA NA 299 99 19 194 ... $ Wind: num 7.4 8 12.6 11.5 14.3 14.9 8.6 13.8 20.1 8.6 ... $ Temp: int 67 72 74 62 56 66 65 59 61 69 ... $Month: int 5 5 5 5 5 5 5 5 5 5 ... $Day: int 1 2 3 4 5 6 7 8 9 10 ... #find number of rows with missing values sum(! complete . cases (airquality)) [1] 42
Ten zbiór danych zawiera 42 wiersze z brakującymi wartościami. Dlatego przed dopasowaniem losowego modelu lasu uzupełnimy brakujące wartości w każdej kolumnie medianami kolumn:
#replace NAs with column medians for (i in 1: ncol (air quality)) { airquality[,i][ is . na (airquality[, i])] <- median (airquality[, i], na . rm = TRUE ) }
Powiązane: Jak przypisać brakujące wartości w R
Poniższy kod pokazuje, jak dopasować losowy model lasu w R przy użyciu funkcji randomForest() z pakietu randomForest .
#make this example reproducible set.seed(1) #fit the random forest model model <- randomForest( formula = Ozone ~ ., data = airquality ) #display fitted model model Call: randomForest(formula = Ozone ~ ., data = airquality) Type of random forest: regression Number of trees: 500 No. of variables tried at each split: 1 Mean of squared residuals: 327.0914 % Var explained: 61 #find number of trees that produce lowest test MSE which.min(model$mse) [1] 82 #find RMSE of best model sqrt(model$mse[ which . min (model$mse)]) [1] 17.64392
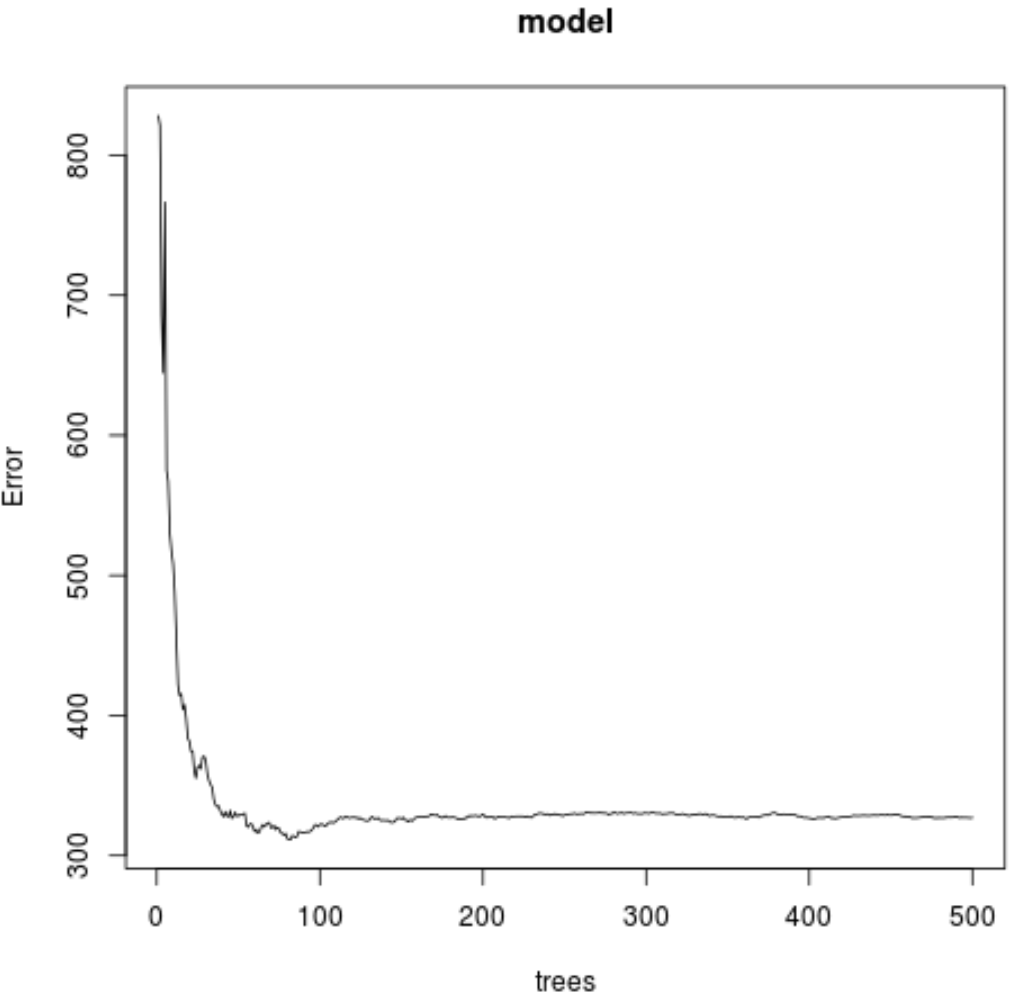
Z wyniku widać, że model, który wygenerował najniższy testowy błąd średniokwadratowy (MSE), wykorzystywał 82 drzewa.
Widzimy również, że średni błąd kwadratowy tego modelu wyniósł 17,64392 . Możemy to traktować jako średnią różnicę między przewidywaną wartością ozonu a rzeczywistą obserwowaną wartością.
Możemy również użyć poniższego kodu, aby utworzyć wykres testu MSE w oparciu o liczbę użytych drzew:
#plot the MSE test by number of trees
plot(model)
Możemy także użyć funkcji varImpPlot() , aby utworzyć wykres przedstawiający ważność każdej zmiennej predykcyjnej w ostatecznym modelu:
#produce variable importance plot
varImpPlot(model)
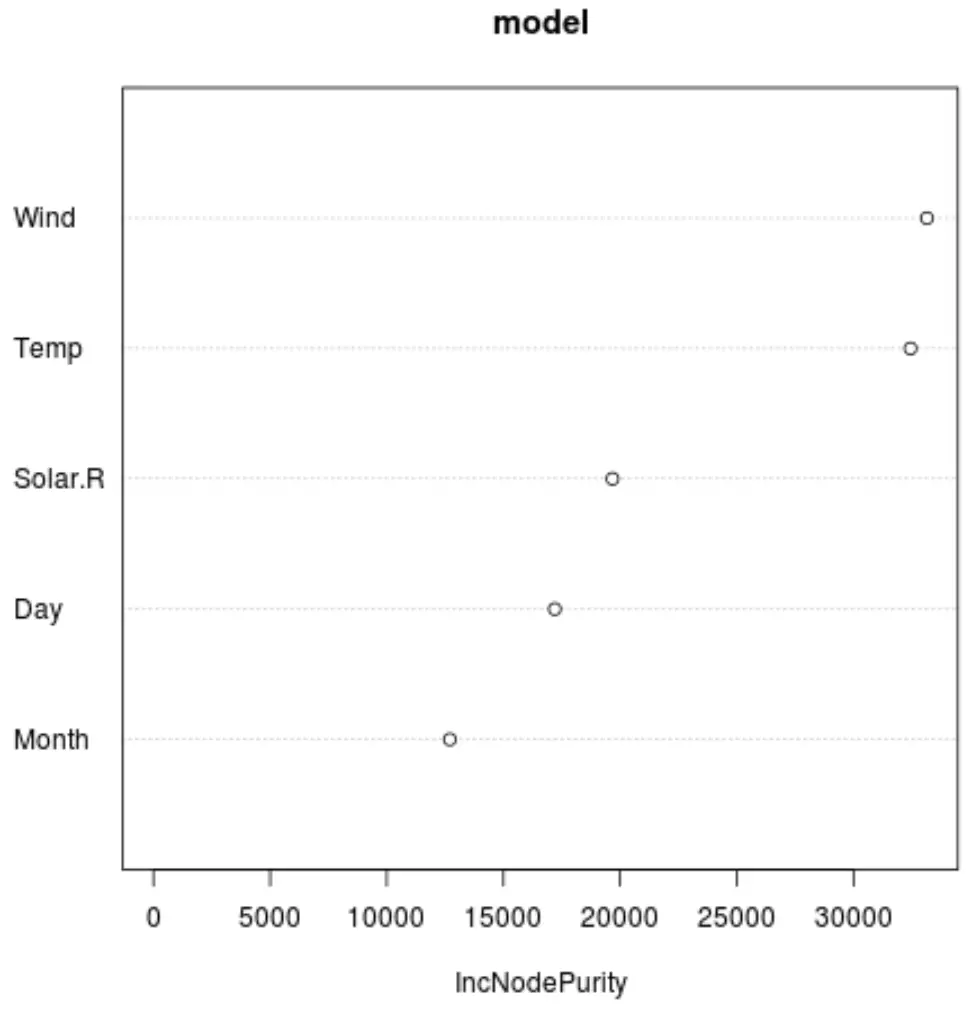
Oś x przedstawia średni wzrost czystości węzłów drzew regresji jako funkcję podziału na różne predyktory wyświetlane na osi y.
Z wykresu widzimy, że najważniejszą zmienną predykcyjną jest Wiatr , a tuż za nim znajduje się Temp .
Krok 3: Dopasuj model
Domyślnie funkcja randomForest() wykorzystuje 500 drzew i (łącznie predyktorów/3) losowo wybranych predyktorów jako potencjalnych kandydatów do każdego podziału. Możemy dostosować te parametry za pomocą funkcji tuneRF() .
Poniższy kod pokazuje, jak znaleźć optymalny model przy użyciu następujących specyfikacji:
- ntreeTry: Liczba drzew do zbudowania.
- mtryStart: początkowa liczba zmiennych predykcyjnych, które należy uwzględnić przy każdym podziale.
- stepFactor: Współczynnik zwiększany do momentu, aż szacowany błąd out-of-bag przestanie się poprawiać o określoną wartość.
- poprawić: wielkość, o jaką należy poprawić błąd wyjścia worka, aby w dalszym ciągu zwiększać współczynnik kroku.
model_tuned <- tuneRF(
x=airquality[,-1], #define predictor variables
y=airquality$Ozone, #define response variable
ntreeTry= 500 ,
mtryStart= 4 ,
stepFactor= 1.5 ,
improve= 0.01 ,
trace= FALSE #don't show real-time progress
)
Ta funkcja generuje następujący wykres, który przedstawia liczbę predyktorów użytych przy każdym podziale podczas konstruowania drzew na osi x i szacowany błąd out-of-bag na osi y:
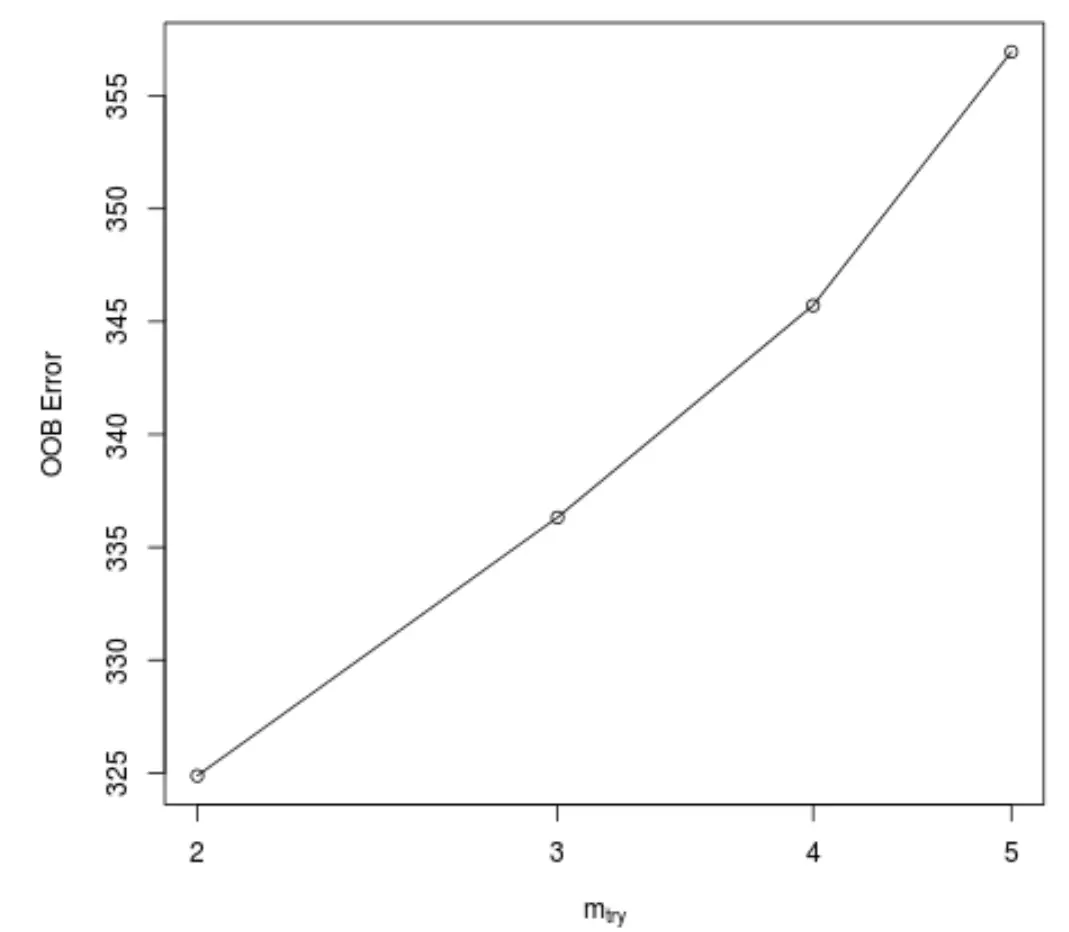
Widzimy, że najniższy błąd OOB uzyskuje się, stosując 2 losowo wybrane predyktory przy każdym podziale podczas budowania drzew.
W rzeczywistości odpowiada to ustawieniu domyślnemu (liczba predyktorów/3 = 6/3 = 2) używanemu przez początkową funkcję randomForest() .
Krok 4: Użyj ostatecznego modelu do przewidywania
Wreszcie możemy użyć skorygowanego modelu lasu losowego do przewidywania nowych obserwacji.
#define new observation new <- data.frame(Solar.R=150, Wind=8, Temp=70, Month=5, Day=5) #use fitted bagged model to predict Ozone value of new observation predict(model, newdata=new) 27.19442
Na podstawie wartości zmiennych predykcyjnych dopasowany losowy model lasu przewiduje, że w tym konkretnym dniu wartość ozonu wyniesie 27,19442 .
Pełny kod R użyty w tym przykładzie można znaleźć tutaj .