K-medoids w r: przykład krok po kroku
Grupowanie to technika uczenia maszynowego, która próbuje znaleźć grupy lub skupienia obserwacji w zbiorze danych.
Celem jest znalezienie takich skupień, w których obserwacje w każdym klastrze będą do siebie dość podobne, podczas gdy obserwacje w różnych skupieniach znacznie się od siebie różnią.
Klastrowanie jest formą uczenia się bez nadzoru , ponieważ po prostu staramy się znaleźć strukturę w zbiorze danych, zamiast przewidywać wartość zmiennej odpowiedzi .
Klastrowanie jest często stosowane w marketingu, gdy firmy mają dostęp do informacji takich jak:
- Przychód domowy
- Wielkość gospodarstwa domowego
- Zawód głowy gospodarstwa domowego
- Odległość do najbliższego obszaru miejskiego
Jeżeli takie informacje są dostępne, można zastosować grupowanie w celu zidentyfikowania gospodarstw domowych, które są podobne i które mogą z większym prawdopodobieństwem zakupić określone produkty lub lepiej reagować na określony rodzaj reklamy.
Jedną z najpowszechniejszych form grupowania jest grupowanie k-średnich .
Niestety na tę metodę mogą wpływać wartości odstające, dlatego często stosowaną alternatywą jest grupowanie k-medoidów .
Co to jest klastrowanie K-Medoids?
Grupowanie K-medoidów to technika, w której każdą obserwację ze zbioru danych umieszczamy w jednym z K klastrów.
Ostatecznym celem jest utworzenie K klastrów, w których obserwacje w każdym klastrze są do siebie dość podobne, podczas gdy obserwacje w różnych klastrach znacznie się od siebie różnią.
W praktyce do przeprowadzenia grupowania K-średnich stosujemy następujące kroki:
1. Wybierz wartość K.
- Najpierw musimy zdecydować, ile skupień chcemy zidentyfikować w danych. Często wystarczy po prostu przetestować kilka różnych wartości K i przeanalizować wyniki, aby zobaczyć, która liczba skupień wydaje się mieć największy sens dla danego problemu.
2. Losowo przypisz każdą obserwację do skupienia początkowego, od 1 do K.
3. Wykonuj poniższą procedurę, aż przypisania klastrów przestaną się zmieniać.
- Dla każdej z gromad K oblicz środek ciężkości gromady. Jest to wektor p median cech dla obserwacji k- tego skupienia.
- Przypisz każdą obserwację do klastra o najbliższym centroidzie. Tutaj najbliżej definiuje się odległość euklidesową .
Uwaga techniczna:
Ponieważ k-medoids oblicza centroidy klastrów przy użyciu median, a nie średnich, jest on zwykle bardziej odporny na wartości odstające niż k-średnie.
W praktyce, jeśli w zbiorze danych nie ma skrajnych wartości odstających, k-średnie i k-medoidy dadzą podobne wyniki.
Klaster K-Medoids w R
Poniższy samouczek zawiera przykład krok po kroku wykonywania grupowania k-medoids w języku R.
Krok 1: Załaduj niezbędne pakiety
Najpierw załadujemy dwa pakiety zawierające kilka przydatnych funkcji do grupowania k-medoids w R.
library (factoextra) library (cluster)
Krok 2: Załaduj i przygotuj dane
W tym przykładzie użyjemy zbioru danych USArrests wbudowanego w R, który zawiera liczbę aresztowań na 100 000 mieszkańców w każdym stanie USA w 1973 r. za morderstwo , napaść i gwałt , a także procent populacji każdego stanu mieszkającej w obszarach miejskich obszary. , UrbanPop .
Poniższy kod pokazuje, jak wykonać następujące czynności:
- Załaduj zbiór danych USArrests
- Usuń wszystkie wiersze z brakującymi wartościami
- Skaluj każdą zmienną w zbiorze danych tak, aby miała średnią 0 i odchylenie standardowe 1
#load data df <-USArrests #remove rows with missing values df <- na. omitted (df) #scale each variable to have a mean of 0 and sd of 1 df <- scale(df) #view first six rows of dataset head(df) Murder Assault UrbanPop Rape Alabama 1.24256408 0.7828393 -0.5209066 -0.003416473 Alaska 0.50786248 1.1068225 -1.2117642 2.484202941 Arizona 0.07163341 1.4788032 0.9989801 1.042878388 Arkansas 0.23234938 0.2308680 -1.0735927 -0.184916602 California 0.27826823 1.2628144 1.7589234 2.067820292 Colorado 0.02571456 0.3988593 0.8608085 1.864967207
Krok 3: Znajdź optymalną liczbę klastrów
Aby wykonać grupowanie k-medoid w R, możemy użyć funkcji pam() , która oznacza „partycjonowanie wokół median” i wykorzystuje następującą składnię:
pam(dane, k, metryka = „euklidesowy”, stojak = FAŁSZ)
Złoto:
- dane: nazwa zbioru danych.
- k: liczba klastrów.
- metryka: metryka używana do obliczania odległości. Wartość domyślna to Euclidean , ale możesz także określić Manhattan .
- stand: Określa, czy normalizować każdą zmienną w zbiorze danych. Wartość domyślna to fałsz.
Ponieważ nie wiemy z góry, jaka liczba skupień jest optymalna, utworzymy dwa różne wykresy, które pomogą nam podjąć decyzję:
1. Liczba skupień w stosunku do sumy kwadratów
Najpierw użyjemy funkcji fviz_nbclust() , aby utworzyć wykres liczby skupień w funkcji sumy kwadratów:
fviz_nbclust(df, pam, method = “ wss ”)
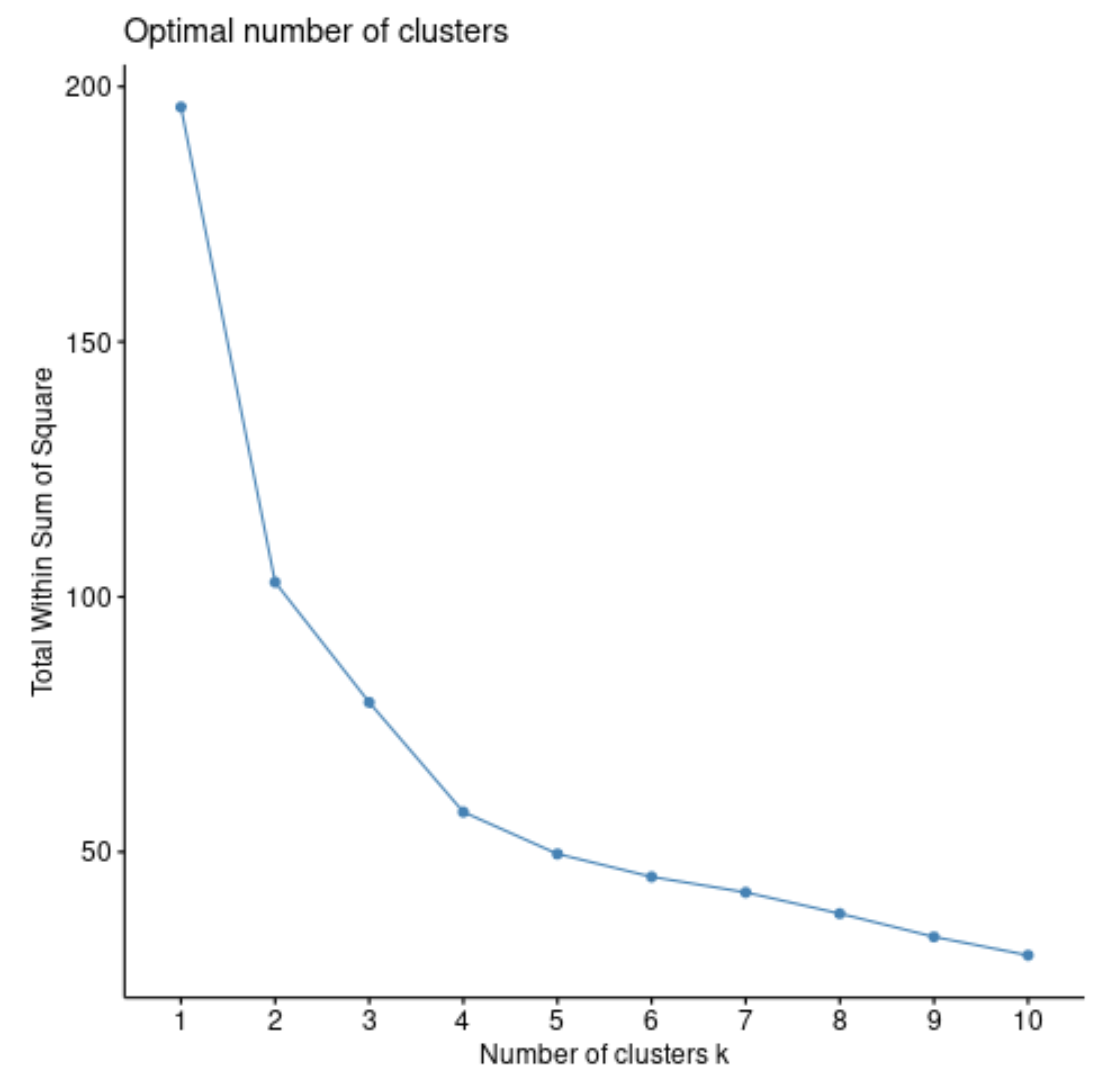
Suma kwadratów na ogół zawsze będzie rosła wraz ze zwiększaniem liczby klastrów. Kiedy więc tworzymy tego typu wykres, szukamy „kolana”, w którym suma kwadratów zaczyna się „zaginać” lub wyrównywać.
Punkt krzywizny wykresu generalnie odpowiada optymalnej liczbie skupień. Powyżej tej wartości prawdopodobne jest wystąpienie nadmiernego dopasowania .
W przypadku tego wykresu wydaje się, że przy k = 4 skupiskach występuje niewielkie załamanie lub „zagięcie”.
2. Liczba klastrów a statystyki luk
Innym sposobem określenia optymalnej liczby skupień jest użycie metryki zwanej statystyką odchyleń , która porównuje całkowitą zmienność wewnątrz skupień dla różnych wartości k z ich wartościami oczekiwanymi dla rozkładu bez grupowania.
Możemy obliczyć statystykę luk dla każdej liczby klastrów za pomocą funkcji clusGap() z pakietu klastrów , a także wykres klastrów w funkcji statystyki luk za pomocą funkcji fviz_gap_stat() :
#calculate gap statistic based on number of clusters gap_stat <- clusGap(df, FUN = pam, K.max = 10, #max clusters to consider B = 50) #total bootstrapped iterations #plot number of clusters vs. gap statistic fviz_gap_stat(gap_stat)
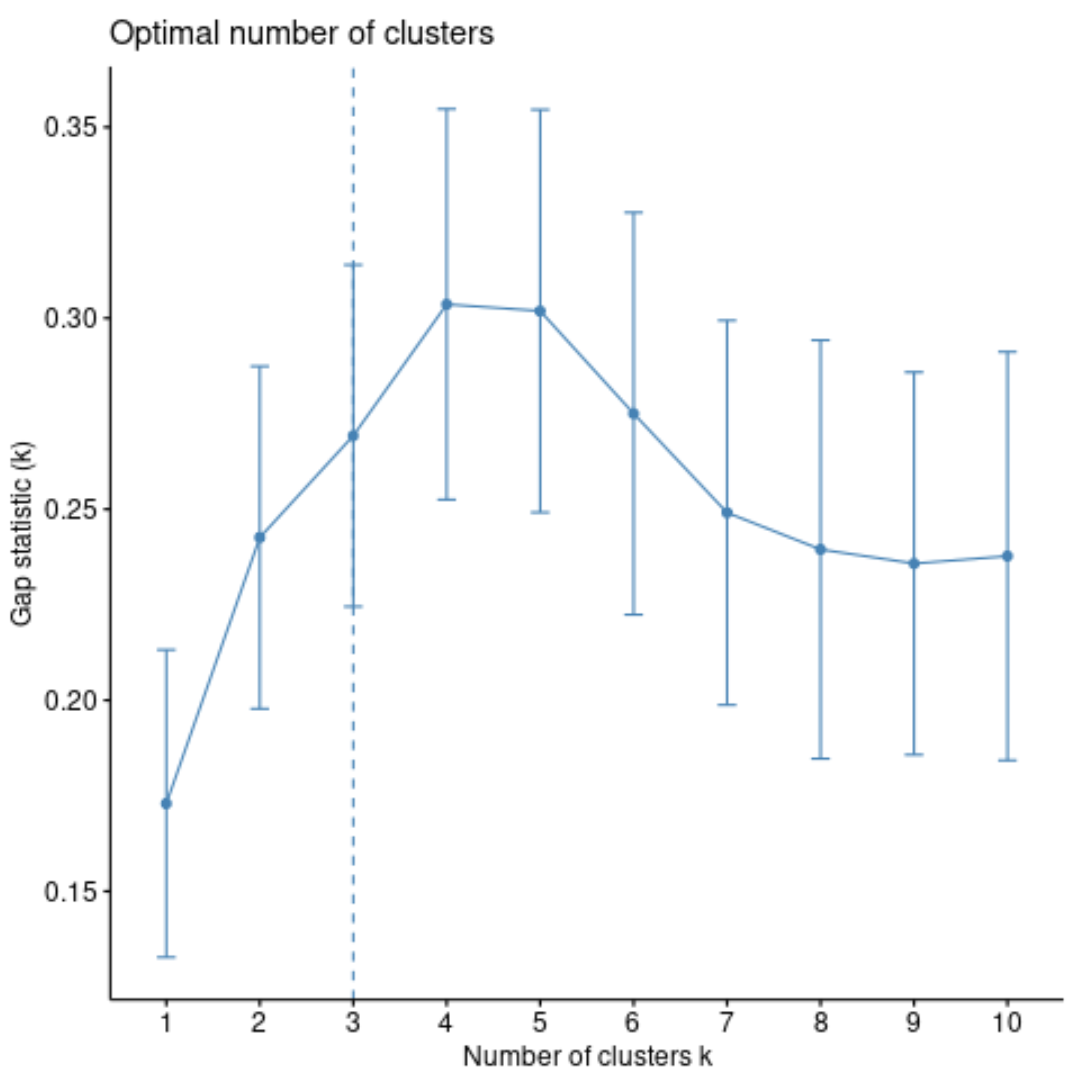
Z wykresu widać, że statystyka luki jest najwyższa przy k = 4 skupień, co odpowiada zastosowanej wcześniej metodzie łokcia.
Krok 4: Wykonaj grupowanie K-Medoids za pomocą Optimal K
Na koniec możemy wykonać grupowanie k-medoidów na zbiorze danych, używając optymalnej wartości k z 4:
#make this example reproducible set.seed(1) #perform k-medoids clustering with k = 4 clusters kmed <- pam(df, k = 4) #view results kmed ID Murder Assault UrbanPop Rape Alabama 1 1.2425641 0.7828393 -0.5209066 -0.003416473 Michigan 22 0.9900104 1.0108275 0.5844655 1.480613993 Oklahoma 36 -0.2727580 -0.2371077 0.1699510 -0.131534211 New Hampshire 29 -1.3059321 -1.3650491 -0.6590781 -1.252564419 Vector clustering: Alabama Alaska Arizona Arkansas California 1 2 2 1 2 Colorado Connecticut Delaware Florida Georgia 2 3 3 2 1 Hawaii Idaho Illinois Indiana Iowa 3 4 2 3 4 Kansas Kentucky Louisiana Maine Maryland 3 3 1 4 2 Massachusetts Michigan Minnesota Mississippi Missouri 3 2 4 1 3 Montana Nebraska Nevada New Hampshire New Jersey 3 3 2 4 3 New Mexico New York North Carolina North Dakota Ohio 2 2 1 4 3 Oklahoma Oregon Pennsylvania Rhode Island South Carolina 3 3 3 3 1 South Dakota Tennessee Texas Utah Vermont 4 1 2 3 4 Virginia Washington West Virginia Wisconsin Wyoming 3 3 4 4 3 Objective function: build swap 1.035116 1.027102 Available components: [1] "medoids" "id.med" "clustering" "objective" "isolation" [6] "clusinfo" "silinfo" "diss" "call" "data"
Należy zauważyć, że wszystkie cztery centroidy klastrów są rzeczywistymi obserwacjami w zbiorze danych. W górnej części wyniku widzimy, że cztery centroidy znajdują się w następujących stanach:
- Alabama
- Michigan
- Oklahoma
- New Hampshire
Możemy wizualizować skupienia na wykresie punktowym, który wyświetla pierwsze dwie główne składowe na osiach, używając funkcji fivz_cluster() :
#plot results of final k-medoids model
fviz_cluster(kmed, data = df)
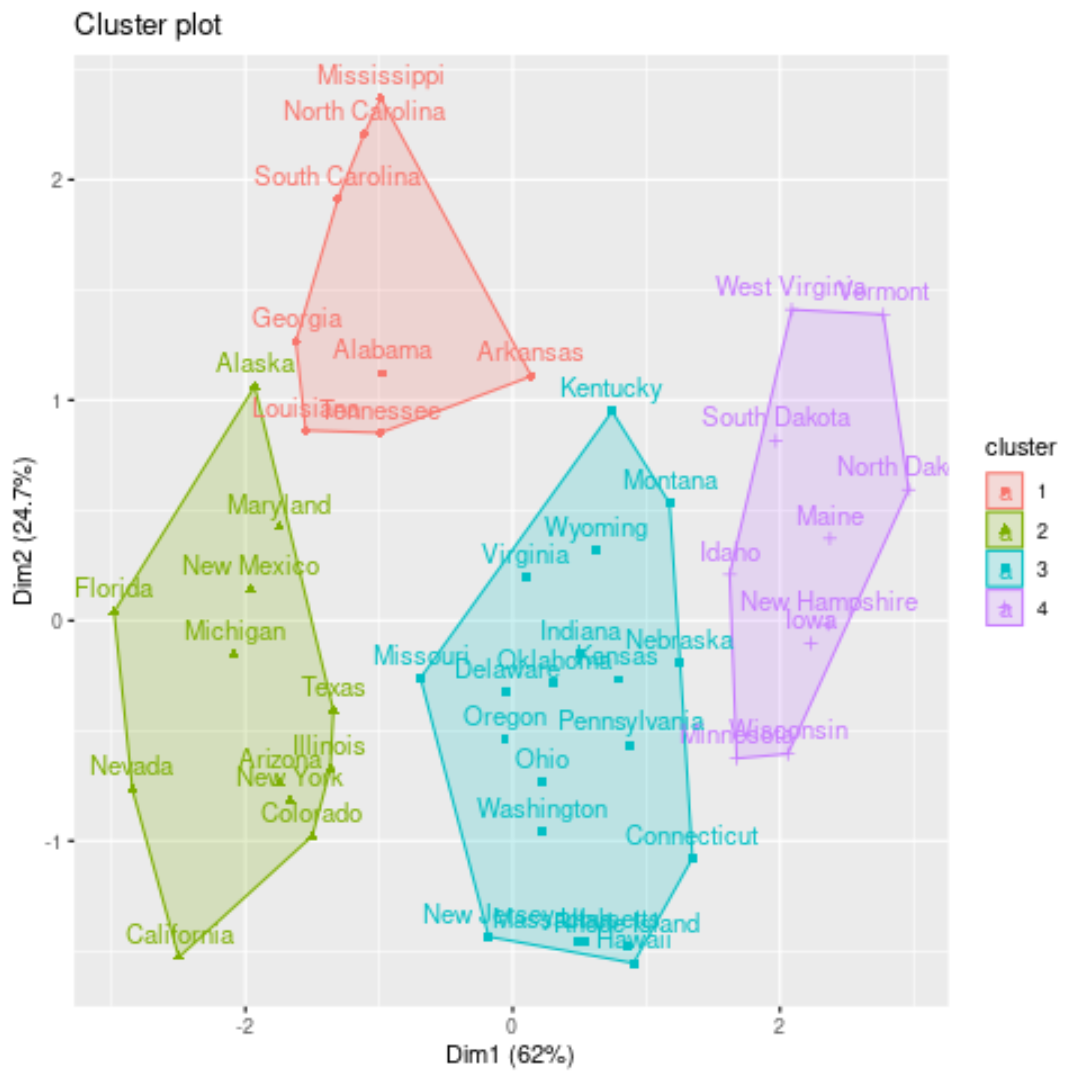
Możemy również dodać przypisania klastrów każdego stanu do oryginalnego zbioru danych:
#add cluster assignment to original data
final_data <- cbind(USArrests, cluster = kmed$cluster)
#view final data
head(final_data)
Murder Assault UrbanPop Rape cluster
Alabama 13.2 236 58 21.2 1
Alaska 10.0 263 48 44.5 2
Arizona 8.1 294 80 31.0 2
Arkansas 8.8 190 50 19.5 1
California 9.0 276 91 40.6 2
Colorado 7.9 204 78 38.7 2
Pełny kod R użyty w tym przykładzie znajdziesz tutaj .