Jak uruchomić bootstrap w r (z przykładami)
Metoda ładowania początkowego to metoda, której można użyć do oszacowania błędu standardowego dowolnej statystyki i ustalenia przedziału ufności dla tej statystyki.
Podstawowy proces ładowania początkowego jest następujący:
- Pobierz k powtórzonych próbek z zamianą z danego zbioru danych.
- Dla każdej próbki oblicz interesującą statystykę.
- Daje to k różnych szacunków dla danej statystyki, które można następnie wykorzystać do obliczenia błędu standardowego statystyki i utworzenia przedziału ufności dla tej statystyki.
Możemy wykonać bootstrap w R, korzystając z następujących funkcji z biblioteki bootstrap :
1. Wygeneruj próbki bootstrap.
boot(dane, statystyki, R, …)
Złoto:
- dane: wektor, macierz lub blok danych
- statystyka: funkcja generująca statystykę, która ma zostać zainicjowana
- Odp.: Liczba powtórzeń bootstrapu
2. Wygeneruj przedział ufności ładowania początkowego.
boot.ci (obiekt startowy, konf, typ)
Złoto:
- bootobject: Obiekt zwracany przez funkcję boot().
- conf: Przedział ufności do obliczenia. Wartość domyślna to 0,95
- type: Typ przedziału ufności do obliczenia. Opcje obejmują „standard”, „podstawowy”, „stud”, „perc”, „bca” i „all” – domyślnie jest to „all”
Poniższe przykłady pokazują, jak wykorzystać te funkcje w praktyce.
Przykład 1: bootstrap pojedynczej statystyki
Poniższy kod pokazuje, jak obliczyć błąd standardowy dla kwadratu R prostego modelu regresji liniowej:
set.seed(0) library (boot) #define function to calculate R-squared rsq_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (summary(fit)$r.square) #return R-squared of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=rsq_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = rsq_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 0.7183433 0.002164339 0.06513426
Z wyników możemy zobaczyć:
- Szacowany współczynnik R-kwadrat dla tego modelu regresji wynosi 0,7183433 .
- Błąd standardowy tego oszacowania wynosi 0,06513426 .
Możemy również szybko wizualizować rozkład próbek ładowanych metodą ładowania początkowego:
plot(reps)
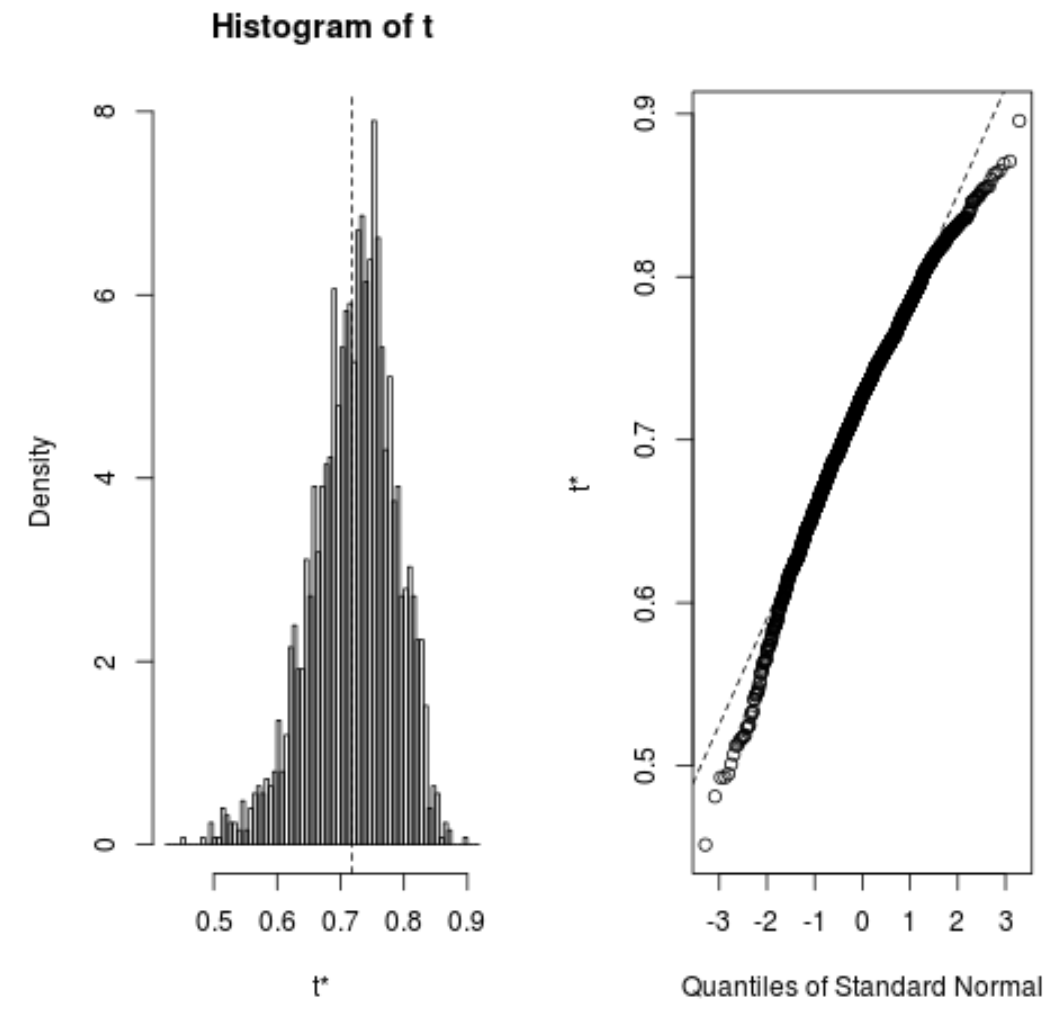
Możemy również użyć poniższego kodu, aby obliczyć 95% przedział ufności dla szacowanego kwadratu R modelu:
#calculate adjusted bootstrap percentile (BCa) interval boot.ci(reps, type=" bca ") CALL: boot.ci(boot.out = reps, type = "bca") Intervals: Level BCa 95% (0.5350, 0.8188) Calculations and Intervals on Original Scale
Z wyniku widzimy, że bootstrapowy 95% przedział ufności dla prawdziwych wartości R-kwadrat wynosi (0,5350, 0,8188).
Przykład 2: ładowanie wielu statystyk
Poniższy kod pokazuje, jak obliczyć błąd standardowy dla każdego współczynnika w modelu wielokrotnej regresji liniowej:
set.seed(0) library (boot) #define function to calculate fitted regression coefficients coef_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (coef(fit)) #return coefficient estimates of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=coef_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = coef_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 29.59985476 -5.058601e-02 1.49354577 t2* -0.04121512 6.549384e-05 0.00527082
Z wyników możemy zobaczyć:
- Oszacowany współczynnik wyrazu wolnego modelu wynosi 29,59985476 , a błąd standardowy tego oszacowania wynosi 1,49354577 .
- Oszacowany współczynnik dla zmiennej predykcyjnej disp w modelu wynosi -0,04121512 , a błąd standardowy tego oszacowania wynosi 0,00527082 .
Możemy również szybko wizualizować rozkład próbek ładowanych metodą bootstrap:
plot(reps, index=1) #intercept of model plot(reps, index=2) #disp predictor variable
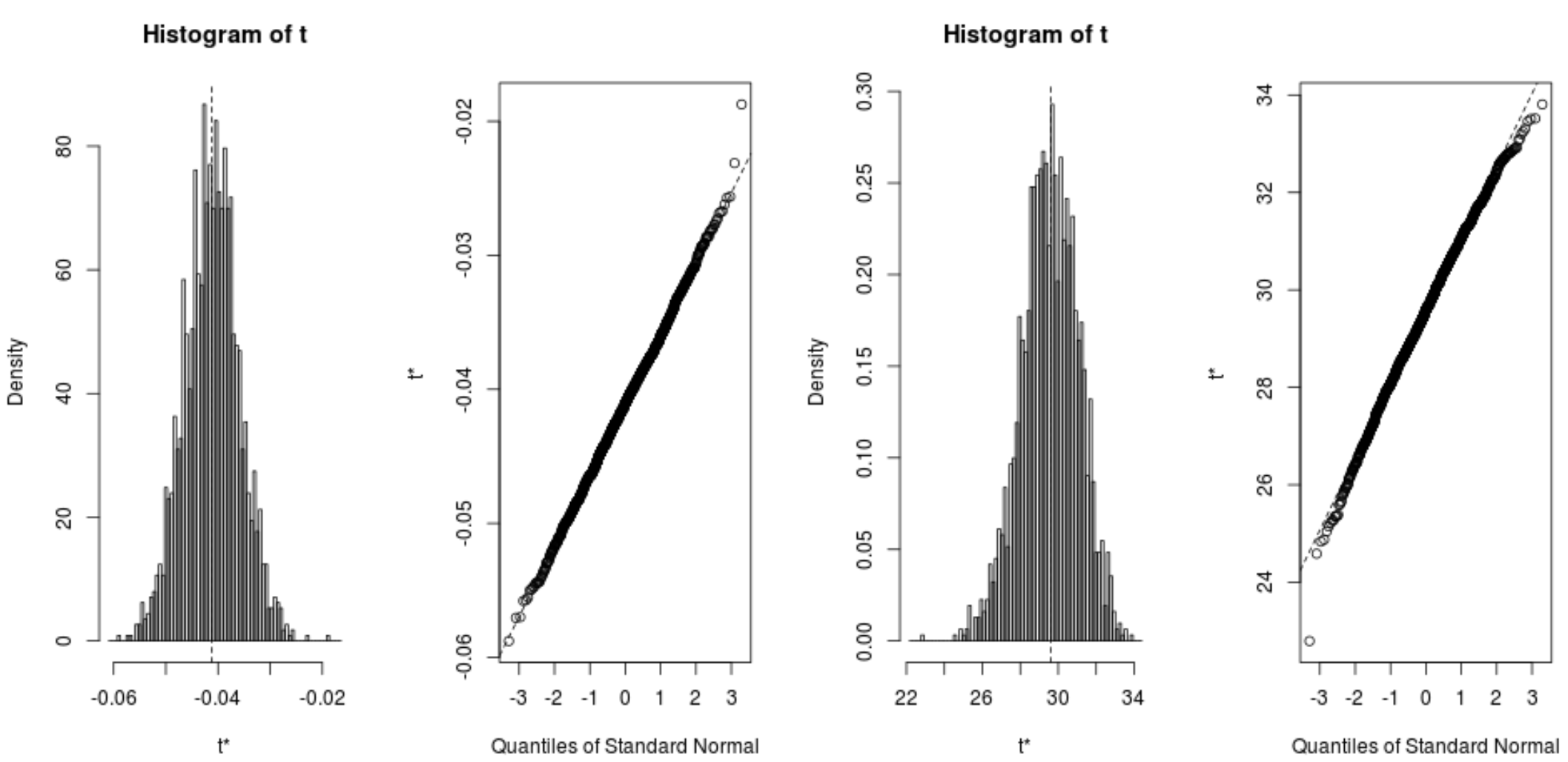
Możemy również użyć poniższego kodu do obliczenia 95% przedziałów ufności dla każdego współczynnika:
#calculate adjusted bootstrap percentile (BCa) intervals boot.ci(reps, type=" bca ", index=1) #intercept of model boot.ci(reps, type=" bca ", index=2) #disp predictor variable CALL: boot.ci(boot.out = reps, type = "bca", index = 1) Intervals: Level BCa 95% (26.78, 32.66) Calculations and Intervals on Original Scale BOOTSTRAP CONFIDENCE INTERVAL CALCULATIONS Based on 2000 bootstrap replicates CALL: boot.ci(boot.out = reps, type = "bca", index = 2) Intervals: Level BCa 95% (-0.0520, -0.0312) Calculations and Intervals on Original Scale
Z wyników widać, że bootstrapowe 95% przedziały ufności dla współczynników modelu są następujące:
- IC dla przechwytywania: (26,78, 32,66)
- CI dla disp : (-.0520, -.0312)
Dodatkowe zasoby
Jak wykonać prostą regresję liniową w R
Jak wykonać wielokrotną regresję liniową w R
Wprowadzenie do przedziałów ufności