Liniowa analiza dyskryminacyjna w r (krok po kroku)
Liniowa analiza dyskryminacyjna to metoda, której można użyć, gdy masz zestaw zmiennych predykcyjnych i chcesz sklasyfikować zmienną odpowiedzi na dwie lub więcej klas.
W tym samouczku przedstawiono krok po kroku przykład przeprowadzania liniowej analizy dyskryminacyjnej w języku R.
Krok 1: Załaduj niezbędne biblioteki
Najpierw załadujemy niezbędne biblioteki dla tego przykładu:
library (MASS)
library (ggplot2)
Krok 2: Załaduj dane
W tym przykładzie użyjemy zestawu danych iris wbudowanego w R. Poniższy kod pokazuje, jak załadować i wyświetlić ten zestaw danych:
#attach iris dataset to make it easy to work with attach(iris) #view structure of dataset str(iris) 'data.frame': 150 obs. of 5 variables: $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ... $ Sepal.Width: num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ... $Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ... $Petal.Width: num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ... $ Species: Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 ...
Widzimy, że zbiór danych zawiera w sumie 5 zmiennych i 150 obserwacji.
Na potrzeby tego przykładu zbudujemy liniowy model analizy dyskryminacyjnej, aby sklasyfikować, do jakiego gatunku należy dany kwiat.
W modelu wykorzystamy następujące zmienne predykcyjne:
- Długość rozdzielacza
- Sepal.Szerokość
- Płatek.Długość
- Płatek.Szerokość
Wykorzystamy je do przewidzenia zmiennej odpowiedzi Gatunek , która obsługuje następujące trzy potencjalne klasy:
- setosa
- wielokolorowy
- Wirginia
Krok 3: Skaluj dane
Jednym z kluczowych założeń liniowej analizy dyskryminacyjnej jest to, że każda ze zmiennych predykcyjnych ma tę samą wariancję. Prostym sposobem zapewnienia spełnienia tego założenia jest skalowanie każdej zmiennej w taki sposób, aby miała średnią 0 i odchylenie standardowe 1.
Możemy to zrobić szybko w R za pomocą funkcjiscale () :
#scale each predictor variable (ie first 4 columns)
iris[1:4] <- scale(iris[1:4])
Możemy użyć funkcji Apply() , aby sprawdzić, czy każda zmienna predykcyjna ma teraz średnią 0 i odchylenie standardowe 1:
#find mean of each predictor variable apply(iris[1:4], 2, mean) Sepal.Length Sepal.Width Petal.Length Petal.Width -4.484318e-16 2.034094e-16 -2.895326e-17 -3.663049e-17 #find standard deviation of each predictor variable apply(iris[1:4], 2, sd) Sepal.Length Sepal.Width Petal.Length Petal.Width 1 1 1 1
Krok 4: Utwórz próbki szkoleniowe i testowe
Następnie podzielimy zbiór danych na zbiór uczący, na którym będziemy trenować model, oraz zbiór testowy, na którym będziemy testować model:
#make this example reproducible set.seed(1) #Use 70% of dataset as training set and remaining 30% as testing set sample <- sample(c( TRUE , FALSE ), nrow (iris), replace = TRUE , prob =c(0.7,0.3)) train <- iris[sample, ] test <- iris[!sample, ]
Krok 5: Dostosuj model LDA
Następnie skorzystamy z funkcji lda() z pakietu MASS , aby dostosować model LDA do naszych danych:
#fit LDA model model <- lda(Species~., data=train) #view model output model Call: lda(Species ~ ., data = train) Prior probabilities of groups: setosa versicolor virginica 0.3207547 0.3207547 0.3584906 Group means: Sepal.Length Sepal.Width Petal.Length Petal.Width setosa -1.0397484 0.8131654 -1.2891006 -1.2570316 versicolor 0.1820921 -0.6038909 0.3403524 0.2208153 virginica 0.9582674 -0.1919146 1.0389776 1.1229172 Coefficients of linear discriminants: LD1 LD2 Sepal.Length 0.7922820 0.5294210 Sepal.Width 0.5710586 0.7130743 Petal.Length -4.0762061 -2.7305131 Petal.Width -2.0602181 2.6326229 Proportion of traces: LD1 LD2 0.9921 0.0079
Oto jak interpretować wyniki modelu:
Prawdopodobieństwa a priori grupowe: reprezentują proporcje każdego gatunku w zbiorze uczącym. Na przykład 35,8% wszystkich obserwacji w zbiorze uczącym dotyczyło gatunku virginica .
Średnie grupowe: wyświetlają średnie wartości każdej zmiennej predykcyjnej dla każdego gatunku.
Liniowe współczynniki dyskryminacyjne: przedstawiają liniową kombinację zmiennych predykcyjnych używanych do uczenia reguły decyzyjnej modelu LDA. Na przykład:
- LD1: 0,792 * długość działki + 0,571 * szerokość działki – 4,076 * długość płatka – 2,06 * szerokość płatka
- LD2: 0,529 * długość działki + 0,713 * szerokość działki – 2,731 * długość płatka + 2,63 * szerokość płatka
Proporcja śledzenia: Wyświetla procent separacji osiągnięty przez każdą liniową funkcję dyskryminacyjną.
Krok 6: Użyj modelu do przewidywania
Po dopasowaniu modelu przy użyciu naszych danych treningowych możemy go użyć do przewidywania danych testowych:
#use LDA model to make predictions on test data predicted <- predict (model, test) names(predicted) [1] "class" "posterior" "x"
Zwraca to listę z trzema zmiennymi:
- class: przewidywana klasa
- posterior: Prawdopodobieństwo późniejsze , że obserwacja należy do każdej klasy
- x: Dyskryminatory liniowe
Możemy szybko zwizualizować każdy z tych wyników dla pierwszych sześciu obserwacji w naszym testowym zbiorze danych:
#view predicted class for first six observations in test set head(predicted$class) [1] setosa setosa setosa setosa setosa setosa Levels: setosa versicolor virginica #view posterior probabilities for first six observations in test set head(predicted$posterior) setosa versicolor virginica 4 1 2.425563e-17 1.341984e-35 6 1 1.400976e-21 4.482684e-40 7 1 3.345770e-19 1.511748e-37 15 1 6.389105e-31 7.361660e-53 17 1 1.193282e-25 2.238696e-45 18 1 6.445594e-22 4.894053e-41 #view linear discriminants for first six observations in test set head(predicted$x) LD1 LD2 4 7.150360 -0.7177382 6 7.961538 1.4839408 7 7.504033 0.2731178 15 10.170378 1.9859027 17 8.885168 2.1026494 18 8.113443 0.7563902
Możemy użyć poniższego kodu, aby sprawdzić, dla jakiego procentu obserwacji model LDA poprawnie przewidział gatunek:
#find accuracy of model
mean(predicted$class==test$Species)
[1] 1
Okazuje się, że model poprawnie przewidział gatunek dla 100% obserwacji w naszym testowym zbiorze danych.
W prawdziwym świecie model LDA rzadko poprawnie przewiduje wyniki każdej klasy, ale ten zbiór danych tęczówki jest po prostu skonstruowany w taki sposób, że algorytmy uczenia maszynowego zwykle działają bardzo dobrze.
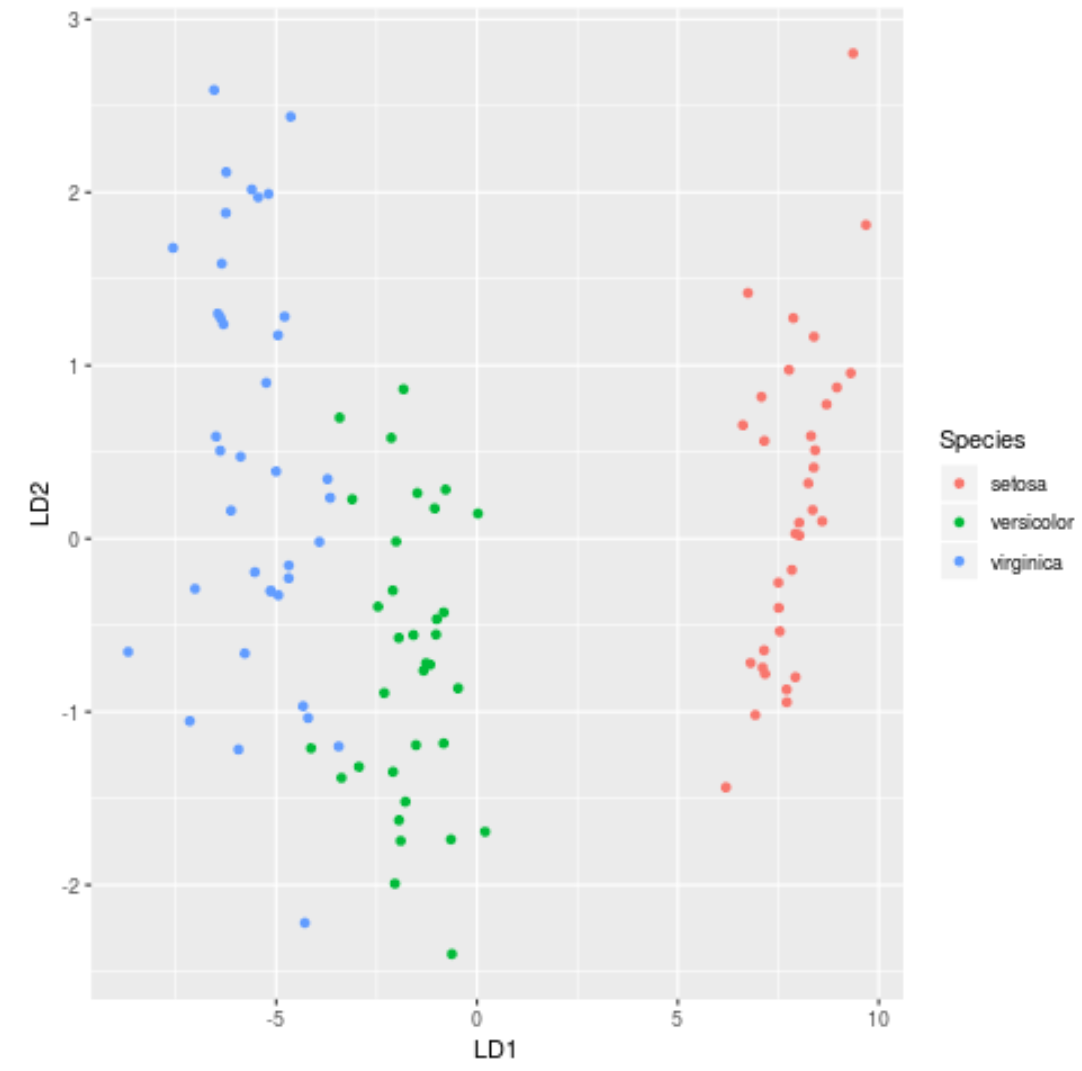
Krok 7: Wizualizuj wyniki
Na koniec możemy utworzyć wykres LDA, aby zwizualizować liniowe wyróżniki modelu i zwizualizować, jak dobrze oddziela on trzy różne gatunki w naszym zbiorze danych:
#define data to plot lda_plot <- cbind(train, predict(model)$x) #createplot ggplot(lda_plot, aes (LD1, LD2)) + geom_point( aes (color=Species))
Pełny kod R użyty w tym samouczku znajdziesz tutaj .