Jak używać pheatmap() w r do tworzenia map cieplnych
Możesz użyć funkcji pheatmap() pakietu pheatmap w R, aby stworzyć wysoce spersonalizowane mapy cieplne.
Poniższe przykłady pokazują, jak w praktyce wykorzystać tę funkcję z następującym fałszywym zbiorem danych:
#make this example reproducible set. seeds (1) #create matrix with fake data values data = matrix(rnorm(100), 20, 5) data[1:10, seq(1, 5, 1)] = data[1:10, seq(1, 5, 1)] + 3 data [11:20, seq(2, 5, 1)] = data [11:20, seq(2, 5, 1)] + 2 data [15:20, seq(2, 5, 1)] = data [15:20, seq(2, 5, 1)] + 4 #add column names and row names colnames(data) = paste(" T ", 1:5, sep = "") rownames(data) = paste(" Gene ", 1:20, sep="") #view matrx data T1 T2 T3 T4 T5 Gene1 2.37354619 3.918977 2.8354764 5.401618 2.431331 Gene2 3.18364332 3.782136 2.7466383 2.960760 2.864821 Gene3 2.16437139 3.074565 3.6969634 3.689739 4.178087 Gene4 4.59528080 1.010648 3.5566632 3.028002 1.476433 Gene5 3.32950777 3.619826 2.3112443 2.256727 3.593946 Gene6 2.17953162 2.943871 2.2925048 3.188792 3.332950 Gene7 3.48742905 2.844204 3.3645820 1.195041 4.063100 Gene8 3.73832471 1.529248 3.7685329 4.465555 2.695816 Gene9 3.57578135 2.521850 2.8876538 3.153253 3.370019 Gene10 2.69461161 3.417942 3.8811077 5.172612 3.267099 Gene11 1.51178117 3.358680 2.3981059 2.475510 1.457480 Gene12 0.38984324 1.897212 1.3879736 1.290054 3.207868 Gene13 -0.62124058 2.387672 2.3411197 2.610726 3.160403 Gene14 -2.21469989 1.946195 0.8706369 1.065902 2.700214 Gene15 1.12493092 4.622940 7.4330237 4.746367 7.586833 Gene16 -0.04493361 5.585005 7.9803999 6.291446 6.558486 Gene17 -0.01619026 5.605710 5.6327785 5.556708 4.723408 Gene18 0.94383621 5.940687 4.9558654 6.001105 5.426735 Gene19 0.82122120 7.100025 6.5697196 6.074341 4.775387 Gene20 0.59390132 6.763176 5.8649454 5.410479 5.526599
Przykład 1: Utwórz podstawową mapę cieplną
Możemy utworzyć mapę cieplną z domyślnymi ustawieniami w mapie cieplnej , aby zwizualizować wszystkie wartości macierzy:
library (heatmap)
#create basic heatmap
pheatmap(data)
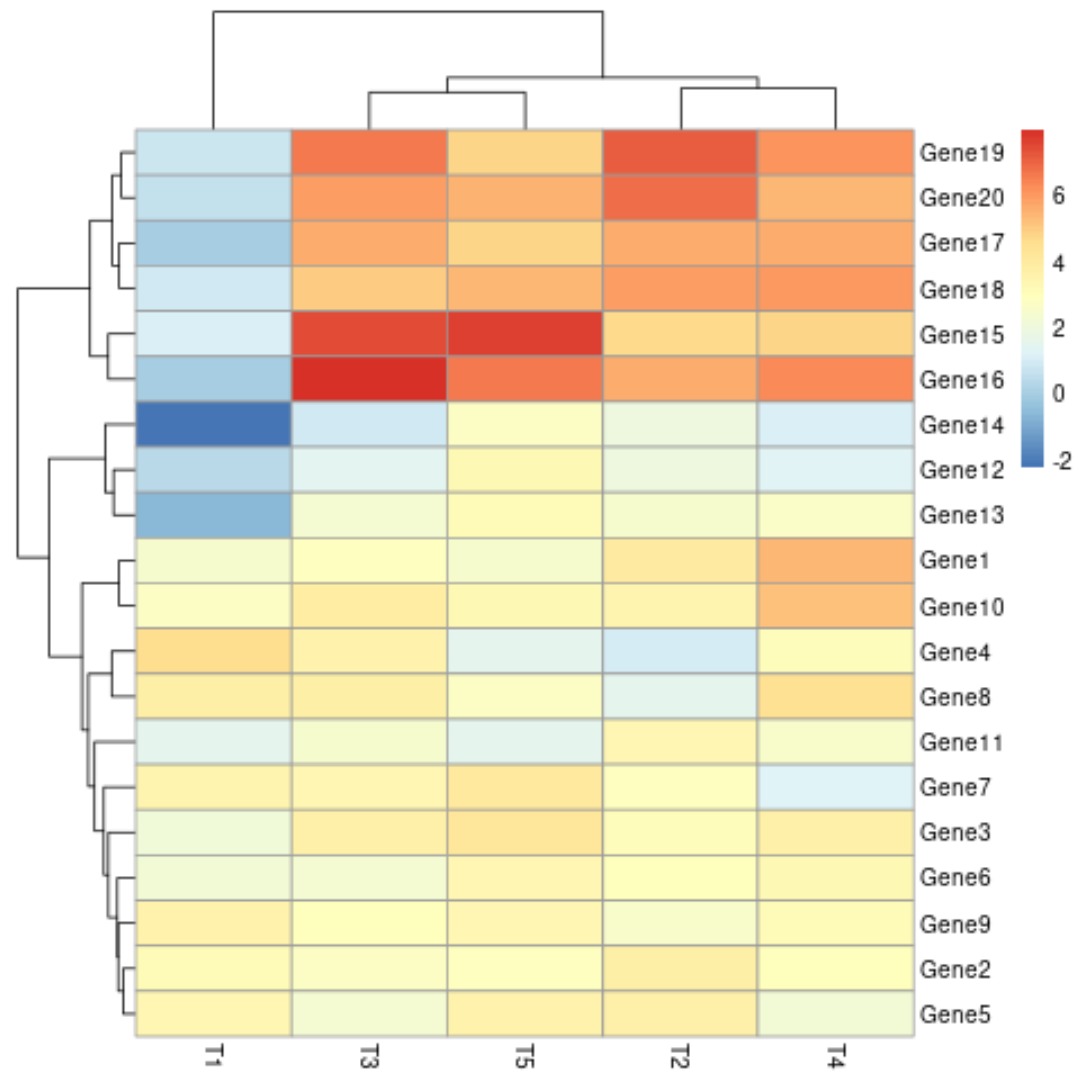
Przykład 2: Utwórz mapę cieplną z etykietami komórek
Możemy stworzyć za pomocą argumentów display_numbers i Fontsize_number , aby wyświetlić wartości liczbowe w każdej komórce mapy cieplnej z określonym rozmiarem czcionki:
library (heatmap)
#create heatmap with numerical labels in cells
pheatmap(data, display_numbers= TRUE , fontsize_number= 12 )
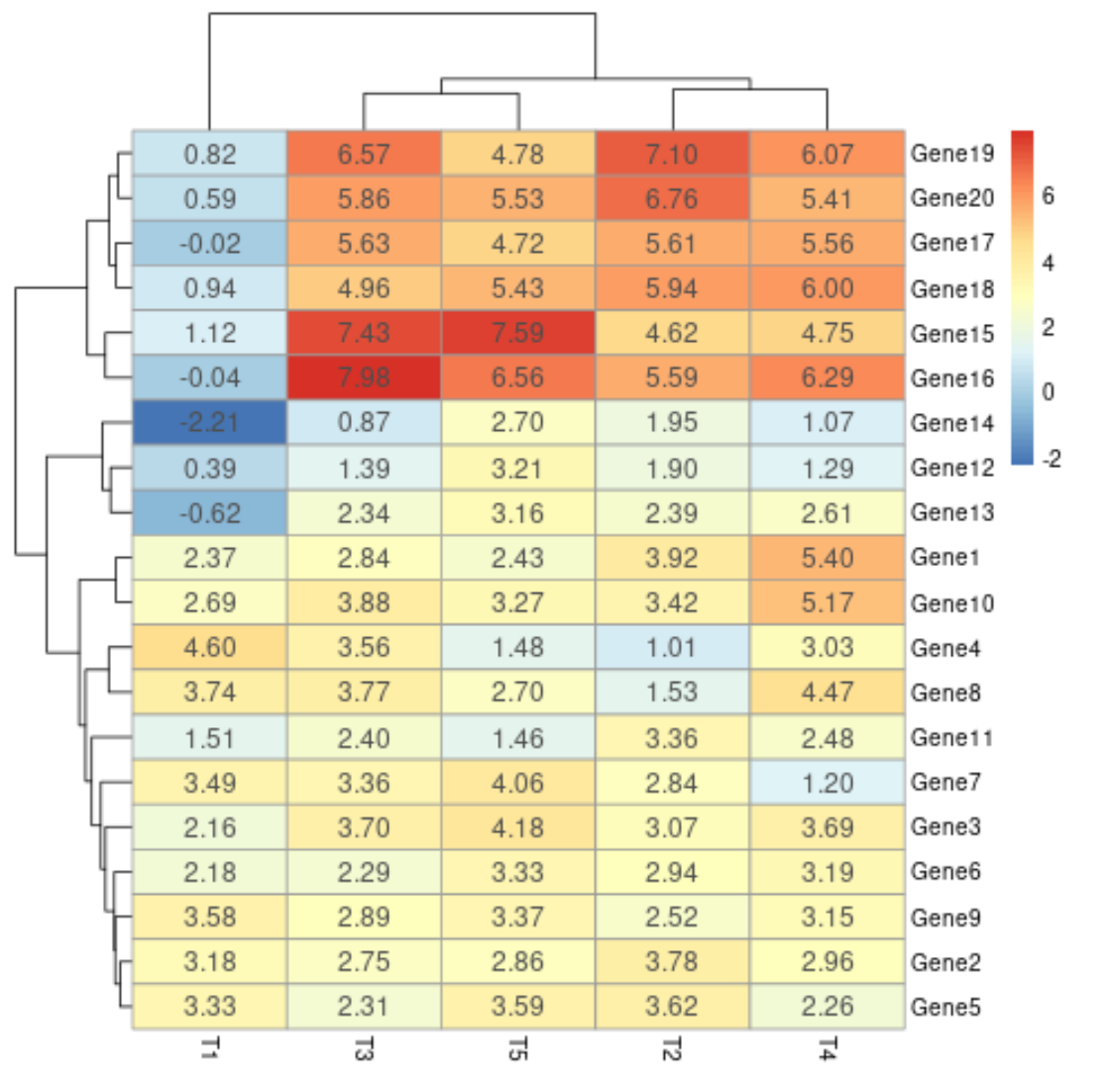
Uwaga : Domyślna wartość parametru numer_rozmiaru czcionki to 8 .
Przykład 3: Utwórz mapę cieplną z niestandardowymi kolorami
Możemy również użyć argumentu colorRampPalette , aby określić, jakich kolorów użyć dla niskich, średnich i wysokich wartości w mapie cieplnej:
library (heatmap)
#create heatmap with custom colors
pheatmap(data, color=colorRampPalette(c(" blue ", " white ", " red "))(20))
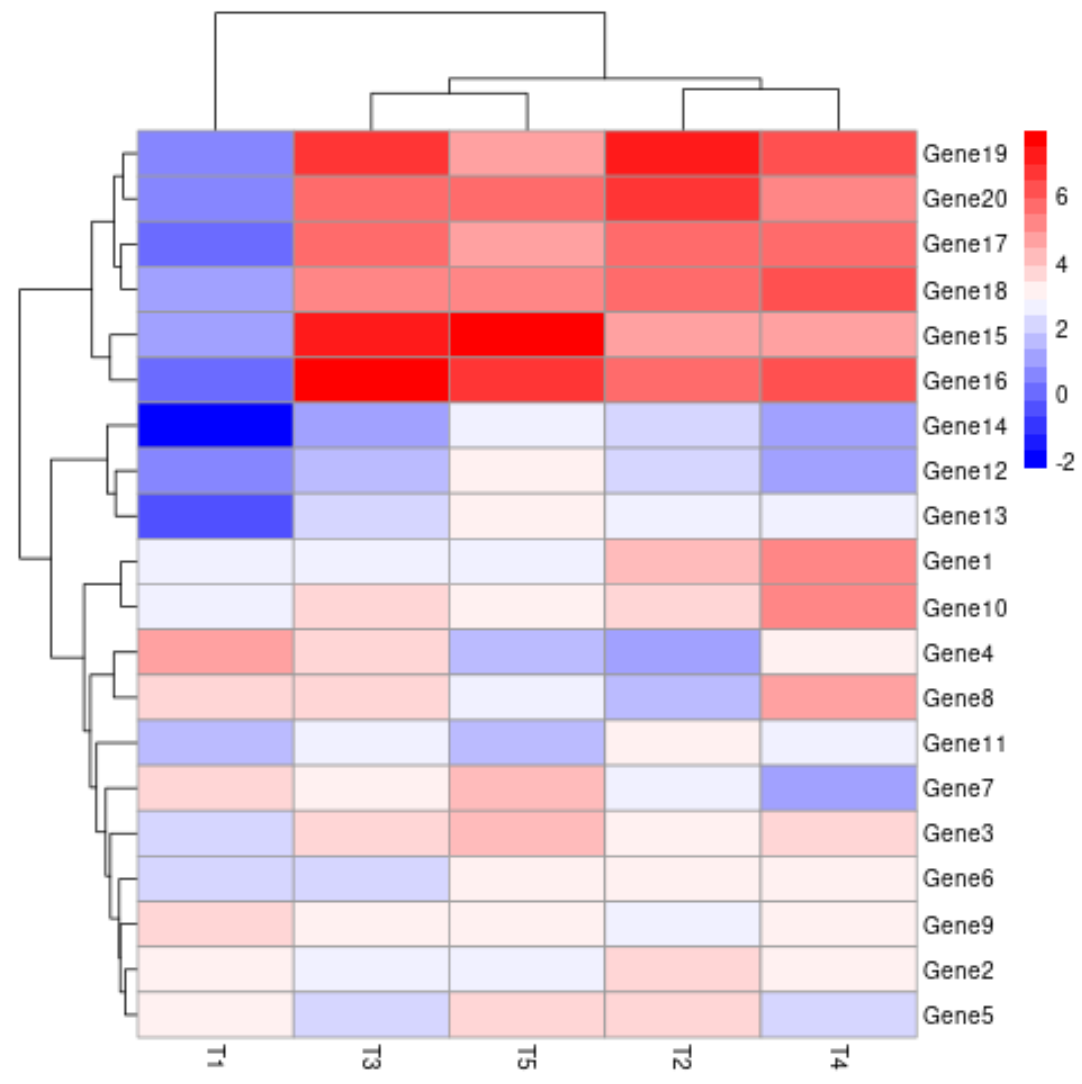
Niskie wartości są teraz wyświetlane na niebiesko , średnie wartości są wyświetlane na biało , a wysokie wartości są wyświetlane na czerwono .
Możesz dowolnie określić kolory, które chcesz stworzyć własną skalę kolorów dla mapy cieplnej.
Dodatkowe zasoby
Poniższe samouczki wyjaśniają, jak wykonywać inne typowe zadania w języku R:
Jak utworzyć mapę cieplną korelacji w R
Jak utworzyć mapę cieplną w R za pomocą ggplot2
Jak wykreślić dane kategoryczne w R