Regresja głównych składowych w r (krok po kroku)
Biorąc pod uwagę zbiór p zmiennych predykcyjnych i zmienną odpowiedzi, wielokrotna regresja liniowa wykorzystuje metodę znaną jako metoda najmniejszych kwadratów, aby zminimalizować resztową sumę kwadratów (RSS):
RSS = Σ(y i – ŷ i ) 2
Złoto:
- Σ : Grecki symbol oznaczający sumę
- y i : rzeczywista wartość odpowiedzi dla i-tej obserwacji
- ŷ i : Przewidywana wartość odpowiedzi na podstawie modelu wielokrotnej regresji liniowej
Jednakże, gdy zmienne predykcyjne są silnie skorelowane, współliniowość może stać się problemem. Może to sprawić, że szacunki współczynników modelu będą niewiarygodne i będą wykazywać dużą wariancję.
Jednym ze sposobów uniknięcia tego problemu jest zastosowanie regresji głównych składowych , która znajduje M kombinacji liniowych (zwanych „głównymi składowymi”) oryginalnych predyktorów p , a następnie wykorzystuje metodę najmniejszych kwadratów do dopasowania modelu regresji liniowej, wykorzystując główne składowe jako predyktory.
W tym samouczku przedstawiono krok po kroku przykład przeprowadzania regresji głównych składowych w języku R.
Krok 1: Załaduj niezbędne pakiety
Najłatwiejszym sposobem przeprowadzenia regresji głównych składowych w R jest użycie funkcji z pakietu pls .
#install pls package (if not already installed) install.packages(" pls ") load pls package library(pls)
Krok 2: Dostosuj model PCR
W tym przykładzie użyjemy wbudowanego zbioru danych R o nazwie mtcars , który zawiera dane o różnych typach samochodów:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
W tym przykładzie dopasujemy model regresji głównych składowych (PCR), wykorzystując hp jako zmienną odpowiedzi i następujące zmienne jako zmienne predykcyjne:
- mpg
- wyświetlacz
- gówno
- waga
- sek
Poniższy kod pokazuje, jak dopasować model PCR do tych danych. Zwróć uwagę na następujące argumenty:
- skala=PRAWDA : Mówi R, że każda zmienna predykcyjna powinna zostać przeskalowana tak, aby miała średnią 0 i odchylenie standardowe 1. Gwarantuje to, że żadna zmienna predykcyjna nie będzie miała zbyt dużego wpływu na model, jeśli jest mierzona w różnych jednostkach. .
- validation=”CV” : Mówi R, aby użył k-krotnej walidacji krzyżowej do oceny wydajności modelu. Zauważ, że domyślnie używa to k=10 fałd. Należy również pamiętać, że zamiast tego można określić „LOOCV”, aby przeprowadzić weryfikację krzyżową Leave-One-Out .
#make this example reproducible set.seed(1) #fit PCR model model <- pcr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
Krok 3: Wybierz liczbę głównych komponentów
Po dostosowaniu modelu musimy określić, ile głównych elementów warto zachować.
Aby to zrobić, po prostu spójrz na średni błąd kwadratowy testu (test RMSE) obliczony za pomocą walidacji krzyżowej k:
#view summary of model fitting
summary(model)
Data:
Y dimension: 32 1
Fit method: svdpc
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comp 2 comps 3 comps 4 comps 5 comps
CV 69.66 44.56 35.64 35.83 36.23 36.67
adjCV 69.66 44.44 35.27 35.43 35.80 36.20
TRAINING: % variance explained
1 comp 2 comps 3 comps 4 comps 5 comps
X 69.83 89.35 95.88 98.96 100.00
hp 62.38 81.31 81.96 81.98 82.03
W rezultacie znajdują się dwie interesujące tabele:
1. WALIDACJA: RMSEP
Ta tabela przedstawia test RMSE obliczony poprzez k-krotną walidację krzyżową. Możemy zobaczyć co następuje:
- Jeśli w modelu użyjemy tylko pierwotnego składnika, RMSE testu wyniesie 69,66 .
- Jeśli dodamy pierwszy główny składnik, test RMSE spadnie do 44,56.
- Jeśli dodamy drugi główny składnik, test RMSE spadnie do 35,64.
Widzimy, że dodanie dodatkowych głównych składników faktycznie powoduje wzrost RMSE testu. Wydaje się zatem, że optymalne byłoby wykorzystanie w ostatecznym modelu tylko dwóch głównych komponentów.
2. SZKOLENIE: Wyjaśniono % wariancji
Ta tabela pokazuje nam procent wariancji zmiennej odpowiedzi wyjaśnionej przez główne składniki. Możemy zobaczyć co następuje:
- Używając tylko pierwszego składnika głównego, możemy wyjaśnić 69,83% zmienności zmiennej odpowiedzi.
- Dodając drugi główny składnik, możemy wyjaśnić 89,35% zmienności zmiennej odpowiedzi.
Należy zauważyć, że nadal będziemy w stanie wyjaśnić większą wariancję, używając większej liczby głównych składników, ale widzimy, że dodanie więcej niż dwóch głównych składników w rzeczywistości nie zwiększa znacząco procentu wyjaśnionej wariancji.
Możemy również wizualizować test RMSE (wraz z testem MSE i testem R-kwadrat) jako funkcję liczby głównych składników za pomocą funkcji validationplot() .
#visualize cross-validation plots
validationplot(model)
validationplot(model, val.type="MSEP")
validationplot(model, val.type="R2")
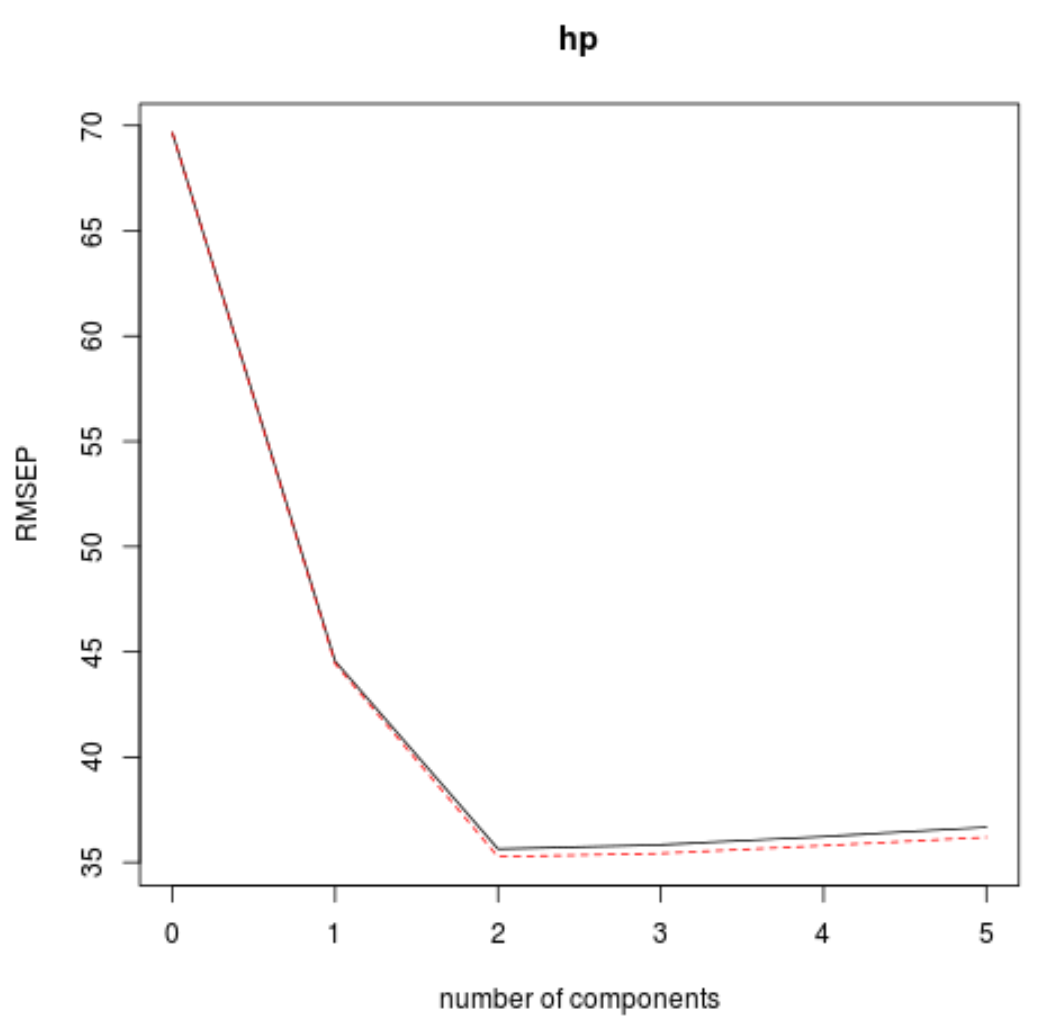
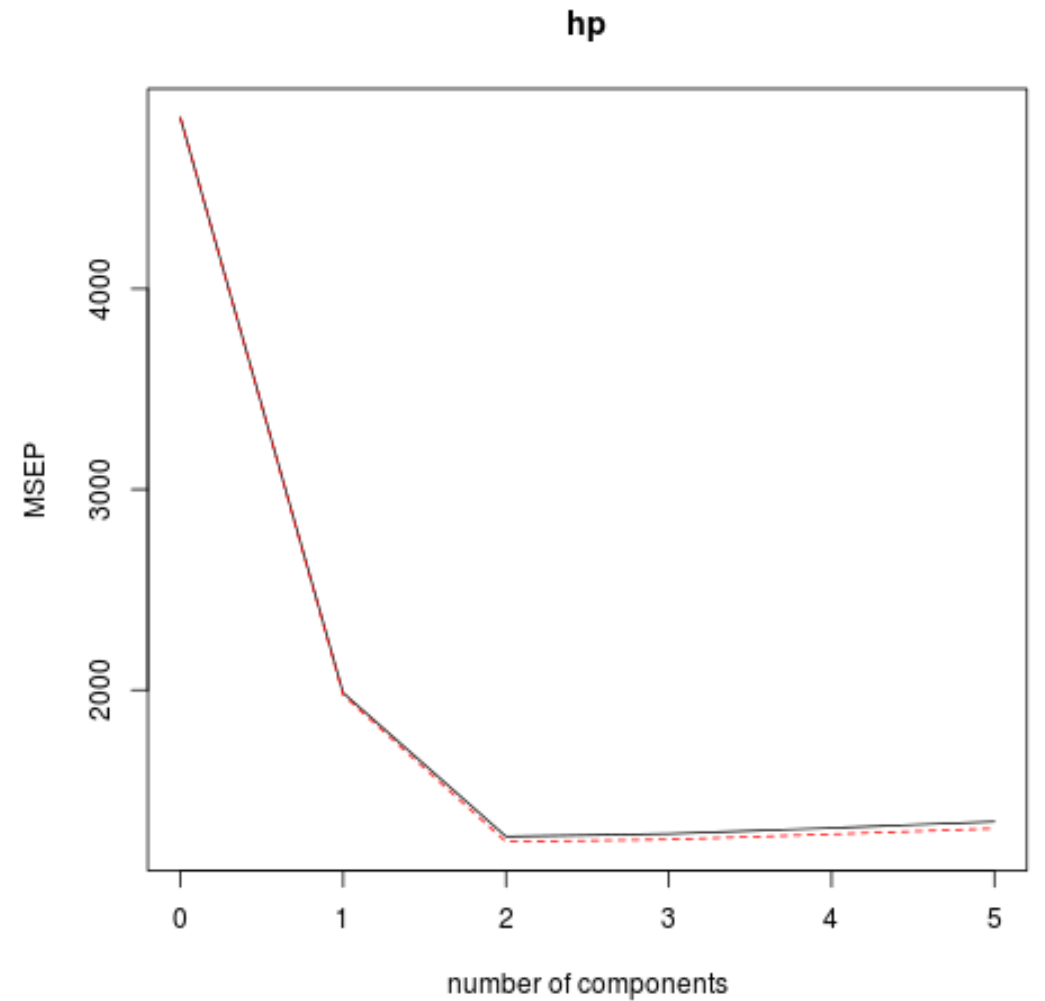
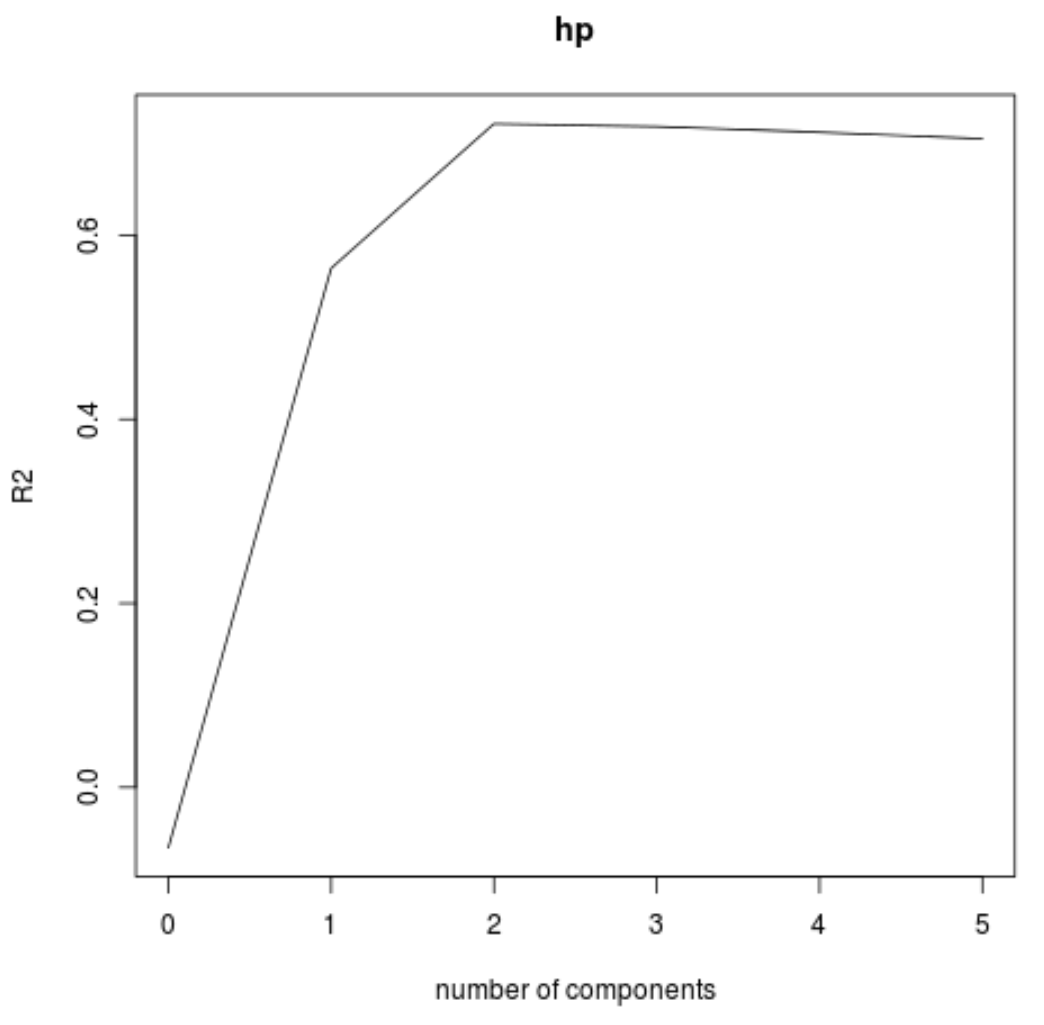
Na każdym wykresie widzimy, że dopasowanie modelu poprawia się po dodaniu dwóch głównych składników, ale ma tendencję do pogarszania się, gdy dodajemy więcej głównych składników.
Zatem optymalny model obejmuje tylko dwa pierwsze główne elementy.
Krok 4: Użyj ostatecznego modelu do przewidywania
Możemy wykorzystać ostateczny dwugłówny model PCR do przewidywania nowych obserwacji.
Poniższy kod pokazuje, jak podzielić oryginalny zbiór danych na zbiór uczący i testowy oraz użyć modelu PCR z dwoma głównymi składnikami do przewidywania zbioru testowego.
#define training and testing sets train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")] y_test <- mtcars[26: nrow (mtcars), c("hp")] test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")] #use model to make predictions on a test set model <- pcr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ") pcr_pred <- predict(model, test, ncomp= 2 ) #calculate RMSE sqrt ( mean ((pcr_pred - y_test)^2)) [1] 56.86549
Widzimy, że RMSE testu wynosi 56,86549 . Jest to średnie odchylenie pomiędzy przewidywaną wartością KM a obserwowaną wartością KM dla obserwacji zestawu testowego.
Pełne wykorzystanie kodu R w tym przykładzie można znaleźć tutaj .