Jak wykonać skalowanie wielowymiarowe w r (z przykładem)
W statystyce skalowanie wielowymiarowe jest sposobem na wizualizację podobieństwa obserwacji w zbiorze danych w abstrakcyjnej przestrzeni kartezjańskiej (zwykle przestrzeni 2D).
Najłatwiejszym sposobem skalowania wielowymiarowego w R jest użycie wbudowanej funkcji cmdscale() , która wykorzystuje następującą podstawową składnię:
cmdscale(d, eig = FALSE, k = 2, …)
Złoto:
- d : Macierz odległości obliczana zazwyczaj za pomocą funkcji dist() .
- eig : czy zwracać wartości własne.
- k : liczba wymiarów, w których należy wyświetlić dane. Wartość domyślna to 2 .
Poniższy przykład pokazuje, jak w praktyce wykorzystać tę funkcję.
Przykład: Skalowanie wielowymiarowe w R
Załóżmy, że mamy następującą ramkę danych w R, która zawiera informacje o różnych koszykarzach:
#create data frame df <- data. frame (points=c(4, 4, 6, 7, 8, 14, 16, 19, 25, 25, 28), assists=c(3, 2, 2, 5, 4, 8, 7, 6, 8, 10, 11), blocks=c(7, 3, 6, 7, 5, 8, 8, 4, 2, 2, 1), rebounds=c(4, 5, 5, 6, 5, 8, 10, 4, 3, 2, 2)) #add row names row. names (df) <- LETTERS[1:11] #view data frame df points assists blocks rebounds A 4 3 7 4 B 4 2 3 5 C 6 2 6 5 D 7 5 7 6 E 8 4 5 5 F 14 8 8 8 G 16 7 8 10 H 19 6 4 4 I 25 8 2 3 D 25 10 2 2 K 28 11 1 2
Możemy użyć poniższego kodu, aby wykonać skalowanie wielowymiarowe za pomocą funkcji cmdscale() i zwizualizować wyniki w przestrzeni 2D:
#calculate distance matrix
d <- dist(df)
#perform multidimensional scaling
fit <- cmdscale(d, eig= TRUE , k= 2 )
#extract (x, y) coordinates of multidimensional scaling
x <- fit$points[,1]
y <- fit$points[,2]
#create scatterplot
plot(x, y, xlab=" Coordinate 1 ", ylab=" Coordinate 2 ",
main=" Multidimensional Scaling Results ", type=" n ")
#add row names of data frame as labels
text(x, y, labels=row. names (df))
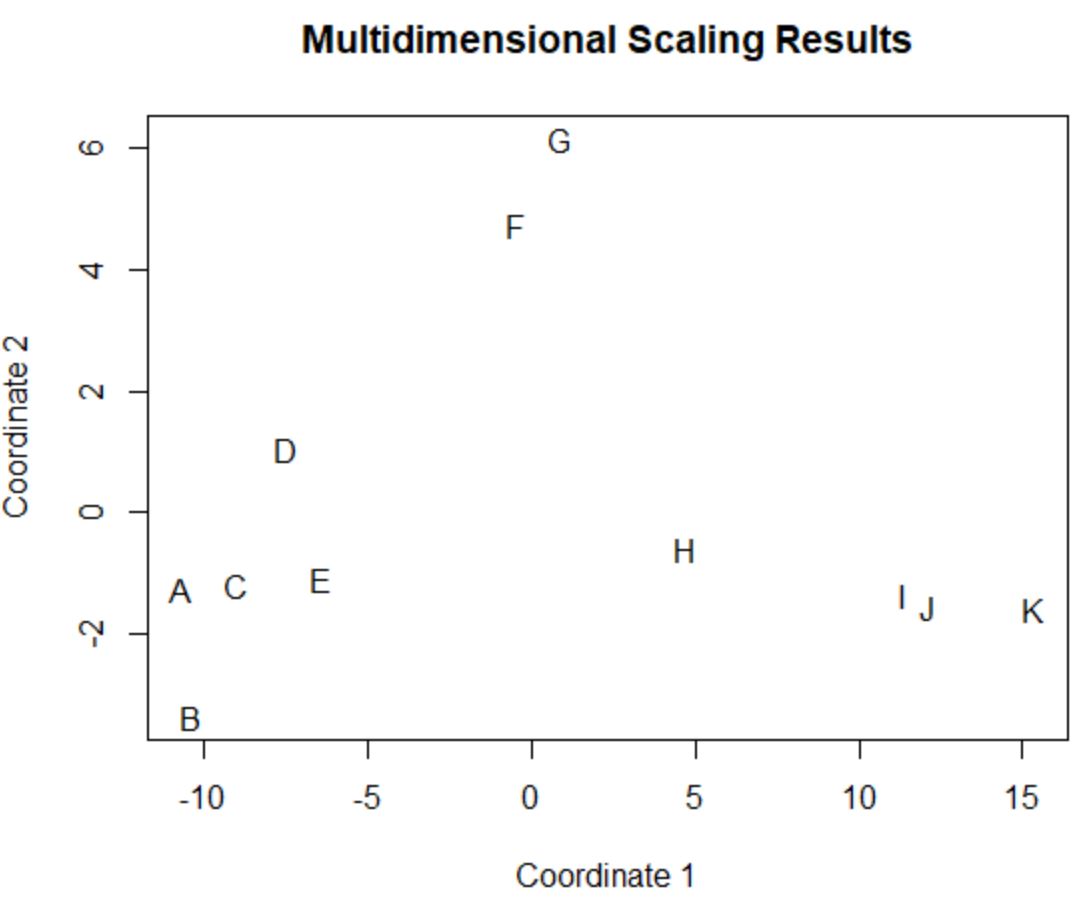
Gracze w oryginalnej ramce danych, którzy mają podobne wartości w oryginalnych czterech kolumnach (punkty, asysty, bloki i zbiórki), są blisko siebie na wykresie.
Na przykład gracze A i C są blisko siebie. Oto ich wartości z oryginalnej ramki danych:
#view data frame values for players A and C df[rownames(df) %in% c(' A ', ' C '), ] points assists blocks rebounds A 4 3 7 4 C 6 2 6 5
Ich wartości punktów, asyst, bloków i zbiórek są dość podobne, co wyjaśnia, dlaczego są tak blisko siebie na wykresie 2D.
Dla kontrastu, rozważ graczy B i K , którzy są daleko od siebie w fabule.
Jeśli odniesiemy się do ich wartości w danych oryginalnych, zobaczymy, że są one zupełnie inne:
#view data frame values for players B and K df[rownames(df) %in% c(' B ', ' K '), ] points assists blocks rebounds B 4 2 3 5 K 28 11 1 2
Zatem wykres 2D to dobry sposób na wizualizację podobieństwa każdego gracza we wszystkich zmiennych w ramce danych.
Gracze o podobnych statystykach są zgrupowani blisko siebie, podczas gdy gracze o bardzo różnych statystykach są dalej od siebie w fabule.
Pamiętaj, że możesz także wyodrębnić dokładne współrzędne (x, y) każdego gracza na wykresie, wpisując fit , czyli nazwę zmiennej, w której zapisaliśmy wyniki funkcji cmdscale() :
#view (x, y) coordinates of points in the plot
fit
[,1] [,2]
A -10.6617577 -1.2511291
B -10.3858237 -3.3450473
C -9.0330408 -1.1968116
D -7.4905743 1.0578445
E -6.4021114 -1.0743669
F -0.4618426 4.7392534
G 0.8850934 6.1460850
H 4.7352436 -0.6004609
I 11.3793381 -1.3563398
J 12.0844168 -1.5494108
K 15.3510585 -1.5696166
Dodatkowe zasoby
Poniższe samouczki wyjaśniają, jak wykonywać inne typowe zadania w języku R:
Jak normalizować dane w R
Jak centrum danych w R
Jak usunąć wartości odstające w R