Análise discriminante linear em r (passo a passo)
A análise discriminante linear é um método que você pode usar quando possui um conjunto de variáveis preditoras e deseja classificar uma variável de resposta em duas ou mais classes.
Este tutorial fornece um exemplo passo a passo de como realizar análise discriminante linear em R.
Etapa 1: carregue as bibliotecas necessárias
Primeiro, carregaremos as bibliotecas necessárias para este exemplo:
library (MASS)
library (ggplot2)
Etapa 2: carregar dados
Para este exemplo, usaremos o conjunto de dados iris integrado em R. O código a seguir mostra como carregar e exibir este conjunto de dados:
#attach iris dataset to make it easy to work with attach(iris) #view structure of dataset str(iris) 'data.frame': 150 obs. of 5 variables: $ Sepal.Length: num 5.1 4.9 4.7 4.6 5 5.4 4.6 5 4.4 4.9 ... $ Sepal.Width: num 3.5 3 3.2 3.1 3.6 3.9 3.4 3.4 2.9 3.1 ... $Petal.Length: num 1.4 1.4 1.3 1.5 1.4 1.7 1.4 1.5 1.4 1.5 ... $Petal.Width: num 0.2 0.2 0.2 0.2 0.2 0.4 0.3 0.2 0.2 0.1 ... $ Species: Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 1 1 1 1 ...
Podemos ver que o conjunto de dados contém 5 variáveis e 150 observações no total.
Para este exemplo, construiremos um modelo de análise discriminante linear para classificar a qual espécie pertence uma determinada flor.
Usaremos as seguintes variáveis preditoras no modelo:
- Sépala.comprimento
- Sépala.Largura
- Pétala.Comprimento
- Pétala.Largura
E vamos usá-los para prever a variável de resposta Species , que suporta as três classes potenciais a seguir:
- setosa
- versicolor
- Virgínia
Etapa 3: dimensionar os dados
Uma das principais suposições da análise discriminante linear é que cada uma das variáveis preditoras tem a mesma variância. Uma maneira simples de garantir que essa suposição seja atendida é dimensionar cada variável de modo que ela tenha média 0 e desvio padrão 1.
Podemos fazer isso rapidamente em R usando a função scale() :
#scale each predictor variable (ie first 4 columns)
iris[1:4] <- scale(iris[1:4])
Podemos usar a função apply() para verificar se cada variável preditora agora tem uma média de 0 e um desvio padrão de 1:
#find mean of each predictor variable apply(iris[1:4], 2, mean) Sepal.Length Sepal.Width Petal.Length Petal.Width -4.484318e-16 2.034094e-16 -2.895326e-17 -3.663049e-17 #find standard deviation of each predictor variable apply(iris[1:4], 2, sd) Sepal.Length Sepal.Width Petal.Length Petal.Width 1 1 1 1
Etapa 4: criar amostras de treinamento e teste
A seguir, dividiremos o conjunto de dados em um conjunto de treinamento para treinar o modelo e um conjunto de teste para testar o modelo:
#make this example reproducible set.seed(1) #Use 70% of dataset as training set and remaining 30% as testing set sample <- sample(c( TRUE , FALSE ), nrow (iris), replace = TRUE , prob =c(0.7,0.3)) train <- iris[sample, ] test <- iris[!sample, ]
Etapa 5: ajuste o modelo LDA
A seguir, usaremos a função lda() do pacote MASS para adaptar o modelo LDA aos nossos dados:
#fit LDA model model <- lda(Species~., data=train) #view model output model Call: lda(Species ~ ., data = train) Prior probabilities of groups: setosa versicolor virginica 0.3207547 0.3207547 0.3584906 Group means: Sepal.Length Sepal.Width Petal.Length Petal.Width setosa -1.0397484 0.8131654 -1.2891006 -1.2570316 versicolor 0.1820921 -0.6038909 0.3403524 0.2208153 virginica 0.9582674 -0.1919146 1.0389776 1.1229172 Coefficients of linear discriminants: LD1 LD2 Sepal.Length 0.7922820 0.5294210 Sepal.Width 0.5710586 0.7130743 Petal.Length -4.0762061 -2.7305131 Petal.Width -2.0602181 2.6326229 Proportion of traces: LD1 LD2 0.9921 0.0079
Veja como interpretar os resultados do modelo:
Probabilidades anteriores do grupo: representam as proporções de cada espécie no conjunto de treinamento. Por exemplo, 35,8% de todas as observações no conjunto de treinamento foram para a espécie virginica .
Médias de grupo: exibem os valores médios de cada variável preditora para cada espécie.
Coeficientes discriminantes lineares: exibem a combinação linear de variáveis preditoras usadas para treinar a regra de decisão do modelo LDA. Por exemplo:
- LD1: 0,792 * comprimento da sépala + 0,571 * largura da sépala – 4,076 * comprimento da pétala – 2,06 * largura da pétala
- LD2: 0,529 * comprimento da sépala + 0,713 * largura da sépala – 2,731 * comprimento da pétala + 2,63 * largura da pétala
Proporção de rastreamento: exibem a porcentagem de separação alcançada por cada função discriminante linear.
Etapa 6: use o modelo para fazer previsões
Depois de ajustar o modelo usando nossos dados de treinamento, podemos usá-lo para fazer previsões sobre nossos dados de teste:
#use LDA model to make predictions on test data predicted <- predict (model, test) names(predicted) [1] "class" "posterior" "x"
Isso retorna uma lista com três variáveis:
- classe: a classe prevista
- posterior: A probabilidade posterior de que uma observação pertença a cada classe
- x: Discriminantes lineares
Podemos visualizar rapidamente cada um desses resultados para as primeiras seis observações em nosso conjunto de dados de teste:
#view predicted class for first six observations in test set head(predicted$class) [1] setosa setosa setosa setosa setosa setosa Levels: setosa versicolor virginica #view posterior probabilities for first six observations in test set head(predicted$posterior) setosa versicolor virginica 4 1 2.425563e-17 1.341984e-35 6 1 1.400976e-21 4.482684e-40 7 1 3.345770e-19 1.511748e-37 15 1 6.389105e-31 7.361660e-53 17 1 1.193282e-25 2.238696e-45 18 1 6.445594e-22 4.894053e-41 #view linear discriminants for first six observations in test set head(predicted$x) LD1 LD2 4 7.150360 -0.7177382 6 7.961538 1.4839408 7 7.504033 0.2731178 15 10.170378 1.9859027 17 8.885168 2.1026494 18 8.113443 0.7563902
Podemos usar o código a seguir para ver qual porcentagem de observações o modelo LDA previu corretamente a espécie:
#find accuracy of model
mean(predicted$class==test$Species)
[1] 1
Acontece que o modelo previu corretamente as espécies para 100% das observações em nosso conjunto de dados de teste.
No mundo real, um modelo LDA raramente prevê corretamente os resultados de cada classe, mas esse conjunto de dados da íris é simplesmente construído de uma forma que os algoritmos de aprendizado de máquina tendem a ter um desempenho muito bom.
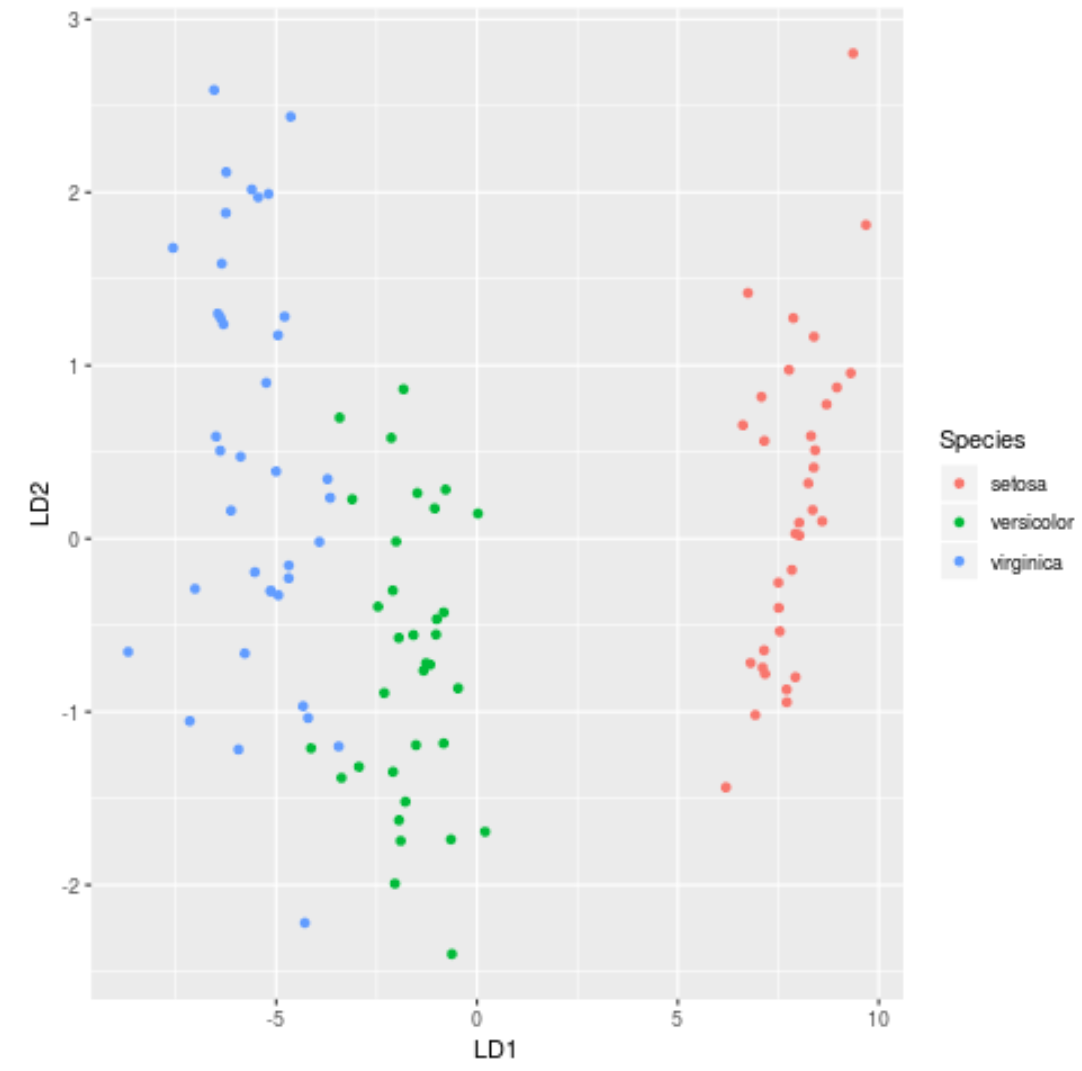
Passo 7: Visualize os resultados
Finalmente, podemos criar um gráfico LDA para visualizar os discriminantes lineares do modelo e visualizar quão bem ele separa as três espécies diferentes em nosso conjunto de dados:
#define data to plot lda_plot <- cbind(train, predict(model)$x) #createplot ggplot(lda_plot, aes (LD1, LD2)) + geom_point( aes (color=Species))
Você pode encontrar o código R completo usado neste tutorial aqui .