Regressão ridge em r (passo a passo)
A regressão Ridge é um método que podemos usar para ajustar um modelo de regressão quando a multicolinearidade está presente nos dados.
Resumindo, a regressão de mínimos quadrados tenta encontrar estimativas de coeficientes que minimizem a soma residual dos quadrados (RSS):
RSS = Σ(y i – ŷ i )2
Ouro:
- Σ : Um símbolo grego que significa soma
- y i : o valor real da resposta para a i-ésima observação
- ŷ i : O valor da resposta prevista com base no modelo de regressão linear múltipla
Por outro lado, a regressão de crista procura minimizar o seguinte:
RSS + λΣβj 2
onde j vai de 1 a p variáveis preditoras e λ ≥ 0.
Este segundo termo da equação é conhecido como penalidade de retirada . Na regressão de crista, selecionamos um valor para λ que produz o teste MSE mais baixo possível (erro quadrático médio).
Este tutorial fornece um exemplo passo a passo de como realizar a regressão de crista em R.
Etapa 1: carregar dados
Para este exemplo, usaremos o conjunto de dados integrado do R chamado mtcars . Usaremos hp como variável de resposta e as seguintes variáveis como preditores:
- mpg
- peso
- merda
- qsec
Para realizar a regressão de crista, usaremos funções do pacote glmnet . Este pacote requer que a variável de resposta seja um vetor e que o conjunto de variáveis preditoras seja da classe data.matrix .
O código a seguir mostra como definir nossos dados:
#define response variable
y <- mtcars$hp
#define matrix of predictor variables
x <- data.matrix(mtcars[, c('mpg', 'wt', 'drat', 'qsec')])
Etapa 2: ajustar o modelo de regressão Ridge
A seguir, usaremos a função glmnet() para ajustar o modelo de regressão Ridge e especificar alpha=0 .
Observe que definir alfa igual a 1 equivale a usar a regressão Lasso e definir alfa com um valor entre 0 e 1 equivale a usar uma rede elástica.
Observe também que a regressão de crista exige que os dados sejam padronizados de modo que cada variável preditora tenha uma média de 0 e um desvio padrão de 1.
Felizmente, glmnet() faz essa padronização automaticamente para você. Se você já padronizou as variáveis, poderá especificar standardize=False .
library (glmnet)
#fit ridge regression model
model <- glmnet(x, y, alpha = 0 )
#view summary of model
summary(model)
Length Class Mode
a0 100 -none- numeric
beta 400 dgCMatrix S4
df 100 -none- numeric
dim 2 -none- numeric
lambda 100 -none- numeric
dev.ratio 100 -none- numeric
nulldev 1 -none- numeric
npasses 1 -none- numeric
jerr 1 -none- numeric
offset 1 -none- logical
call 4 -none- call
nobs 1 -none- numeric
Etapa 3: escolha um valor ideal para Lambda
A seguir, identificaremos o valor lambda que produz o menor erro quadrático médio de teste (MSE) usando validação cruzada k-fold .
Felizmente, glmnet tem a função cv.glmnet() que executa automaticamente a validação cruzada k-fold usando k = 10 vezes.
#perform k-fold cross-validation to find optimal lambda value
cv_model <- cv. glmnet (x, y, alpha = 0 )
#find optimal lambda value that minimizes test MSE
best_lambda <- cv_model$ lambda . min
best_lambda
[1] 10.04567
#produce plot of test MSE by lambda value
plot(cv_model)
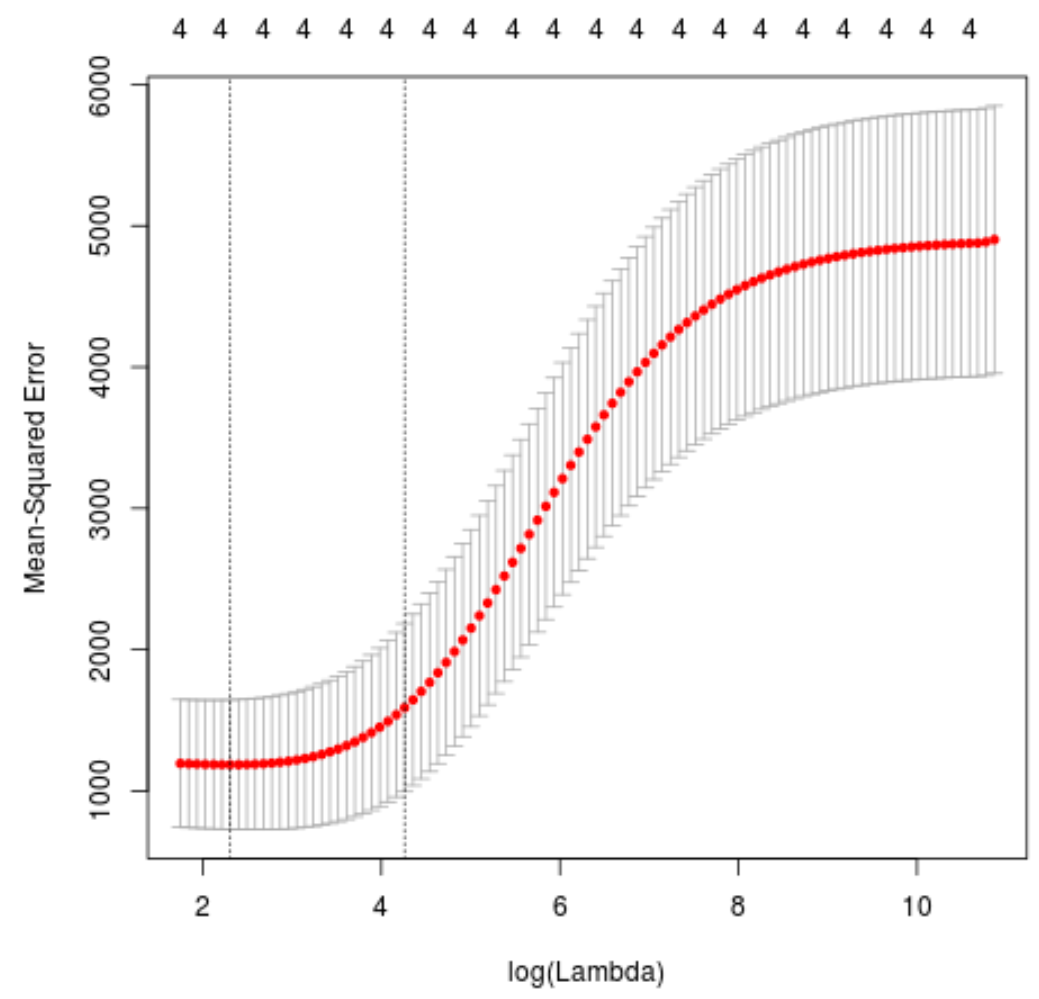
O valor lambda que minimiza o teste MSE é 10.04567 .
Passo 4: Analise o modelo final
Finalmente, podemos analisar o modelo final produzido pelo valor lambda ótimo.
Podemos usar o seguinte código para obter as estimativas dos coeficientes para este modelo:
#find coefficients of best model
best_model <- glmnet(x, y, alpha = 0 , lambda = best_lambda)
coef(best_model)
5 x 1 sparse Matrix of class "dgCMatrix"
s0
(Intercept) 475.242646
mpg -3.299732
wt 19.431238
drat -1.222429
qsec -17.949721
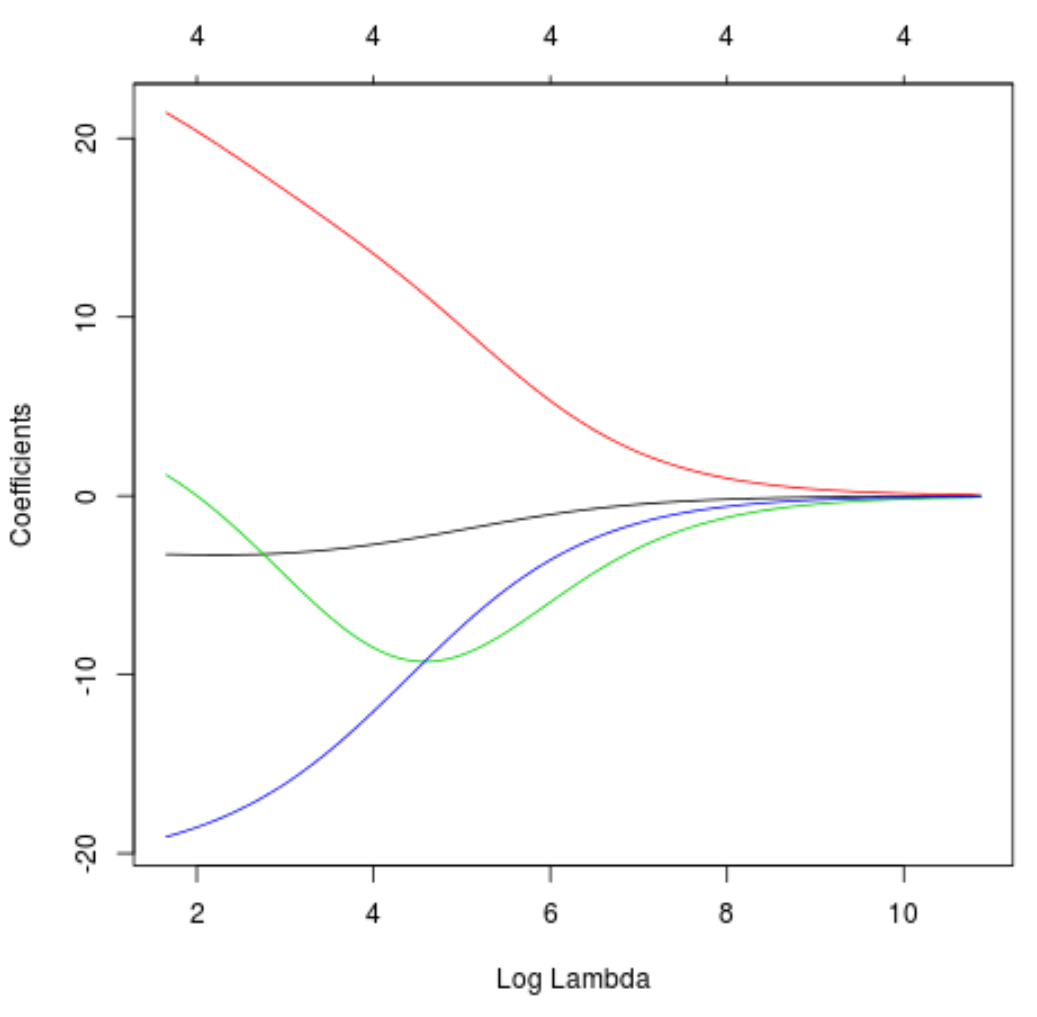
Também podemos produzir um gráfico Trace para visualizar como as estimativas dos coeficientes mudaram devido ao aumento no lambda:
#produce Ridge trace plot
plot(model, xvar = " lambda ")
Finalmente, podemos calcular o R-quadrado do modelo nos dados de treinamento:
#use fitted best model to make predictions
y_predicted <- predict (model, s = best_lambda, newx = x)
#find OHS and SSE
sst <- sum ((y - mean (y))^2)
sse <- sum ((y_predicted - y)^2)
#find R-Squared
rsq <- 1 - sse/sst
rsq
[1] 0.7999513
O R ao quadrado é 0,7999513 . Ou seja, o melhor modelo conseguiu explicar 79,99% da variação nos valores de resposta dos dados de treinamento.
Você pode encontrar o código R completo usado neste exemplo aqui .