Teste brown-forsythe em r: exemplo passo a passo
Uma ANOVA unidirecional é usada para determinar se há ou não uma diferença significativa entre as médias de três ou mais grupos independentes.
Uma das suposições de uma ANOVA unidirecional é que as variâncias das populações das quais as amostras são extraídas são iguais.
Uma das maneiras mais comuns de testar isso é usar um teste de Brown-Forsythe , que é um teste estatístico que usa as seguintes suposições :
- H 0 : As variâncias entre as populações são iguais.
- HA : As diferenças entre as populações não são iguais.
Se o valor p do teste estiver abaixo de um certo nível de significância (por exemplo, α = 0,05), então rejeitamos a hipótese nula e concluímos que as variâncias não são iguais entre diferentes populações.
Este tutorial fornece um exemplo passo a passo de como realizar um teste de Brown-Forsythe em R.
Passo 1: Insira os dados
Suponha que gostaríamos de saber se três programas de treino diferentes levam ou não a diferentes níveis de perda de peso.
Para testar isso, recrutamos 90 pessoas e designamos aleatoriamente 30 para usar cada programa. Em seguida, medimos a perda de peso de cada pessoa após um mês.
O seguinte conjunto de dados contém informações sobre a quantidade de peso perdido com cada programa:
#make this example reproducible set.seed(0) #create data frame data <- data.frame(program = as . factor ( rep (c(" A ", " B ", " C "), each = 30)), weight_loss = c( runif (30, 0, 3), runif (30, 0, 5), runif (30, 1, 7))) #view first six rows of data frame head(data) # program weight_loss #1 A 2.6900916 #2 A 0.7965260 #3 A 1.1163717 #4 A 1.7185601 #5 A 2.7246234 #6 A 0.6050458
Etapa 2: resumir e visualizar os dados
Antes de realizar um teste de Brown-Forsythe, podemos criar box plots para visualizar a variação na perda de peso para cada grupo:
boxplot(weight_loss ~ program, data = data)
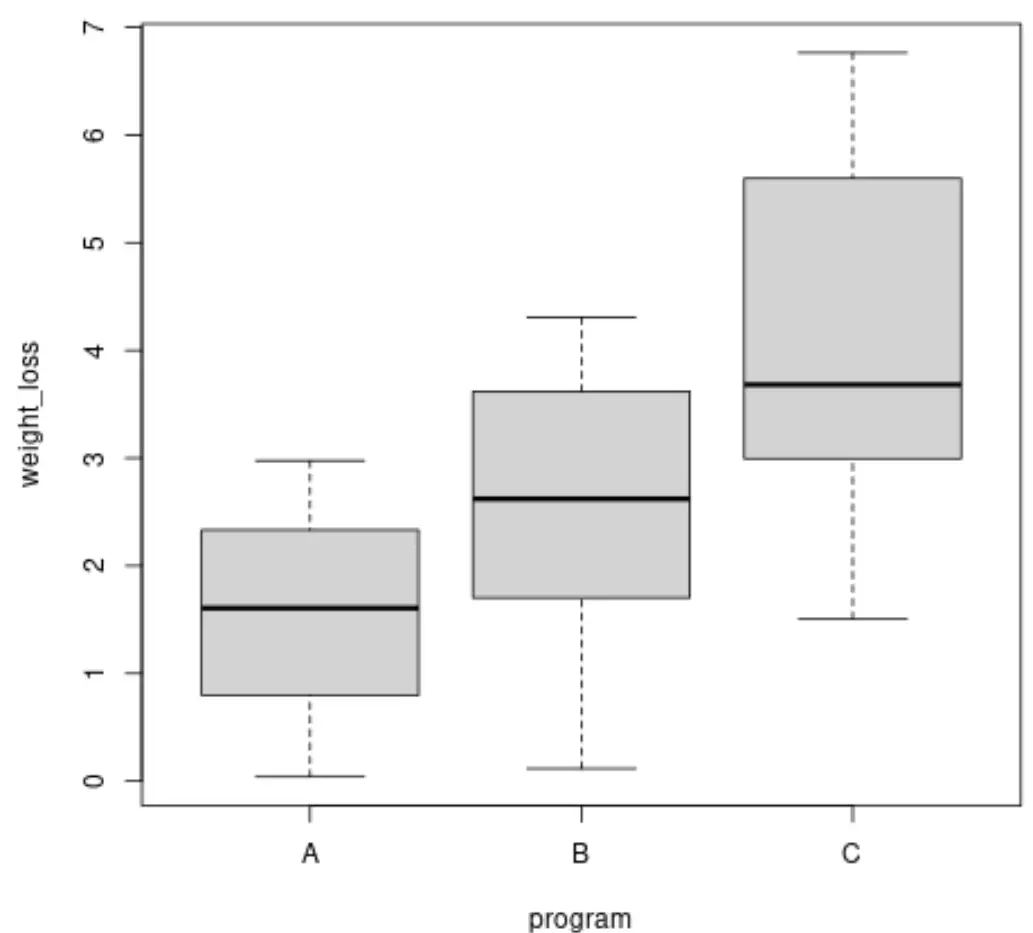
Também podemos calcular a variância da perda de peso em cada grupo:
#load dplyr package library (dplyr) #calculate variance of weight loss by group data %>% group_by (program) %>% summarize (var=var(weight_loss)) # A tibble: 3 x 2 program var 1 A 0.819 2 B 1.53 3 C 2.46
Podemos ver que as variâncias entre os grupos diferem, mas para determinar se essas diferenças são estatisticamente significativas , podemos realizar o teste de Brown-Forsythe.
Etapa 3: execute o teste Brown-Forsythe
Para realizar um teste de Brown-Forsythe em R, podemos usar a função bf.test() do pacote onewaytests :
#load onewaytests package library (onewaytests) #perform Brown-Forsythe test bf.test(weight_loss ~ program, data = data) Brown-Forsythe Test (alpha = 0.05) -------------------------------------------------- ----------- data: weight_loss and program statistic: 30.83304 num df: 2 name df: 74.0272 p.value: 1.816529e-10 Result: Difference is statistically significant. -------------------------------------------------- -----------
O valor p do teste acaba sendo inferior a 0,000 e, como o resultado indica, as diferenças nas variâncias entre os três grupos são estatisticamente significativas.
Próximos passos
Se você não conseguir rejeitar a hipótese nula do teste de Brown-Forsythe, poderá realizar uma ANOVA unidirecional nos dados.
No entanto, se você rejeitar a hipótese nula, isso significa que a suposição de igualdade de variâncias foi violada. Neste caso, você tem duas opções:
1. Faça uma ANOVA unidirecional de qualquer maneira.
Acontece que uma ANOVA unidirecional é realmente robusta para variâncias desiguais, desde que a maior variância não seja maior que 4 vezes a menor variância.
Na etapa 2 do exemplo acima, descobrimos que a menor variância foi 0,819 e a maior variância foi 2,46. Portanto, a razão entre a maior e a menor variância é 2,46 / 0,819 = 3,003 .
Como esse valor é menor que 4, poderíamos simplesmente fazer a ANOVA unidirecional.
2. Faça um teste de Kruskal-Wallis
Se a razão entre a maior variância e a menor variância for maior que 4, pode-se optar por realizar um teste de Kruskal-Wallis . Isto é considerado o equivalente não paramétrico da ANOVA unidirecional.
Você pode encontrar um exemplo passo a passo de um teste de Kruskal-Wallis em R aqui .