Análise discriminante linear em python (passo a passo)
A análise discriminante linear é um método que você pode usar quando possui um conjunto de variáveis preditoras e deseja classificar uma variável de resposta em duas ou mais classes.
Este tutorial fornece um exemplo passo a passo de como realizar análise discriminante linear em Python.
Etapa 1: carregue as bibliotecas necessárias
Primeiro, carregaremos as funções e bibliotecas necessárias para este exemplo:
from sklearn. model_selection import train_test_split
from sklearn. model_selection import RepeatedStratifiedKFold
from sklearn. model_selection import cross_val_score
from sklearn. discriminant_analysis import LinearDiscriminantAnalysis
from sklearn import datasets
import matplotlib. pyplot as plt
import pandas as pd
import numpy as np
Etapa 2: carregar dados
Para este exemplo, usaremos o conjunto de dados iris da biblioteca sklearn. O código a seguir mostra como carregar este conjunto de dados e convertê-lo em um DataFrame do pandas para facilitar o uso:
#load iris dataset iris = datasets. load_iris () #convert dataset to pandas DataFrame df = pd.DataFrame(data = np.c_[iris[' data '], iris[' target ']], columns = iris[' feature_names '] + [' target ']) df[' species '] = pd. Categorical . from_codes (iris.target, iris.target_names) df.columns = [' s_length ', ' s_width ', ' p_length ', ' p_width ', ' target ', ' species '] #view first six rows of DataFrame df. head () s_length s_width p_length p_width target species 0 5.1 3.5 1.4 0.2 0.0 setosa 1 4.9 3.0 1.4 0.2 0.0 setosa 2 4.7 3.2 1.3 0.2 0.0 setosa 3 4.6 3.1 1.5 0.2 0.0 setosa 4 5.0 3.6 1.4 0.2 0.0 setosa #find how many total observations are in dataset len( df.index ) 150
Podemos ver que o conjunto de dados contém 150 observações no total.
Para este exemplo, construiremos um modelo de análise discriminante linear para classificar a qual espécie pertence uma determinada flor.
Usaremos as seguintes variáveis preditoras no modelo:
- Comprimento da sépala
- Largura da sépala
- Comprimento da pétala
- Largura da pétala
E vamos usá-los para prever a variável de resposta Species , que suporta as três classes potenciais a seguir:
- setosa
- versicolor
- Virgínia
Etapa 3: ajuste o modelo LDA
A seguir, ajustaremos o modelo LDA aos nossos dados usando a função LinearDiscriminantAnalsys do sklearn:
#define predictor and response variables X = df[[' s_length ',' s_width ',' p_length ',' p_width ']] y = df[' species '] #Fit the LDA model model = LinearDiscriminantAnalysis() model. fit (x,y)
Etapa 4: use o modelo para fazer previsões
Depois de ajustar o modelo usando nossos dados, podemos avaliar o desempenho do modelo usando validação cruzada estratificada repetida de k-folds.
Para este exemplo usaremos 10 dobras e 3 repetições:
#Define method to evaluate model
cv = RepeatedStratifiedKFold(n_splits= 10 , n_repeats= 3 , random_state= 1 )
#evaluate model
scores = cross_val_score(model, X, y, scoring=' accuracy ', cv=cv, n_jobs=-1)
print( np.mean (scores))
0.9777777777777779
Podemos observar que o modelo alcançou uma precisão média de 97,78% .
Também podemos usar o modelo para prever a qual classe pertence uma nova flor, com base nos valores de entrada:
#define new observation new = [5, 3, 1, .4] #predict which class the new observation belongs to model. predict ([new]) array(['setosa'], dtype='<U10')
Vemos que o modelo prevê que esta nova observação pertence à espécie chamada setosa .
Passo 5: Visualize os resultados
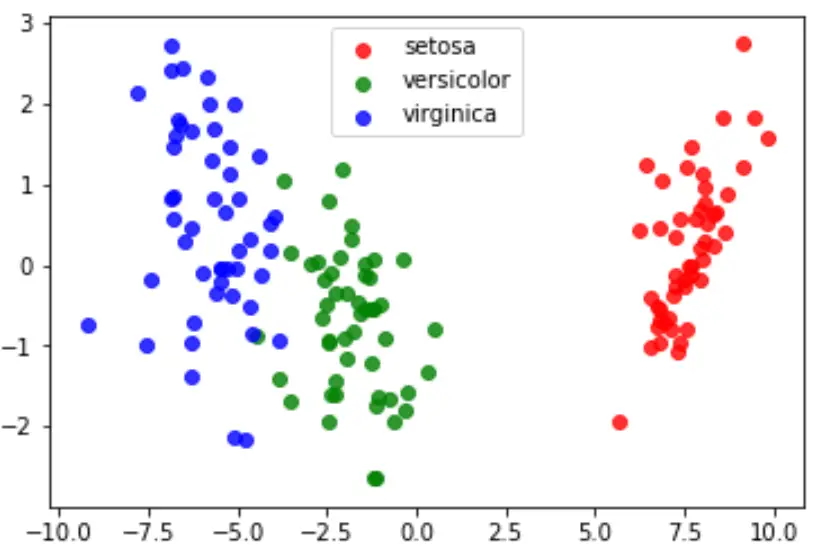
Finalmente, podemos criar um gráfico LDA para visualizar os discriminantes lineares do modelo e visualizar quão bem ele separa as três espécies diferentes em nosso conjunto de dados:
#define data to plot X = iris.data y = iris.target model = LinearDiscriminantAnalysis() data_plot = model. fit (x,y). transform (X) target_names = iris. target_names #create LDA plot plt. figure () colors = [' red ', ' green ', ' blue '] lw = 2 for color, i, target_name in zip(colors, [0, 1, 2], target_names): plt. scatter (data_plot[y == i, 0], data_plot[y == i, 1], alpha=.8, color=color, label=target_name) #add legend to plot plt. legend (loc=' best ', shadow= False , scatterpoints=1) #display LDA plot plt. show ()
Você pode encontrar o código Python completo usado neste tutorial aqui .