Como ajustar árvores de classificação e regressão em r
Quando a relação entre um conjunto de variáveis preditoras e uma variável de resposta é linear, métodos como a regressão linear múltipla podem produzir modelos preditivos precisos.
No entanto, quando a relação entre um conjunto de preditores e uma resposta é mais complexa, os métodos não lineares podem frequentemente produzir modelos mais precisos.
Um desses métodos são as árvores de classificação e regressão (CART), que usam um conjunto de variáveis preditoras para criar árvores de decisão que preveem o valor de uma variável de resposta.
Se a variável resposta for contínua podemos construir árvores de regressão e se a variável resposta for categórica podemos construir árvores de classificação.
Este tutorial explica como criar árvores de regressão e classificação em R.
Exemplo 1: Construindo uma Árvore de Regressão em R
Para este exemplo, usaremos o conjunto de dados Hitters do pacote ISLR , que contém diversas informações sobre 263 jogadores profissionais de beisebol.
Usaremos esse conjunto de dados para construir uma árvore de regressão que usa as variáveis preditoras de home runs e anos jogados para prever o salário de um determinado jogador.
Use as etapas a seguir para criar esta árvore de regressão.
Passo 1: Carregue os pacotes necessários.
Primeiro, carregaremos os pacotes necessários para este exemplo:
library (ISLR) #contains Hitters dataset library (rpart) #for fitting decision trees library (rpart.plot) #for plotting decision trees
Etapa 2: Construa a árvore de regressão inicial.
Primeiro, construiremos uma grande árvore de regressão inicial. Podemos garantir que a árvore é grande usando um valor pequeno para cp , que significa “parâmetro de complexidade”.
Isso significa que realizaremos divisões adicionais na árvore de regressão, desde que o R-quadrado geral do modelo aumente pelo menos o valor especificado por cp.
Em seguida, usaremos a função printcp() para imprimir os resultados do modelo:
#build the initial tree
tree <- rpart(Salary ~ Years + HmRun, data=Hitters, control=rpart. control (cp= .0001 ))
#view results
printcp(tree)
Variables actually used in tree construction:
[1] HmRun Years
Root node error: 53319113/263 = 202734
n=263 (59 observations deleted due to missingness)
CP nsplit rel error xerror xstd
1 0.24674996 0 1.00000 1.00756 0.13890
2 0.10806932 1 0.75325 0.76438 0.12828
3 0.01865610 2 0.64518 0.70295 0.12769
4 0.01761100 3 0.62652 0.70339 0.12337
5 0.01747617 4 0.60891 0.70339 0.12337
6 0.01038188 5 0.59144 0.66629 0.11817
7 0.01038065 6 0.58106 0.65697 0.11687
8 0.00731045 8 0.56029 0.67177 0.11913
9 0.00714883 9 0.55298 0.67881 0.11960
10 0.00708618 10 0.54583 0.68034 0.11988
11 0.00516285 12 0.53166 0.68427 0.11997
12 0.00445345 13 0.52650 0.68994 0.11996
13 0.00406069 14 0.52205 0.68988 0.11940
14 0.00264728 15 0.51799 0.68874 0.11916
15 0.00196586 16 0.51534 0.68638 0.12043
16 0.00016686 17 0.51337 0.67577 0.11635
17 0.00010000 18 0.51321 0.67576 0.11615
n=263 (59 observations deleted due to missingness)
Etapa 3: podar a árvore.
A seguir, podaremos a árvore de regressão para encontrar o valor ideal a ser usado para cp (o parâmetro de complexidade) que leva ao menor erro de teste.
Observe que o valor ideal para cp é aquele que leva ao menor erro x na saída anterior, que representa o erro nas observações dos dados de validação cruzada.
#identify best cp value to use
best <- tree$cptable[which. min (tree$cptable[," xerror "])," CP "]
#produce a pruned tree based on the best cp value
pruned_tree <- prune (tree, cp=best)
#plot the pruned tree
prp(pruned_tree,
faclen= 0 , #use full names for factor labels
extra= 1 , #display number of obs. for each terminal node
roundint= F , #don't round to integers in output
digits= 5 ) #display 5 decimal places in output

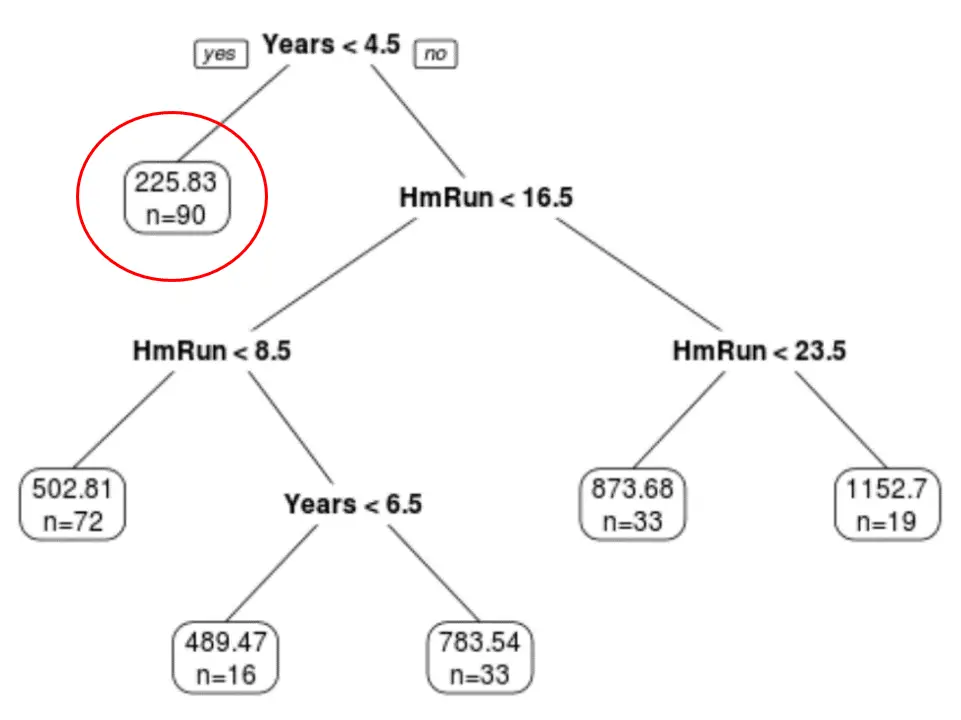
Podemos ver que a árvore podada final possui seis nós terminais. Cada nó folha exibe o salário previsto dos jogadores naquele nó, bem como o número de observações do conjunto de dados original que pertencem a essa categoria.
Por exemplo, podemos ver que no conjunto de dados original, havia 90 jogadores com menos de 4,5 anos de experiência e seu salário médio era de US$ 225,83 mil.
Etapa 4: use a árvore para fazer previsões.
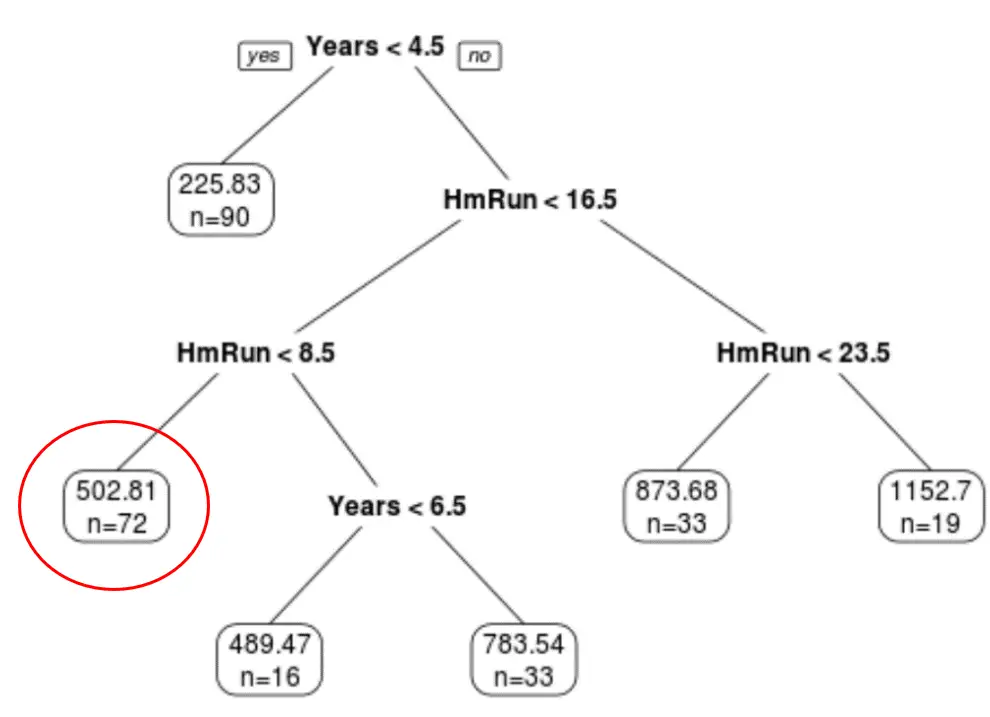
Podemos usar a árvore podada final para prever o salário de um determinado jogador com base em seus anos de experiência e na média de home runs.
Por exemplo, um jogador que tem 7 anos de experiência e 4 home runs em média tem um salário esperado de $ 502,81 mil .
Podemos usar a função prever() em R para confirmar isso:
#define new player
new <- data.frame(Years=7, HmRun=4)
#use pruned tree to predict salary of this player
predict(pruned_tree, newdata=new)
502.8079
Exemplo 2: Construindo uma árvore de classificação em R
Para este exemplo, usaremos o conjunto de dados ptitanic do pacote rpart.plot , que contém diversas informações sobre os passageiros a bordo do Titanic.
Usaremos esse conjunto de dados para criar uma árvore de classificação que usa as variáveis preditoras class , sex e age para prever se um determinado passageiro sobreviveu ou não.
Use as etapas a seguir para criar esta árvore de classificação.
Passo 1: Carregue os pacotes necessários.
Primeiro, carregaremos os pacotes necessários para este exemplo:
library (rpart) #for fitting decision trees library (rpart.plot) #for plotting decision trees
Etapa 2: Construa a árvore de classificação inicial.
Primeiro, construiremos uma grande árvore de classificação inicial. Podemos garantir que a árvore é grande usando um valor pequeno para cp , que significa “parâmetro de complexidade”.
Isso significa que realizaremos outras divisões na árvore de classificação, desde que o ajuste geral do modelo aumente pelo menos o valor especificado por cp.
Em seguida, usaremos a função printcp() para imprimir os resultados do modelo:
#build the initial tree
tree <- rpart(survived~pclass+sex+age, data=ptitanic, control=rpart. control (cp= .0001 ))
#view results
printcp(tree)
Variables actually used in tree construction:
[1] age pclass sex
Root node error: 500/1309 = 0.38197
n=1309
CP nsplit rel error xerror xstd
1 0.4240 0 1.000 1.000 0.035158
2 0.0140 1 0.576 0.576 0.029976
3 0.0095 3 0.548 0.578 0.030013
4 0.0070 7 0.510 0.552 0.029517
5 0.0050 9 0.496 0.528 0.029035
6 0.0025 11 0.486 0.532 0.029117
7 0.0020 19 0.464 0.536 0.029198
8 0.0001 22 0.458 0.528 0.029035
Etapa 3: podar a árvore.
A seguir, podaremos a árvore de regressão para encontrar o valor ideal a ser usado para cp (o parâmetro de complexidade) que leva ao menor erro de teste.
Observe que o valor ideal para cp é aquele que leva ao menor erro x na saída anterior, que representa o erro nas observações dos dados de validação cruzada.
#identify best cp value to use
best <- tree$cptable[which. min (tree$cptable[," xerror "])," CP "]
#produce a pruned tree based on the best cp value
pruned_tree <- prune (tree, cp=best)
#plot the pruned tree
prp(pruned_tree,
faclen= 0 , #use full names for factor labels
extra= 1 , #display number of obs. for each terminal node
roundint= F , #don't round to integers in output
digits= 5 ) #display 5 decimal places in output
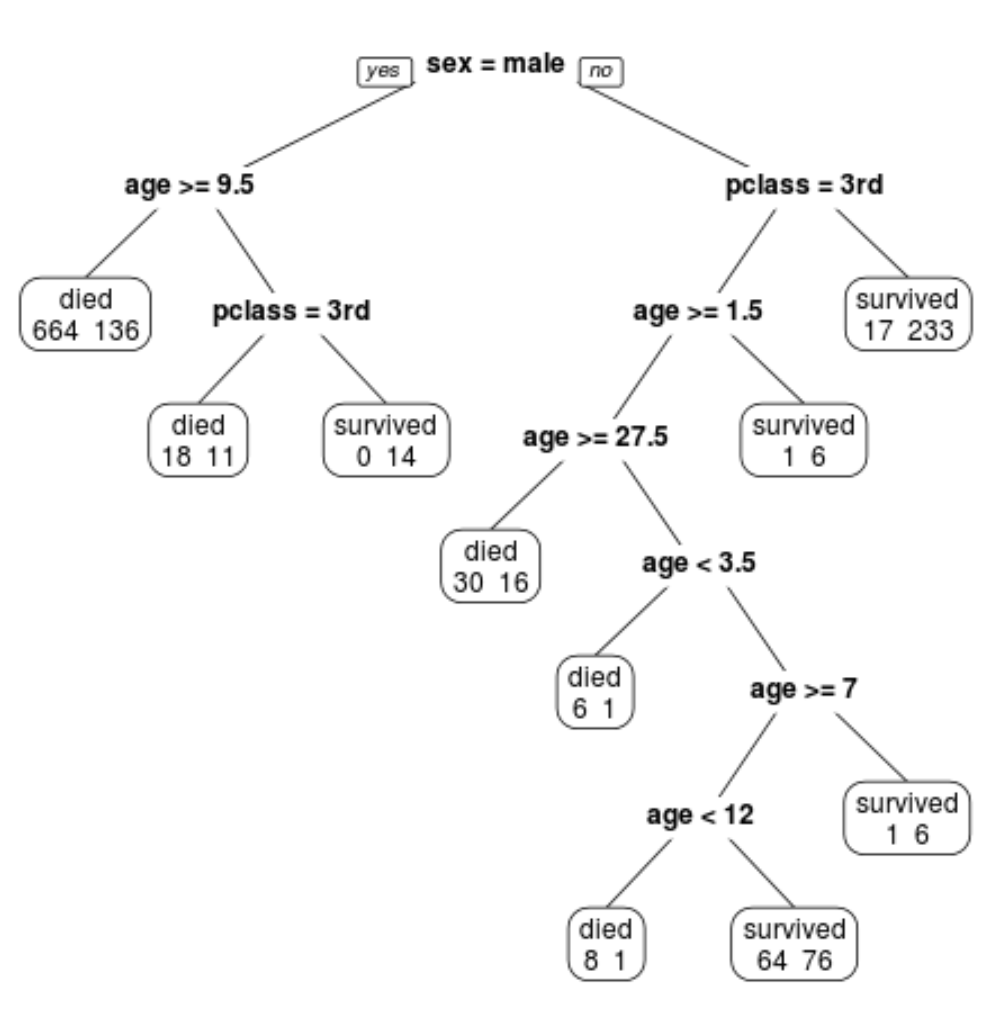
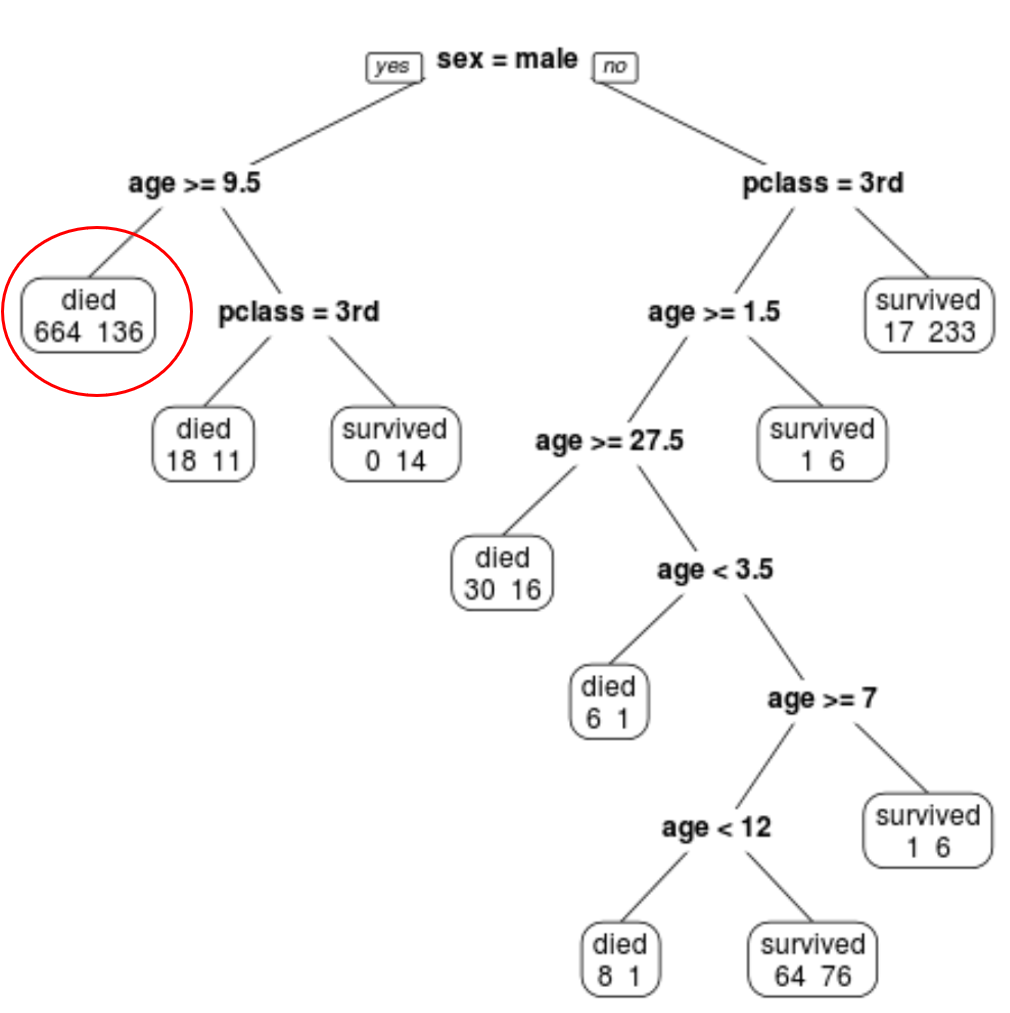
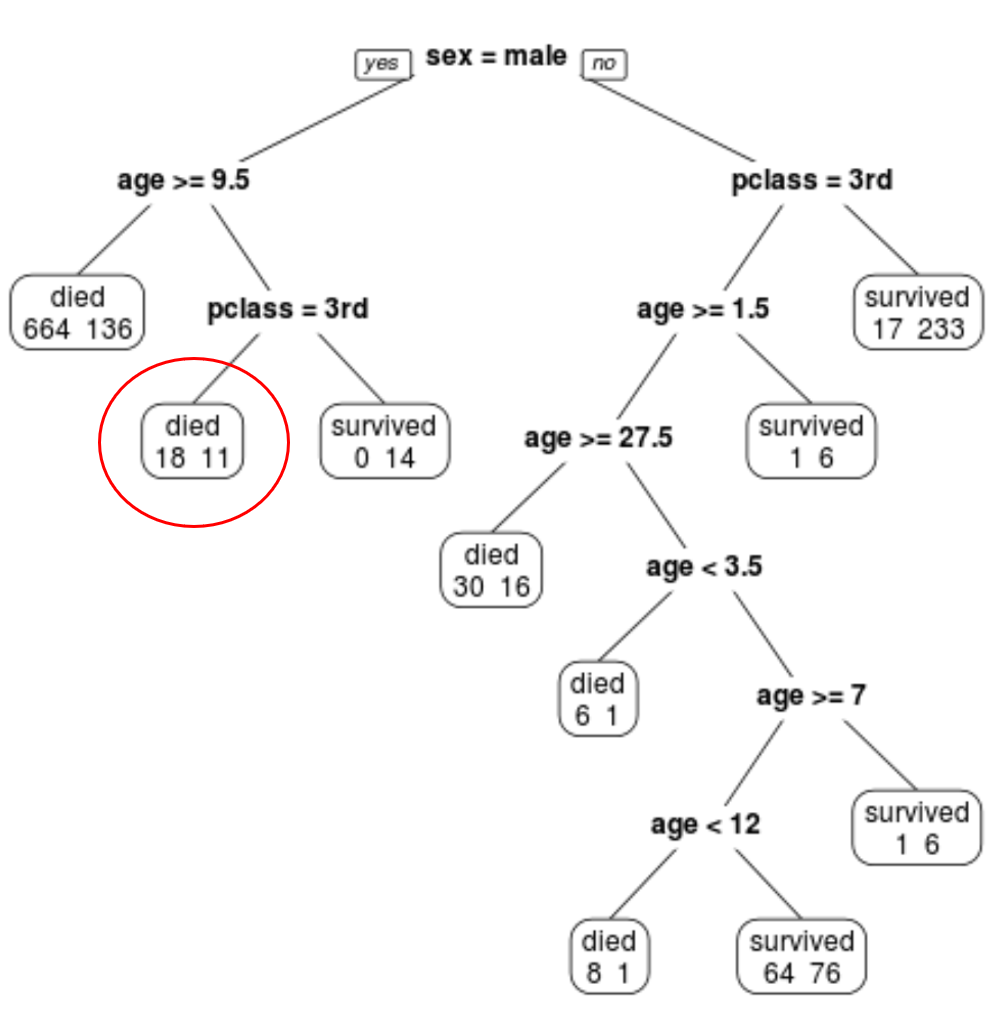
Podemos ver que a árvore podada final possui 10 nós terminais. Cada nó terminal indica o número de passageiros que morreram, bem como o número de sobreviventes.
Por exemplo, no nó mais à esquerda vemos que 664 passageiros morreram e 136 sobreviveram.
Etapa 4: use a árvore para fazer previsões.
Podemos usar a árvore podada final para prever a probabilidade de um determinado passageiro sobreviver com base em sua classe, idade e sexo.
Por exemplo, um passageiro do sexo masculino com 8 anos e na 1ª classe tem uma probabilidade de sobrevivência de 29/11 = 37,9%.
Você pode encontrar o código R completo usado nesses exemplos aqui .