Como inicializar em r (com exemplos)
Bootstrapping é um método que pode ser usado para estimar o erro padrão de qualquer estatística e produzir um intervalo de confiança para a estatística.
O processo básico para inicialização é o seguinte:
- Pegue k amostras replicadas com substituição de um determinado conjunto de dados.
- Para cada amostra, calcule a estatística de interesse.
- Isso fornece k estimativas diferentes para uma determinada estatística, que você pode usar para calcular o erro padrão da estatística e criar um intervalo de confiança para a estatística.
Podemos inicializar em R usando as seguintes funções da biblioteca de bootstrap :
1. Gere amostras de bootstrap.
inicialização(dados, estatísticas, R,…)
Ouro:
- dados: um vetor, matriz ou bloco de dados
- estatística: uma função que produz a(s) estatística(s) a ser(em) iniciada(s)
- A: Número de repetições de bootstrap
2. Gere um intervalo de confiança de bootstrap.
boot.ci(objeto de inicialização, conf, tipo)
Ouro:
- bootobject: Um objeto retornado pela função boot()
- conf: O intervalo de confiança a ser calculado. O valor padrão é 0,95
- type: Tipo de intervalo de confiança a ser calculado. As opções incluem ‘padrão’, ‘básico’, ‘stud’, ‘perc’, ‘bca’ e ‘all’ – o padrão é ‘all’
Os exemplos a seguir mostram como usar essas funções na prática.
Exemplo 1: inicializar uma única estatística
O código a seguir mostra como calcular o erro padrão para o R-quadrado de um modelo de regressão linear simples:
set.seed(0) library (boot) #define function to calculate R-squared rsq_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (summary(fit)$r.square) #return R-squared of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=rsq_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = rsq_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 0.7183433 0.002164339 0.06513426
Dos resultados podemos ver:
- O R-quadrado estimado para este modelo de regressão é 0,7183433 .
- O erro padrão para esta estimativa é 0,06513426 .
Também podemos visualizar rapidamente a distribuição das amostras inicializadas:
plot(reps)
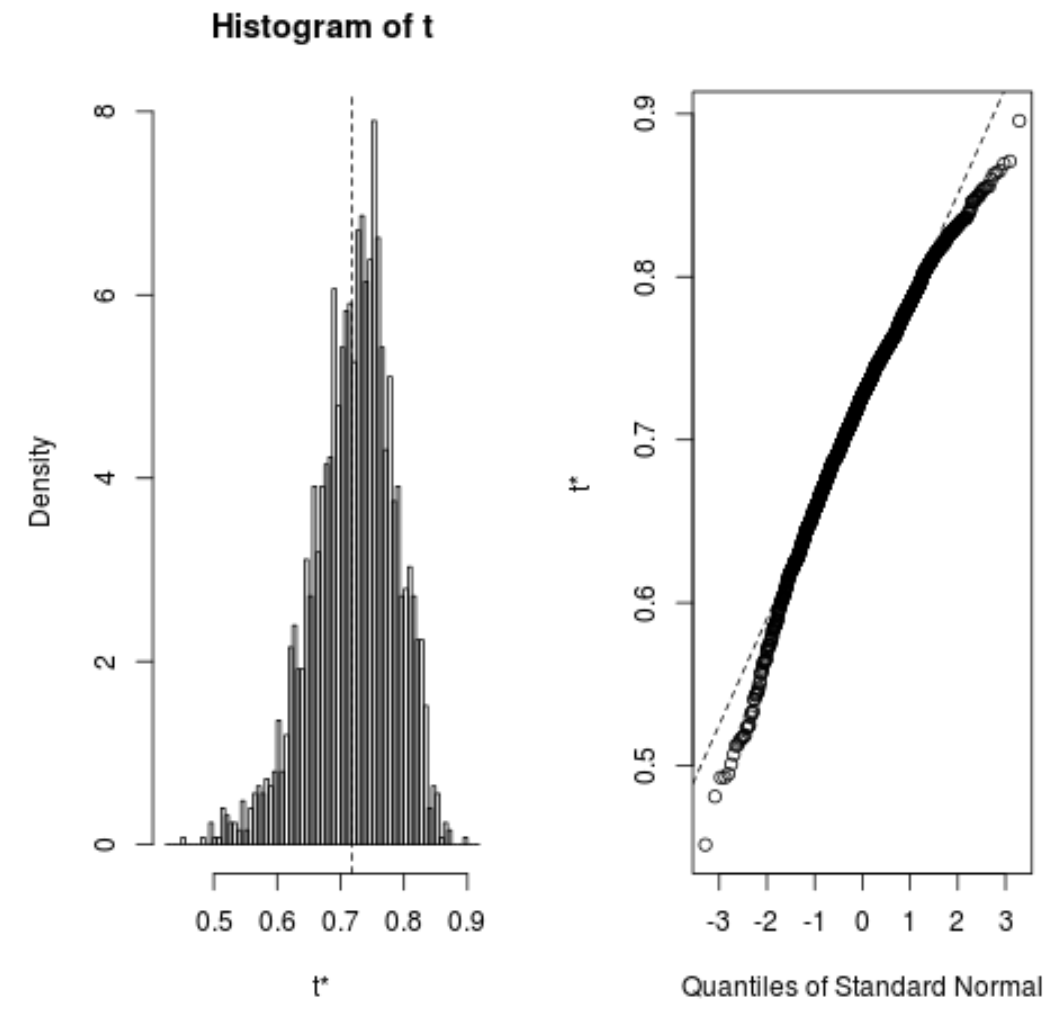
Também podemos usar o código a seguir para calcular o intervalo de confiança de 95% para o R-quadrado estimado do modelo:
#calculate adjusted bootstrap percentile (BCa) interval boot.ci(reps, type=" bca ") CALL: boot.ci(boot.out = reps, type = "bca") Intervals: Level BCa 95% (0.5350, 0.8188) Calculations and Intervals on Original Scale
A partir do resultado, podemos ver que o intervalo de confiança de 95% inicializado para os verdadeiros valores de R ao quadrado é (0,5350, 0,8188).
Exemplo 2: inicializar estatísticas múltiplas
O código a seguir mostra como calcular o erro padrão para cada coeficiente em um modelo de regressão linear múltipla:
set.seed(0) library (boot) #define function to calculate fitted regression coefficients coef_function <- function (formula, data, indices) { d <- data[indices,] #allows boot to select sample fit <- lm(formula, data=d) #fit regression model return (coef(fit)) #return coefficient estimates of model } #perform bootstrapping with 2000 replications reps <- boot(data=mtcars, statistic=coef_function, R=2000, formula=mpg~disp) #view results of boostrapping reps ORDINARY NONPARAMETRIC BOOTSTRAP Call: boot(data = mtcars, statistic = coef_function, R = 2000, formula = mpg ~ available) Bootstrap Statistics: original bias std. error t1* 29.59985476 -5.058601e-02 1.49354577 t2* -0.04121512 6.549384e-05 0.00527082
Dos resultados podemos ver:
- O coeficiente estimado para o intercepto do modelo é 29,59985476 e o erro padrão desta estimativa é 1,49354577 .
- O coeficiente estimado para a variável preditora disp no modelo é -0,04121512 e o erro padrão desta estimativa é 0,00527082 .
Também podemos visualizar rapidamente a distribuição das amostras inicializadas:
plot(reps, index=1) #intercept of model plot(reps, index=2) #disp predictor variable
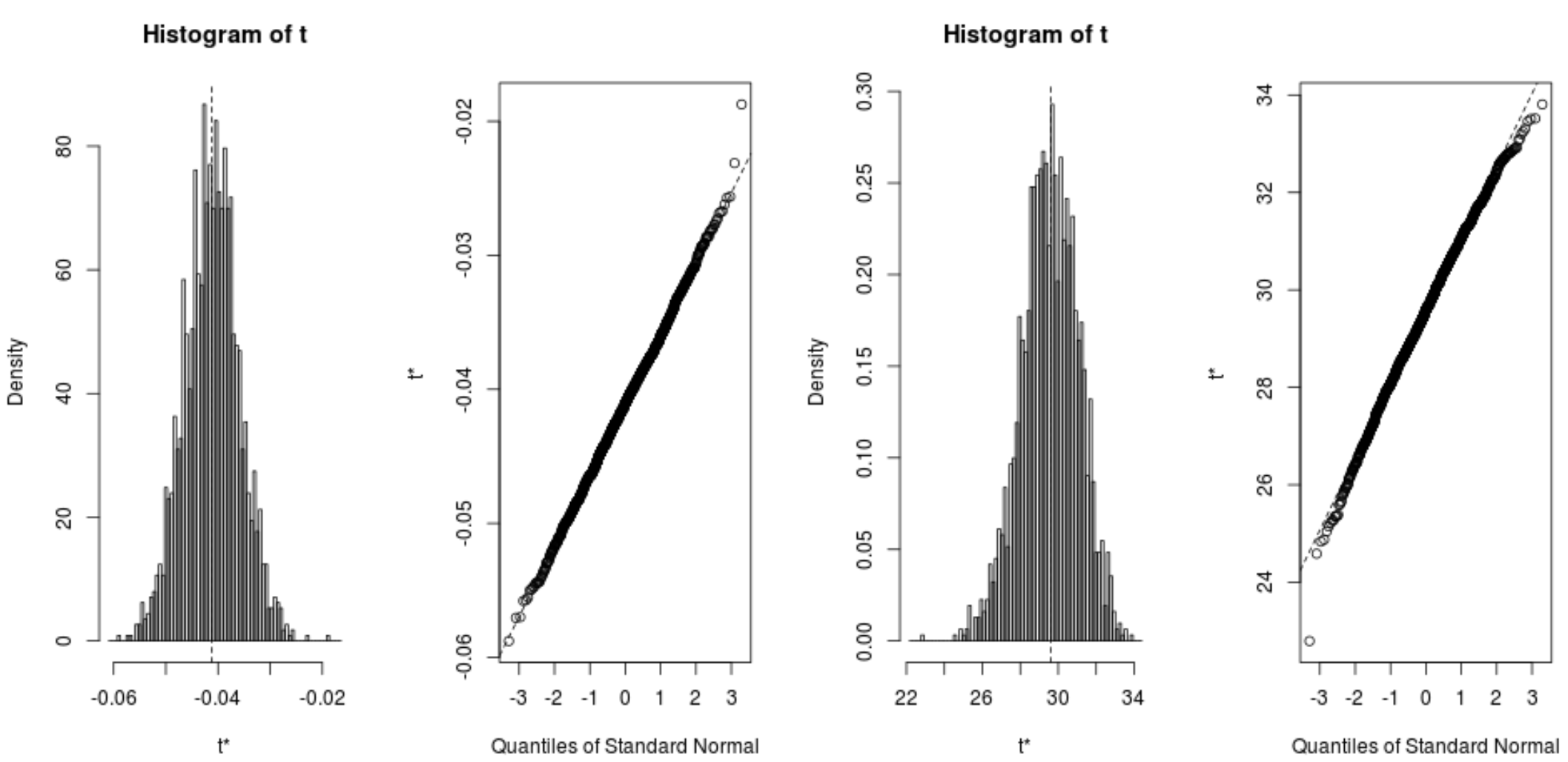
Também podemos usar o seguinte código para calcular os intervalos de confiança de 95% para cada coeficiente:
#calculate adjusted bootstrap percentile (BCa) intervals boot.ci(reps, type=" bca ", index=1) #intercept of model boot.ci(reps, type=" bca ", index=2) #disp predictor variable CALL: boot.ci(boot.out = reps, type = "bca", index = 1) Intervals: Level BCa 95% (26.78, 32.66) Calculations and Intervals on Original Scale BOOTSTRAP CONFIDENCE INTERVAL CALCULATIONS Based on 2000 bootstrap replicates CALL: boot.ci(boot.out = reps, type = "bca", index = 2) Intervals: Level BCa 95% (-0.0520, -0.0312) Calculations and Intervals on Original Scale
A partir dos resultados, podemos ver que os intervalos de confiança de 95% inicializados para os coeficientes do modelo são os seguintes:
- IC para interceptação: (26,78, 32,66)
- CI para disp : (-0,0520, -0,0312)
Recursos adicionais
Como realizar regressão linear simples em R
Como realizar regressão linear múltipla em R
Introdução aos intervalos de confiança