Regressão de componentes principais em r (passo a passo)
Dado um conjunto de p variáveis preditoras e uma variável de resposta, a regressão linear múltipla usa um método conhecido como mínimos quadrados para minimizar a soma residual dos quadrados (RSS):
RSS = Σ(y i – ŷ i ) 2
Ouro:
- Σ : Um símbolo grego que significa soma
- y i : o valor real da resposta para a i-ésima observação
- ŷ i : O valor da resposta prevista com base no modelo de regressão linear múltipla
No entanto, quando as variáveis preditoras são altamente correlacionadas, a multicolinearidade pode se tornar um problema. Isso pode tornar as estimativas dos coeficientes do modelo pouco confiáveis e exibir alta variância.
Uma maneira de evitar esse problema é usar a regressão de componentes principais , que encontra M combinações lineares (chamadas de “componentes principais”) dos p preditores originais e, em seguida, usa mínimos quadrados para ajustar um modelo de regressão linear usando os componentes principais como preditores.
Este tutorial fornece um exemplo passo a passo de como realizar a regressão de componentes principais em R.
Passo 1: Carregue os pacotes necessários
A maneira mais fácil de realizar a regressão de componentes principais em R é usar funções no pacote pls .
#install pls package (if not already installed) install.packages(" pls ") load pls package library(pls)
Passo 2: Ajustar o modelo PCR
Para este exemplo, usaremos o conjunto de dados R integrado chamado mtcars , que contém dados sobre diferentes tipos de carros:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
Para este exemplo, ajustaremos um modelo de regressão de componentes principais (PCR) usando hp como variável de resposta e as seguintes variáveis como variáveis preditoras:
- mpg
- mostrar
- merda
- peso
- qsec
O código a seguir mostra como ajustar o modelo PCR a esses dados. Observe os seguintes argumentos:
- scale=TRUE : diz a R que cada uma das variáveis preditoras deve ser dimensionada para ter uma média de 0 e um desvio padrão de 1. Isso garante que nenhuma variável preditora tenha muita influência no modelo se for medida em unidades diferentes. .
- validação=”CV” : diz ao R para usar a validação cruzada k-fold para avaliar o desempenho do modelo. Observe que isso usa k=10 dobras por padrão. Observe também que você pode especificar “LOOCV” para realizar a validação cruzada Leave-One-Out .
#make this example reproducible set.seed(1) #fit PCR model model <- pcr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
Etapa 3: Escolha o número de componentes principais
Depois de ajustar o modelo, precisamos determinar quantos componentes principais valem a pena manter.
Para fazer isso, basta observar a raiz do erro quadrático médio do teste (teste RMSE) calculado pela validação k-cruzada:
#view summary of model fitting
summary(model)
Data:
Y dimension: 32 1
Fit method: svdpc
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comp 2 comps 3 comps 4 comps 5 comps
CV 69.66 44.56 35.64 35.83 36.23 36.67
adjCV 69.66 44.44 35.27 35.43 35.80 36.20
TRAINING: % variance explained
1 comp 2 comps 3 comps 4 comps 5 comps
X 69.83 89.35 95.88 98.96 100.00
hp 62.38 81.31 81.96 81.98 82.03
Existem duas tabelas interessantes no resultado:
1. VALIDAÇÃO: RMSEP
Esta tabela nos mostra o teste RMSE calculado pela validação cruzada k-fold. Podemos ver o seguinte:
- Se usarmos apenas o termo original no modelo, o RMSE do teste será 69,66 .
- Se somarmos o primeiro componente principal, o teste RMSE cai para 44,56.
- Se adicionarmos o segundo componente principal, o teste RMSE cai para 35,64.
Podemos ver que a adição de componentes principais adicionais resulta, na verdade, em um aumento no RMSE do teste. Assim, parece que seria ótimo usar apenas dois componentes principais no modelo final.
2. TREINAMENTO: % de variância explicada
Esta tabela nos informa a porcentagem de variância na variável resposta explicada pelos componentes principais. Podemos ver o seguinte:
- Utilizando apenas a primeira componente principal, podemos explicar 69,83% da variação da variável resposta.
- Adicionando o segundo componente principal, podemos explicar 89,35% da variação da variável resposta.
Observe que ainda seremos capazes de explicar mais variância usando mais componentes principais, mas podemos ver que adicionar mais de dois componentes principais não aumenta muito a porcentagem de variância explicada.
Também podemos visualizar o teste RMSE (junto com o teste MSE e R-quadrado) como uma função do número de componentes principais usando a função validaçãoplot() .
#visualize cross-validation plots
validationplot(model)
validationplot(model, val.type="MSEP")
validationplot(model, val.type="R2")
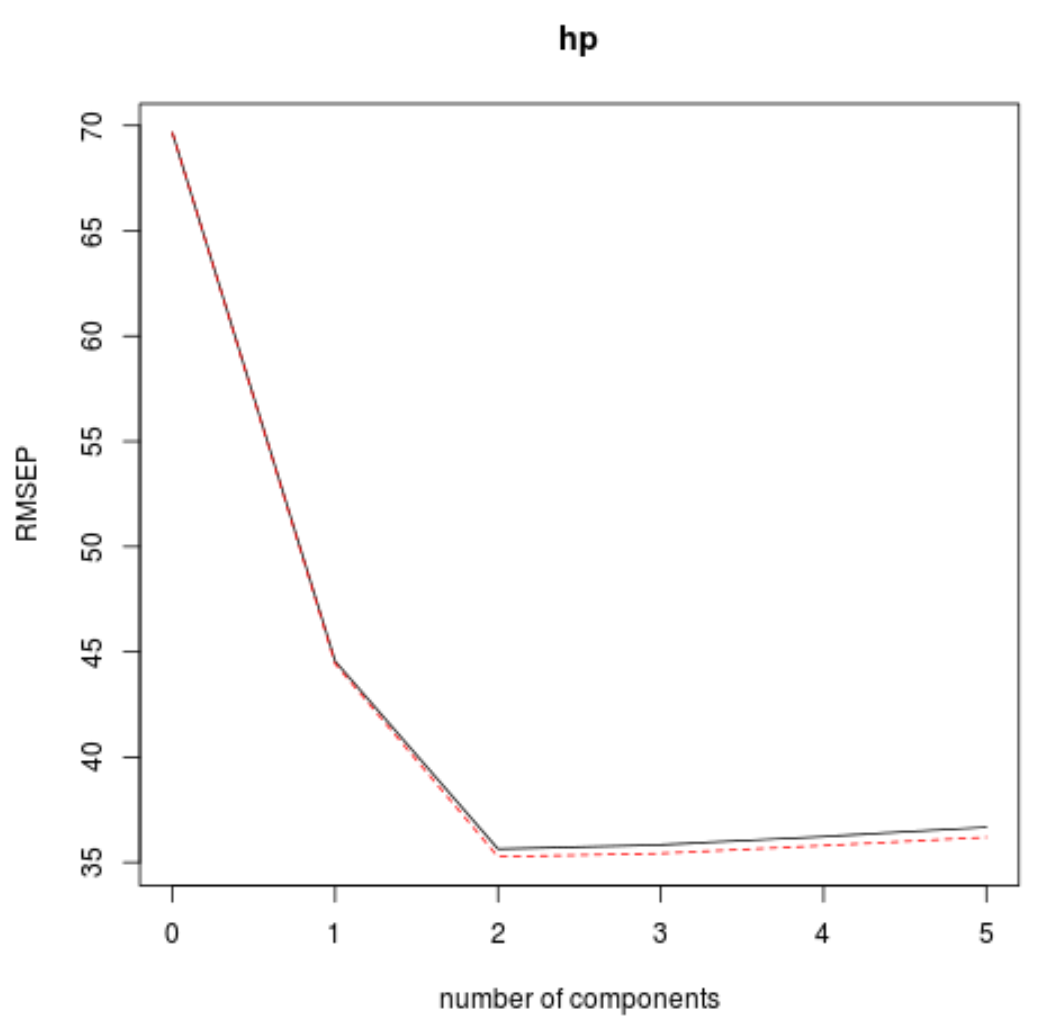
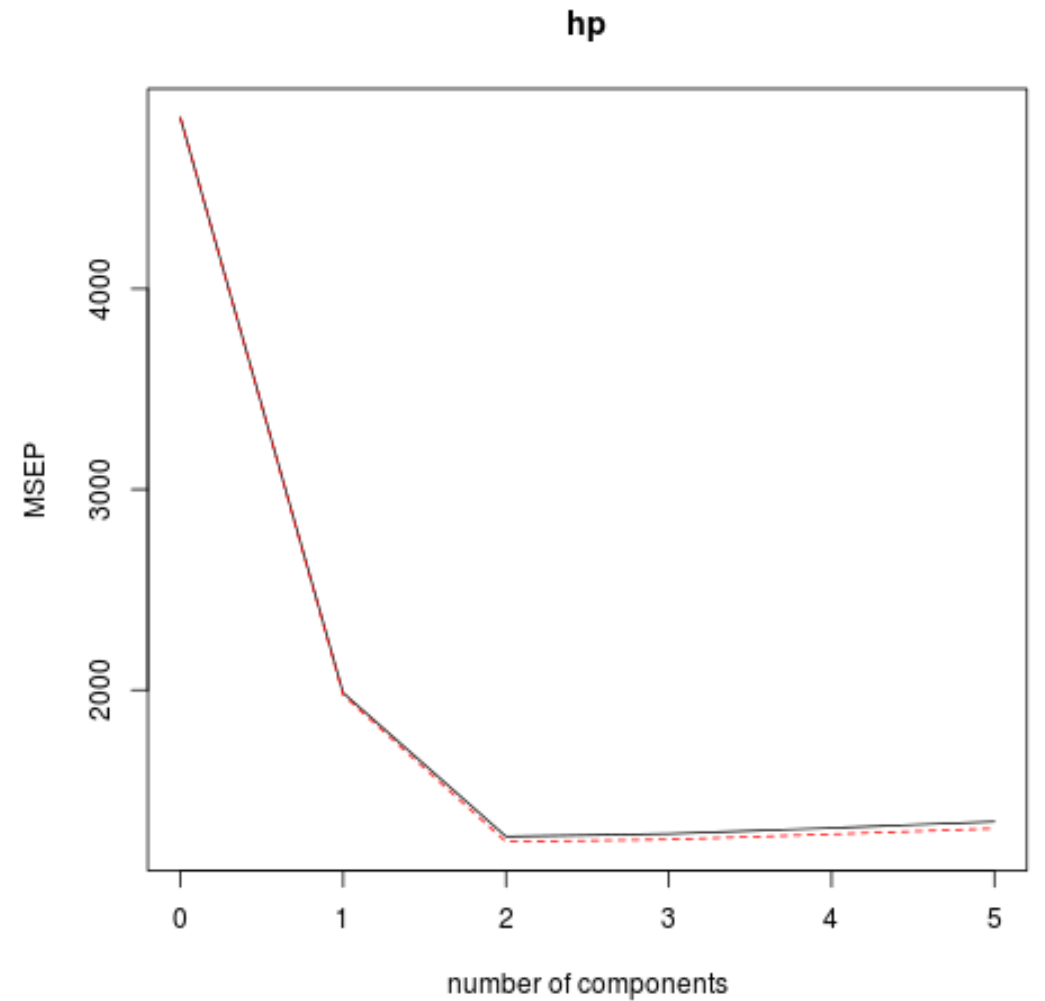
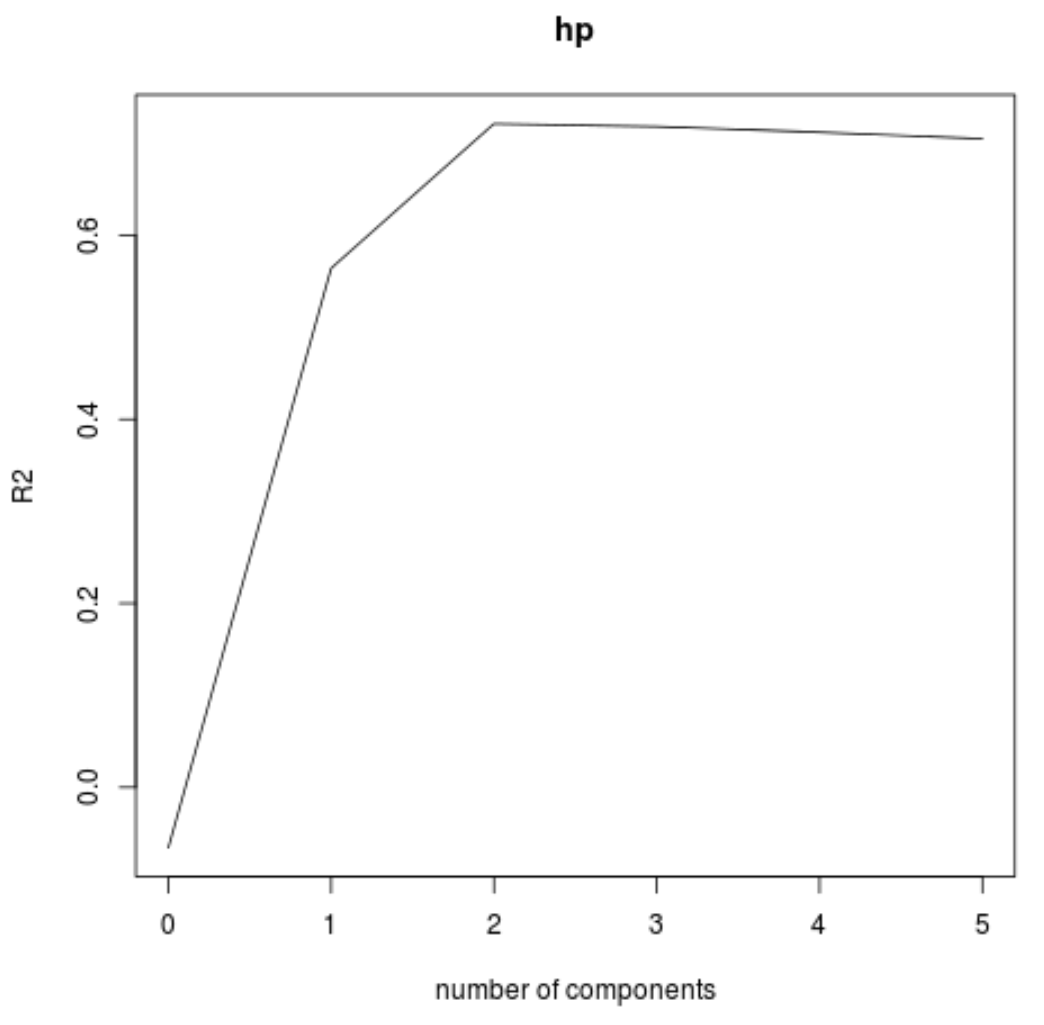
Em cada gráfico, podemos ver que o ajuste do modelo melhora ao adicionar dois componentes principais, mas tende a piorar quando adicionamos mais componentes principais.
Assim, o modelo ótimo inclui apenas os dois primeiros componentes principais.
Etapa 4: use o modelo final para fazer previsões
Podemos usar o modelo final de PCR de dois componentes principais para fazer previsões sobre novas observações.
O código a seguir mostra como dividir o conjunto de dados original em um conjunto de treinamento e teste e usar o modelo PCR com dois componentes principais para fazer previsões no conjunto de teste.
#define training and testing sets train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")] y_test <- mtcars[26: nrow (mtcars), c("hp")] test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")] #use model to make predictions on a test set model <- pcr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ") pcr_pred <- predict(model, test, ncomp= 2 ) #calculate RMSE sqrt ( mean ((pcr_pred - y_test)^2)) [1] 56.86549
Vemos que o RMSE do teste é 56,86549 . Este é o desvio médio entre o valor de HP previsto e o valor de HP observado para as observações do conjunto de teste.
O uso completo do código R neste exemplo pode ser encontrado aqui .