Como calcular resíduos padronizados em r
Um resíduo é a diferença entre um valor observado e um valor previsto em um modelo de regressão .
É calculado da seguinte forma:
Residual = Valor observado – Valor previsto
Se plotarmos os valores observados e sobrepormos a linha de regressão ajustada, os resíduos de cada observação seriam a distância vertical entre a observação e a linha de regressão:
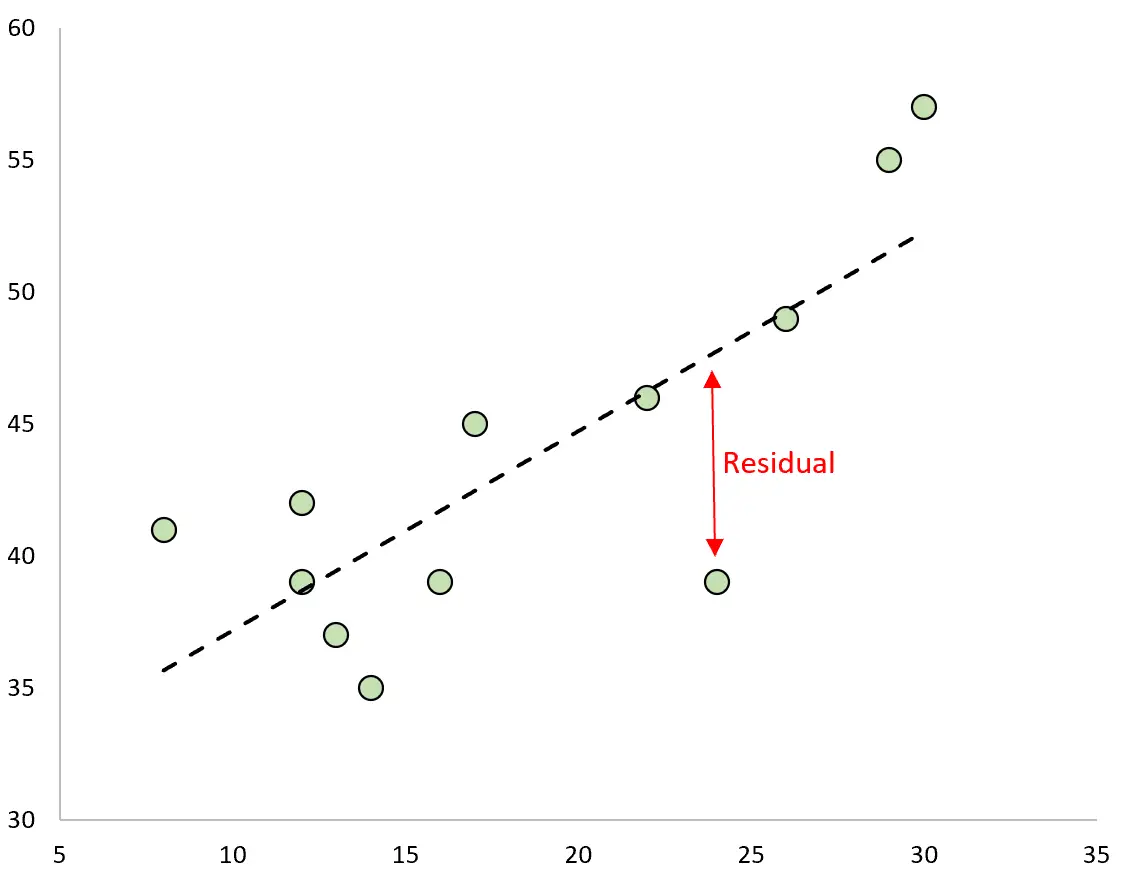
Um tipo de resíduo que costumamos usar para identificar valores discrepantes em um modelo de regressão é chamado de resíduo padronizado .
É calculado da seguinte forma:
r eu = e eu / s(e eu ) = e eu / RSE√ 1-h ii
Ouro:
- e i : O i- ésimo resíduo
- RSE: erro padrão residual do modelo
- h ii : O surgimento da i-ésima observação
Na prática, muitas vezes consideramos qualquer resíduo padronizado cujo valor absoluto seja superior a 3 como um valor atípico.
Este tutorial fornece um exemplo passo a passo de como calcular resíduos padronizados em R.
Passo 1: Insira os dados
Primeiro, criaremos um pequeno conjunto de dados para trabalhar em R:
#create data data <- data.frame(x=c(8, 12, 12, 13, 14, 16, 17, 22, 24, 26, 29, 30), y=c(41, 42, 39, 37, 35, 39, 45, 46, 39, 49, 55, 57)) #viewdata data xy 1 8 41 2 12 42 3 12 39 4 13 37 5 14 35 6 16 39 7 17 45 8 22 46 9 24 39 10 26 49 11 29 55 12 30 57
Passo 2: Ajustar o modelo de regressão
A seguir, usaremos a função lm() para ajustar um modelo de regressão linear simples :
#fit model model <- lm(y ~ x, data=data) #view model summary summary(model) Call: lm(formula = y ~ x, data = data) Residuals: Min 1Q Median 3Q Max -8.7578 -2.5161 0.0292 3.3457 5.3268 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 29.6309 3.6189 8.188 9.6e-06 *** x 0.7553 0.1821 4.148 0.00199 ** --- Significant. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 Residual standard error: 4.442 on 10 degrees of freedom Multiple R-squared: 0.6324, Adjusted R-squared: 0.5956 F-statistic: 17.2 on 1 and 10 DF, p-value: 0.001988
Passo 3: Calcular resíduos padronizados
A seguir, usaremos a função integrada rstandard() para calcular os resíduos padronizados do modelo:
#calculate the standardized residuals standard_res <- rstandard(model) #view the standardized residuals standard_res 1 2 3 4 5 6 1.40517322 0.81017562 0.07491009 -0.59323342 -1.24820530 -0.64248883 7 8 9 10 11 12 0.59610905 -0.05876884 -2.11711982 -0.06655600 0.91057211 1.26973888
Podemos adicionar os resíduos padronizados ao quadro de dados original, se desejarmos:
#column bind standardized residuals back to original data frame final_data <- cbind(data, standard_res) #view data frame xy standard_res 1 8 41 1.40517322 2 12 42 0.81017562 3 12 39 0.07491009 4 13 37 -0.59323342 5 14 35 -1.24820530 6 16 39 -0.64248883 7 17 45 0.59610905 8 22 46 -0.05876884 9 24 39 -2.11711982 10 26 49 -0.06655600 11 29 55 0.91057211 12 30 57 1.26973888
Podemos então classificar cada observação da maior para a menor com base em seu resíduo padronizado para ter uma ideia de quais observações estão mais próximas dos valores discrepantes:
#sort standardized residuals descending
final_data[ order (-standard_res),]
xy standard_res
1 8 41 1.40517322
12 30 57 1.26973888
11 29 55 0.91057211
2 12 42 0.81017562
7 17 45 0.59610905
3 12 39 0.07491009
8 22 46 -0.05876884
10 26 49 -0.06655600
4 13 37 -0.59323342
6 16 39 -0.64248883
5 14 35 -1.24820530
9 24 39 -2.11711982
A partir dos resultados, podemos ver que nenhum dos resíduos padronizados excede o valor absoluto de 3. Assim, nenhuma das observações parece ser discrepante.
Passo 4: Visualize os resíduos padronizados
Finalmente, podemos criar um gráfico de dispersão para visualizar os valores da variável preditora em relação aos resíduos padronizados:
#plot predictor variable vs. standardized residuals
plot(final_data$x, standard_res, ylab=' Standardized Residuals ', xlab=' x ')
#add horizontal line at 0
abline(0, 0)
Recursos adicionais
O que são resíduos?
O que são resíduos padronizados?
Introdução à regressão linear múltipla