Как выполнить тест на отсутствие соответствия в r (шаг за шагом)
Тест на отсутствие соответствия используется, чтобы определить, обеспечивает ли полная регрессионная модель значительно лучшее соответствие набору данных, чем сокращенная версия модели.
Например, предположим, что мы хотим использовать количество изученных часов , чтобы предсказать результаты экзаменов для студентов определенного колледжа. Мы можем решить адаптировать следующие две регрессионные модели:
Полная модель: оценка = β 0 + B 1 (часы) + B 2 (часы) 2
Сокращенная модель: оценка = β 0 + B 1 (часы)
В следующем пошаговом примере показано, как выполнить тест на отсутствие соответствия в R, чтобы определить, обеспечивает ли полная модель значительно лучшее соответствие, чем уменьшенная модель.
Шаг 1. Создайте и визуализируйте набор данных
Сначала мы воспользуемся следующим кодом, чтобы создать набор данных, содержащий количество учебных часов и баллы, полученные на экзаменах для 50 студентов:
#make this example reproducible set. seeds (1) #create dataset df <- data. frame (hours = runif (50, 5, 15), score=50) df$score = df$score + df$hours^3/150 + df$hours* runif (50, 1, 2) #view first six rows of data head(df) hours score 1 7.655087 64.30191 2 8.721239 70.65430 3 10.728534 73.66114 4 14.082078 86.14630 5 7.016819 59.81595 6 13.983897 83.60510
Далее мы создадим диаграмму рассеяния, чтобы визуализировать взаимосвязь между часами и баллами:
#load ggplot2 visualization package library (ggplot2) #create scatterplot ggplot(df, aes (x=hours, y=score)) + geom_point()
Шаг 2. Подберите к набору данных две разные модели.
Далее мы адаптируем к набору данных две разные модели регрессии:
#fit full model full <- lm(score ~ poly (hours,2), data=df) #fit reduced model reduced <- lm(score ~ hours, data=df)
Шаг 3. Проведите тест на отсутствие прилегания.
Далее мы воспользуемся командой anova() , чтобы выполнить тест на отсутствие соответствия между двумя моделями:
#lack of fit test
anova(full, reduced)
Analysis of Variance Table
Model 1: score ~ poly(hours, 2)
Model 2: score ~ hours
Res.Df RSS Df Sum of Sq F Pr(>F)
1 47 368.48
2 48 451.22 -1 -82.744 10.554 0.002144 **
---
Significant. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Статистика F-теста равна 10,554 , а соответствующее значение p — 0,002144 . Поскольку это значение p меньше 0,05, мы можем отклонить нулевую гипотезу теста и сделать вывод, что полная модель статистически значительно лучше подходит, чем сокращенная модель.
Шаг 4. Визуализируйте окончательную модель
Наконец, мы можем визуализировать окончательную модель (полную модель) на основе исходного набора данных:
ggplot(df, aes (x=hours, y=score)) +
geom_point() +
stat_smooth(method=' lm ', formula = y ~ poly (x,2), size = 1) +
xlab(' Hours Studied ') +
ylab(' Score ')
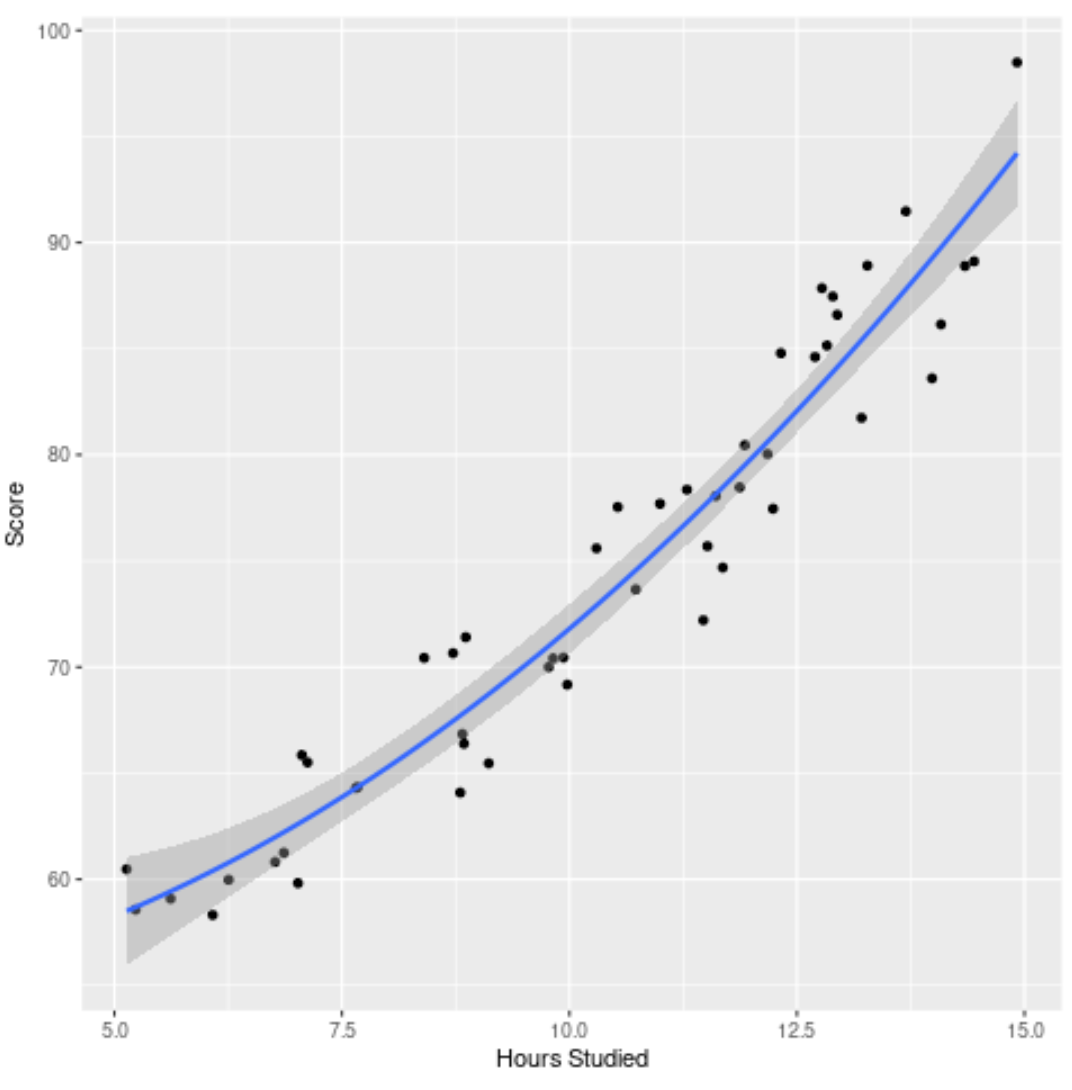
Мы видим, что кривая модели довольно хорошо соответствует данным.
Дополнительные ресурсы
Как выполнить простую линейную регрессию в R
Как выполнить множественную линейную регрессию в R
Как выполнить полиномиальную регрессию в R