Частичные наименьшие квадраты в r (шаг за шагом)
Одна из наиболее распространенных проблем, с которыми вы можете столкнуться в машинном обучении, — это мультиколлинеарность . Это происходит, когда две или более переменных-предикторов в наборе данных сильно коррелируют.
Когда это происходит, модель может хорошо соответствовать набору обучающих данных, но может плохо работать с новым набором данных, которого она никогда не видела, поскольку он не соответствует набору обучающих данных. Обучающий набор.
Один из способов обойти эту проблему — использовать метод частичных наименьших квадратов , который работает следующим образом:
- Стандартизируйте переменные-предикторы и отклики.
- Вычислите M линейных комбинаций (называемых «компонентами PLS») исходных переменных-предсказателей p , которые объясняют значительную величину изменений как в переменной ответа, так и в переменных-предикторах.
- Используйте метод наименьших квадратов, чтобы подобрать модель линейной регрессии с использованием компонентов PLS в качестве предикторов.
- Используйте k-кратную перекрестную проверку , чтобы найти оптимальное количество компонентов PLS для сохранения в модели.
В этом руководстве представлен пошаговый пример выполнения метода частичных наименьших квадратов в R.
Шаг 1. Загрузите необходимые пакеты
Самый простой способ выполнить частичный наименьшие квадраты в R — использовать функции из пакета pls .
#install pls package (if not already installed) install.packages(" pls ") load pls package library(pls)
Шаг 2. Подберите модель частичных наименьших квадратов.
В этом примере мы будем использовать встроенный набор данных R под названием mtcars , который содержит данные о различных типах автомобилей:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
В этом примере мы подберем модель частичных наименьших квадратов (PLS), используя hp в качестве переменной ответа и следующие переменные в качестве переменных-предикторов:
- миль на галлон
- отображать
- дерьмо
- масса
- qsec
Следующий код показывает, как подогнать модель PLS к этим данным. Обратите внимание на следующие аргументы:
- Scale=TRUE : это сообщает R, что каждая из переменных в наборе данных должна быть масштабирована так, чтобы иметь среднее значение 0 и стандартное отклонение 1. Это гарантирует, что ни одна переменная-предиктор не будет иметь слишком большого влияния на модель, если она измеряется в разных единицах.
- validation=”CV” : указывает R использовать k-кратную перекрестную проверку для оценки производительности модели. Обратите внимание, что по умолчанию используется k=10 складок. Также обратите внимание, что вместо этого вы можете указать «LOOCV», чтобы выполнить перекрестную проверку Leave-One-Out .
#make this example reproducible set.seed(1) #fit PCR model model <- plsr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
Шаг 3. Выберите количество компонентов PLS.
После того, как мы подогнали модель, нам нужно определить, сколько компонентов PLS следует сохранить.
Для этого просто посмотрите на среднеквадратическую ошибку теста (тест RMSE), рассчитанную с помощью проверки k-cross:
#view summary of model fitting
summary(model)
Data:
Y dimension: 32 1
Fit method: kernelpls
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comp 2 comps 3 comps 4 comps 5 comps
CV 69.66 40.57 35.48 36.22 36.74 36.67
adjCV 69.66 40.41 35.12 35.80 36.27 36.20
TRAINING: % variance explained
1 comp 2 comps 3 comps 4 comps 5 comps
X 68.66 89.27 95.82 97.94 100.00
hp 71.84 81.74 82.00 82.02 82.03
В результате получились две интересные таблицы:
1. ВАЛИДАЦИЯ: RMSEP
В этой таблице показан тест RMSE, рассчитанный путем перекрестной проверки в k-кратном размере. Мы можем увидеть следующее:
- Если мы используем только исходный термин в модели, RMSE теста составит 69,66 .
- Если мы добавим первый компонент PLS, тест RMSE упадет до 40,57.
- Если мы добавим второй компонент PLS, тест RMSE упадет до 35,48.
Мы видим, что добавление дополнительных компонентов PLS фактически приводит к увеличению RMSE теста. Таким образом, оказывается, что оптимально было бы использовать в итоговой модели только два компонента PLS.
2. ОБУЧЕНИЕ: объяснение % дисперсии
В этой таблице указан процент отклонения переменной ответа, объясняемый компонентами PLS. Мы можем увидеть следующее:
- Используя только первый компонент PLS, мы можем объяснить 68,66% вариаций переменной ответа.
- Добавив второй компонент PLS, мы можем объяснить 89,27% вариации переменной ответа.
Обратите внимание, что мы по-прежнему сможем объяснить большую дисперсию, используя больше компонентов PLS, но мы видим, что добавление более двух компонентов PLS на самом деле не увеличивает процент объясняемой дисперсии.
Мы также можем визуализировать тест RMSE (наряду с тестом MSE и R-квадрат) как функцию количества компонентов PLS, используя функцию validationplot() .
#visualize cross-validation plots validationplot(model) validationplot(model, val.type=" MSEP ") validationplot(model, val.type=" R2 ")
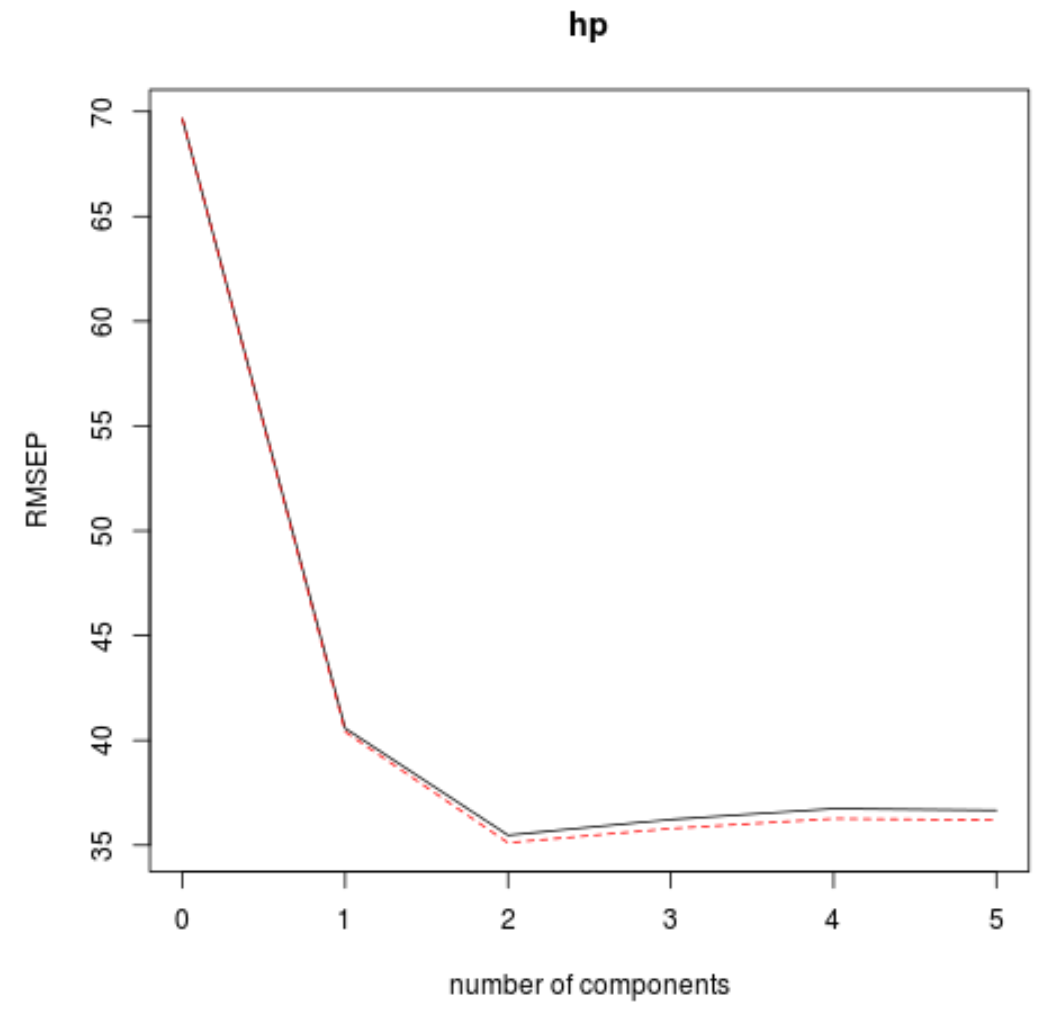
На каждом графике мы видим, что соответствие модели улучшается при добавлении двух компонентов PLS, но имеет тенденцию ухудшаться при добавлении большего количества компонентов PLS.
Таким образом, оптимальная модель включает только первые два компонента PLS.
Шаг 4. Используйте окончательную модель для прогнозирования
Мы можем использовать окончательную модель с двумя компонентами PLS, чтобы прогнозировать новые наблюдения.
В следующем коде показано, как разделить исходный набор данных на обучающий и тестовый набор и использовать окончательную модель с двумя компонентами PLS для прогнозирования тестового набора.
#define training and testing sets train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")] y_test <- mtcars[26: nrow (mtcars), c("hp")] test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")] #use model to make predictions on a test set model <- plsr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ") pcr_pred <- predict(model, test, ncomp= 2 ) #calculate RMSE sqrt ( mean ((pcr_pred - y_test)^2)) [1] 54.89609
Мы видим, что RMSE теста оказывается равным 54,89609 . Это среднее отклонение между прогнозируемым значением hp и наблюдаемым значением hp для наблюдений тестового набора.
Обратите внимание, что эквивалентная регрессионная модель главных компонентов с двумя главными компонентами дала тестовое RMSE 56,86549 . Таким образом, модель PLS немного превзошла модель PCR для этого набора данных.
Полное использование кода R в этом примере можно найти здесь .