К-медоиды в r: пошаговый пример
Кластеризация — это метод машинного обучения, который пытается найти группы или кластеры наблюдений в наборе данных.
Цель состоит в том, чтобы найти кластеры, в которых наблюдения внутри каждого кластера очень похожи друг на друга, а наблюдения в разных кластерах сильно отличаются друг от друга.
Кластеризация — это форма обучения без учителя , поскольку мы просто пытаемся найти структуру в наборе данных, а не предсказать значение переменной ответа .
Кластеризация часто используется в маркетинге, когда предприятия имеют доступ к такой информации, как:
- Семейный доход
- Размер семьи
- Глава семьи Профессия
- Расстояние до ближайшего населенного пункта
Когда эта информация доступна, кластеризацию можно использовать для выявления схожих домохозяйств, которые с большей вероятностью будут покупать определенные продукты или лучше реагировать на определенный тип рекламы.
Одна из наиболее распространенных форм кластеризации известна как кластеризация k-средних .
К сожалению, на этот метод могут влиять выбросы, поэтому часто используемой альтернативой является кластеризация k-medoids .
Что такое кластеризация K-Medoids?
Кластеризация K-медоидов — это метод, при котором мы помещаем каждое наблюдение из набора данных в один из K- кластеров.
Конечная цель состоит в том, чтобы иметь K кластеров, в которых наблюдения внутри каждого кластера очень похожи друг на друга, в то время как наблюдения в разных кластерах сильно отличаются друг от друга.
На практике мы используем следующие шаги для выполнения кластеризации K-средних:
1. Выберите значение К.
- Во-первых, нам нужно решить, сколько кластеров мы хотим идентифицировать в данных. Часто нам просто нужно протестировать несколько разных значений K и проанализировать результаты, чтобы увидеть, какое количество кластеров кажется наиболее подходящим для конкретной задачи.
2. Случайным образом отнесите каждое наблюдение к начальному кластеру от 1 до K.
3. Выполняйте следующую процедуру, пока назначения кластера не перестанут меняться.
- Для каждого из K кластеров вычислите центр тяжести кластера. Это вектор p- медиан признаков для наблюдений k- го кластера.
- Назначьте каждое наблюдение кластеру с ближайшим центроидом. Здесь ближайший определяется с использованием евклидова расстояния .
Техническое примечание:
Поскольку k-medoids вычисляет центроиды кластера, используя медианы, а не средние значения, он имеет тенденцию быть более устойчивым к выбросам, чем k-средние.
На практике, если в наборе данных нет экстремальных выбросов, k-средние и k-медоиды дадут аналогичные результаты.
Кластеризация K-медоидов в R
В следующем руководстве представлен пошаговый пример выполнения кластеризации k-medoids в R.
Шаг 1. Загрузите необходимые пакеты
Сначала мы загрузим два пакета, содержащие несколько полезных функций для кластеризации k-медоидов в R.
library (factoextra) library (cluster)
Шаг 2. Загрузите и подготовьте данные
В этом примере мы будем использовать набор данных USArrests , встроенный в R, который содержит количество арестов на 100 000 человек в каждом штате США в 1973 году за убийства , нападения и изнасилования , а также процент населения каждого штата, проживающего в городах. области. , Урбанпоп .
Следующий код показывает, как сделать следующее:
- Загрузить набор данных USArrests
- Удалить все строки с пропущенными значениями
- Масштабируйте каждую переменную в наборе данных так, чтобы ее среднее значение было равно 0, а стандартное отклонение — 1.
#load data df <-USArrests #remove rows with missing values df <- na. omitted (df) #scale each variable to have a mean of 0 and sd of 1 df <- scale(df) #view first six rows of dataset head(df) Murder Assault UrbanPop Rape Alabama 1.24256408 0.7828393 -0.5209066 -0.003416473 Alaska 0.50786248 1.1068225 -1.2117642 2.484202941 Arizona 0.07163341 1.4788032 0.9989801 1.042878388 Arkansas 0.23234938 0.2308680 -1.0735927 -0.184916602 California 0.27826823 1.2628144 1.7589234 2.067820292 Colorado 0.02571456 0.3988593 0.8608085 1.864967207
Шаг 3. Найдите оптимальное количество кластеров
Чтобы выполнить кластеризацию k-medoid в R, мы можем использовать функцию pam() , которая означает «разбиение вокруг медиан» и использует следующий синтаксис:
pam(данные, k, метрика = «Евклидова», стенд = ЛОЖЬ)
Золото:
- данные: Имя набора данных.
- k: количество кластеров.
- метрика: метрика, используемая для расчета расстояния. По умолчанию используется евклидово значение, но вы также можете указать манхэттенское .
- стенд: следует ли нормализовать каждую переменную в наборе данных. Значение по умолчанию неверно.
Поскольку мы заранее не знаем, какое количество кластеров является оптимальным, мы создадим два разных графика, которые помогут нам принять решение:
1. Количество кластеров относительно общего количества в сумме квадратов
Сначала мы воспользуемся функцией fviz_nbclust() , чтобы построить график зависимости количества кластеров от суммы квадратов:
fviz_nbclust(df, pam, method = “ wss ”)
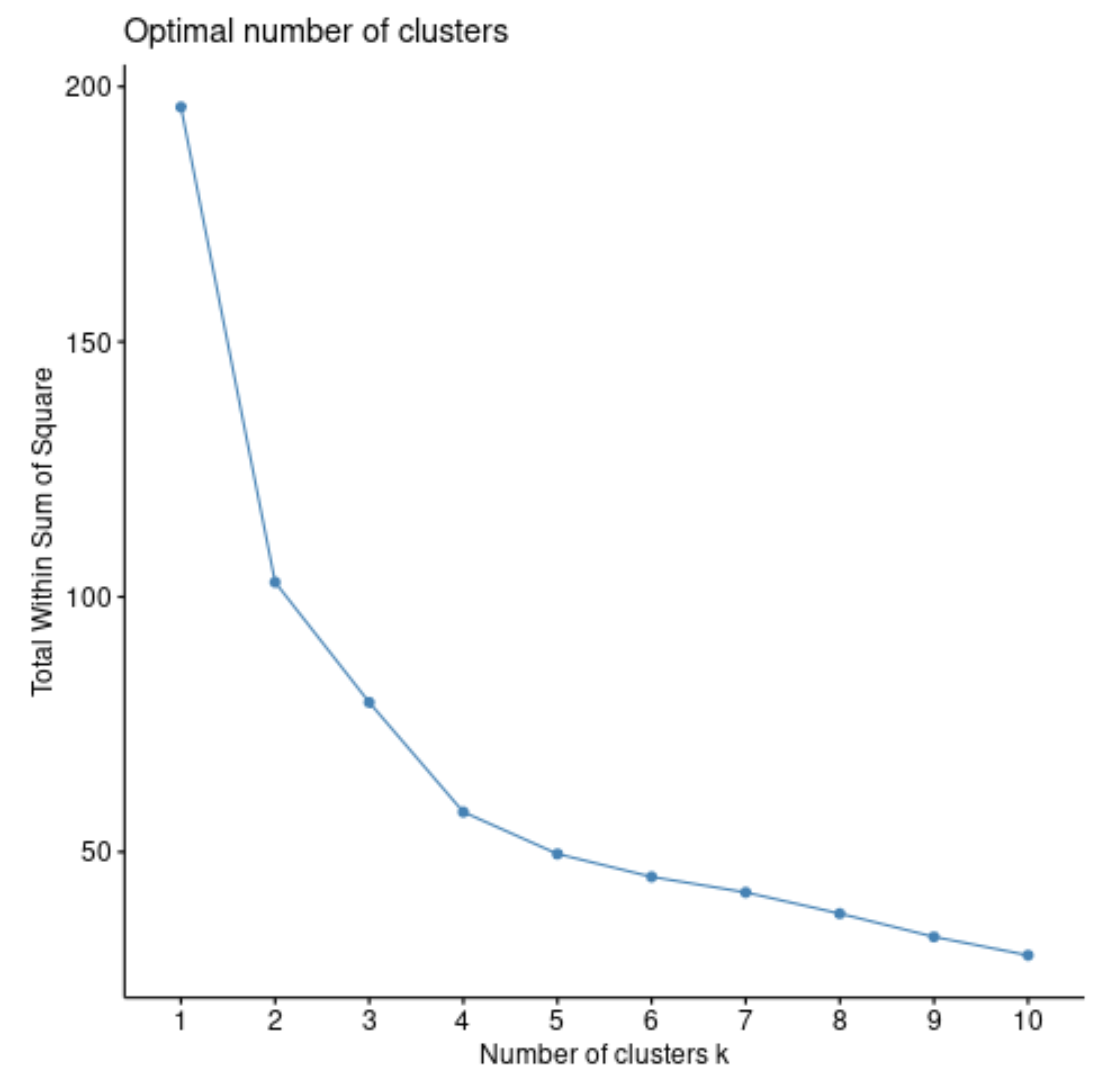
Общая сумма квадратов обычно всегда будет увеличиваться по мере увеличения количества кластеров. Поэтому, когда мы создаем этот тип графика, мы ищем «колено», где сумма квадратов начинает «изгибаться» или выравниваться.
Точка кривизны графика обычно соответствует оптимальному количеству кластеров. За пределами этой цифры, скорее всего, произойдет переобучение .
На этом графике видно, что имеется небольшой излом или «изгиб» при k = 4 кластерах.
2. Количество кластеров в сравнении со статистикой пробелов
Другой способ определить оптимальное количество кластеров — использовать метрику, называемую статистикой отклонений , которая сравнивает общую внутрикластерную вариацию для разных значений k с их ожидаемыми значениями для распределения без кластеризации.
Мы можем вычислить статистику разрыва для каждого количества кластеров, используя функцию clusGap() из пакета кластеров , а также построить график зависимости кластеров от статистики разрывов, используя функцию fviz_gap_stat() :
#calculate gap statistic based on number of clusters gap_stat <- clusGap(df, FUN = pam, K.max = 10, #max clusters to consider B = 50) #total bootstrapped iterations #plot number of clusters vs. gap statistic fviz_gap_stat(gap_stat)
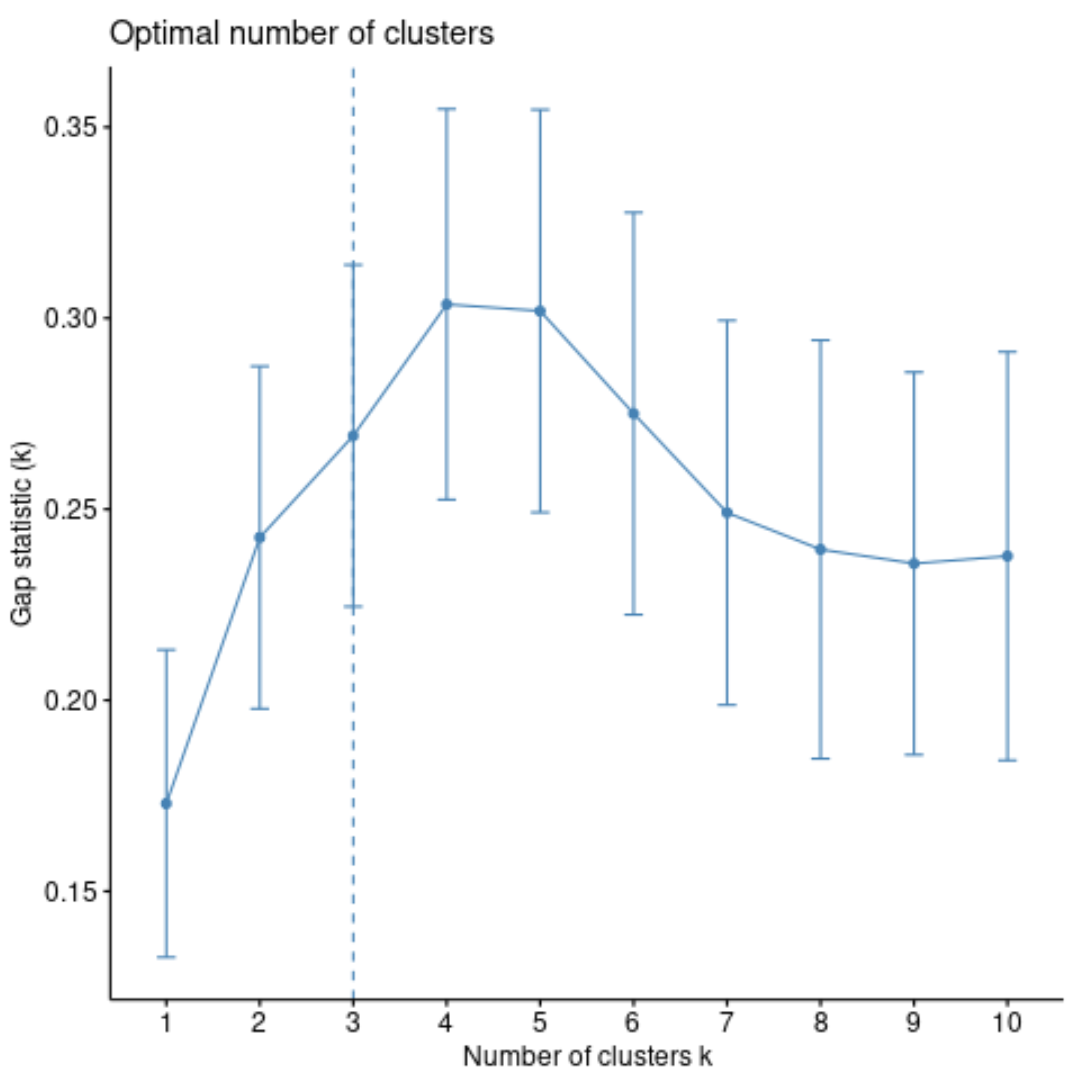
На графике мы видим, что статистика разрывов самая высокая при k = 4 кластерах, что соответствует методу локтя, который мы использовали ранее.
Шаг 4. Выполните кластеризацию K-Medoids с помощью Optimal K.
Наконец, мы можем выполнить кластеризацию k-медоидов в наборе данных, используя оптимальное значение k , равное 4:
#make this example reproducible set.seed(1) #perform k-medoids clustering with k = 4 clusters kmed <- pam(df, k = 4) #view results kmed ID Murder Assault UrbanPop Rape Alabama 1 1.2425641 0.7828393 -0.5209066 -0.003416473 Michigan 22 0.9900104 1.0108275 0.5844655 1.480613993 Oklahoma 36 -0.2727580 -0.2371077 0.1699510 -0.131534211 New Hampshire 29 -1.3059321 -1.3650491 -0.6590781 -1.252564419 Vector clustering: Alabama Alaska Arizona Arkansas California 1 2 2 1 2 Colorado Connecticut Delaware Florida Georgia 2 3 3 2 1 Hawaii Idaho Illinois Indiana Iowa 3 4 2 3 4 Kansas Kentucky Louisiana Maine Maryland 3 3 1 4 2 Massachusetts Michigan Minnesota Mississippi Missouri 3 2 4 1 3 Montana Nebraska Nevada New Hampshire New Jersey 3 3 2 4 3 New Mexico New York North Carolina North Dakota Ohio 2 2 1 4 3 Oklahoma Oregon Pennsylvania Rhode Island South Carolina 3 3 3 3 1 South Dakota Tennessee Texas Utah Vermont 4 1 2 3 4 Virginia Washington West Virginia Wisconsin Wyoming 3 3 4 4 3 Objective function: build swap 1.035116 1.027102 Available components: [1] "medoids" "id.med" "clustering" "objective" "isolation" [6] "clusinfo" "silinfo" "diss" "call" "data"
Обратите внимание, что все четыре центроида кластера являются фактическими наблюдениями в наборе данных. В верхней части вывода мы видим, что четыре центроида представляют собой следующие состояния:
- Алабама
- Мичиган
- Оклахома
- Нью-Гемпшир
Мы можем визуализировать кластеры на диаграмме рассеяния, которая отображает первые два главных компонента на осях, используя функцию fivz_cluster() :
#plot results of final k-medoids model
fviz_cluster(kmed, data = df)
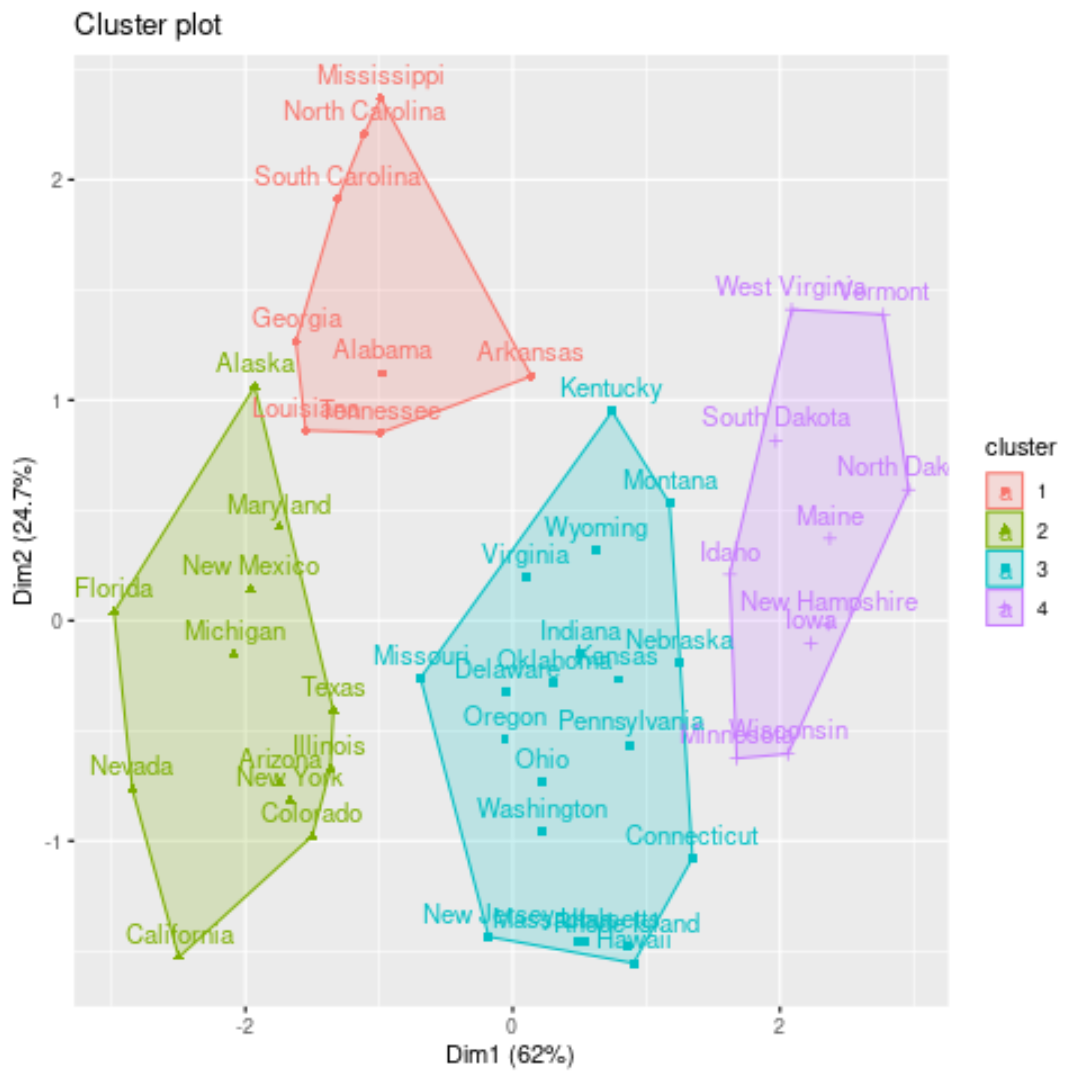
Мы также можем добавить назначения кластеров каждого состояния в исходный набор данных:
#add cluster assignment to original data
final_data <- cbind(USArrests, cluster = kmed$cluster)
#view final data
head(final_data)
Murder Assault UrbanPop Rape cluster
Alabama 13.2 236 58 21.2 1
Alaska 10.0 263 48 44.5 2
Arizona 8.1 294 80 31.0 2
Arkansas 8.8 190 50 19.5 1
California 9.0 276 91 40.6 2
Colorado 7.9 204 78 38.7 2
Полный код R, использованный в этом примере, вы можете найти здесь .