วิธีดำเนินการทดสอบ log rank ใน r
การทดสอบอันดับบันทึก เป็นวิธีที่ใช้กันทั่วไปในการเปรียบเทียบเส้นโค้งการอยู่รอดระหว่างสองกลุ่ม
การทดสอบนี้ใช้ สมมติฐาน ดังต่อไปนี้:
H 0 : ไม่มีความแตกต่างในการอยู่รอดระหว่างทั้งสองกลุ่ม
HA : มี ความแตกต่างในการเอาชีวิตรอดระหว่างทั้งสองกลุ่ม
หาก ค่า p ของการทดสอบต่ำกว่าระดับนัยสำคัญที่กำหนด (เช่น α = 0.05) เราก็สามารถปฏิเสธสมมติฐานที่เป็นโมฆะได้และสรุปว่ามีหลักฐานเพียงพอที่จะบอกว่ามีความแตกต่างในการอยู่รอดระหว่างทั้งสองกลุ่ม
หากต้องการทำการทดสอบระดับบันทึกใน R เราสามารถใช้ฟังก์ชัน survdiff() จากแพ็คเกจ survival ซึ่งใช้ไวยากรณ์ต่อไปนี้:
Survdiff(ความอยู่รอด(เวลา, สถานะ) ~ ตัวทำนาย, ข้อมูล)
ฟังก์ชันนี้จะส่งคืนสถิติการทดสอบไคสแควร์และค่า p ที่สอดคล้องกัน
ตัวอย่างต่อไปนี้แสดงวิธีใช้ฟังก์ชันนี้เพื่อทำการทดสอบระดับบันทึกใน R
ตัวอย่าง: การทดสอบอันดับบันทึกใน R
สำหรับตัวอย่างนี้ เราจะใช้ชุดข้อมูล รังไข่ จากแพ็คเกจ การอยู่รอด ชุดข้อมูลนี้ประกอบด้วยข้อมูลต่อไปนี้เกี่ยวกับผู้ป่วย 26 ราย:
- ระยะเวลารอดชีวิต (เป็นเดือน) หลังจากได้รับการวินิจฉัยว่าเป็นมะเร็งรังไข่
- มีการเซ็นเซอร์เวลาเอาชีวิตรอดหรือไม่
- ประเภทการรักษาที่ได้รับ (rx = 1 หรือ rx = 2)
รหัสต่อไปนี้แสดงวิธีการแสดงหกแถวแรกของชุดข้อมูลนี้:
library (survival) #view first six rows of dataset head(ovarian) futime fustat age resid.ds rx ecog.ps 1 59 1 72.3315 2 1 1 2 115 1 74.4932 2 1 1 3 156 1 66.4658 2 1 2 4 421 0 53.3644 2 2 1 5,431 1 50.3397 2 1 1 6 448 0 56.4301 1 1 2
รหัสต่อไปนี้แสดงวิธีดำเนินการทดสอบระดับบันทึกเพื่อตรวจสอบว่ามีความแตกต่างในการรอดชีวิตระหว่างผู้ป่วยที่ได้รับการรักษาที่แตกต่างกันหรือไม่:
#perform log rank test
survdiff(Surv(futime, fustat) ~ rx, data=ovarian)
Call:
survdiff(formula = Surv(futime, fustat) ~ rx, data = ovarian)
N Observed Expected (OE)^2/E (OE)^2/V
rx=1 13 7 5.23 0.596 1.06
rx=2 13 5 6.77 0.461 1.06
Chisq= 1.1 on 1 degrees of freedom, p= 0.3
สถิติการทดสอบไคสแควร์คือ 1.1 โดยมีระดับความอิสระ 1 องศา และค่า p ที่สอดคล้องกันคือ 0.3 เนื่องจากค่า p นี้ไม่น้อยกว่า 0.05 เราจึงไม่สามารถปฏิเสธสมมติฐานว่างได้
กล่าวอีกนัยหนึ่ง เราไม่มีหลักฐานเพียงพอที่จะกล่าวว่ามีความแตกต่างที่มีนัยสำคัญทางสถิติในการรอดชีวิตระหว่างการรักษาทั้งสอง
นอกจากนี้เรายังสามารถวาดเส้นโค้งการเอาชีวิตรอดสำหรับแต่ละกลุ่มได้โดยใช้ไวยากรณ์ต่อไปนี้:
#plot survival curves for each treatment group plot(survfit(Surv(futime, fustat) ~ rx, data = ovarian), xlab = " Time ", ylab = “ Overall survival probability ”)
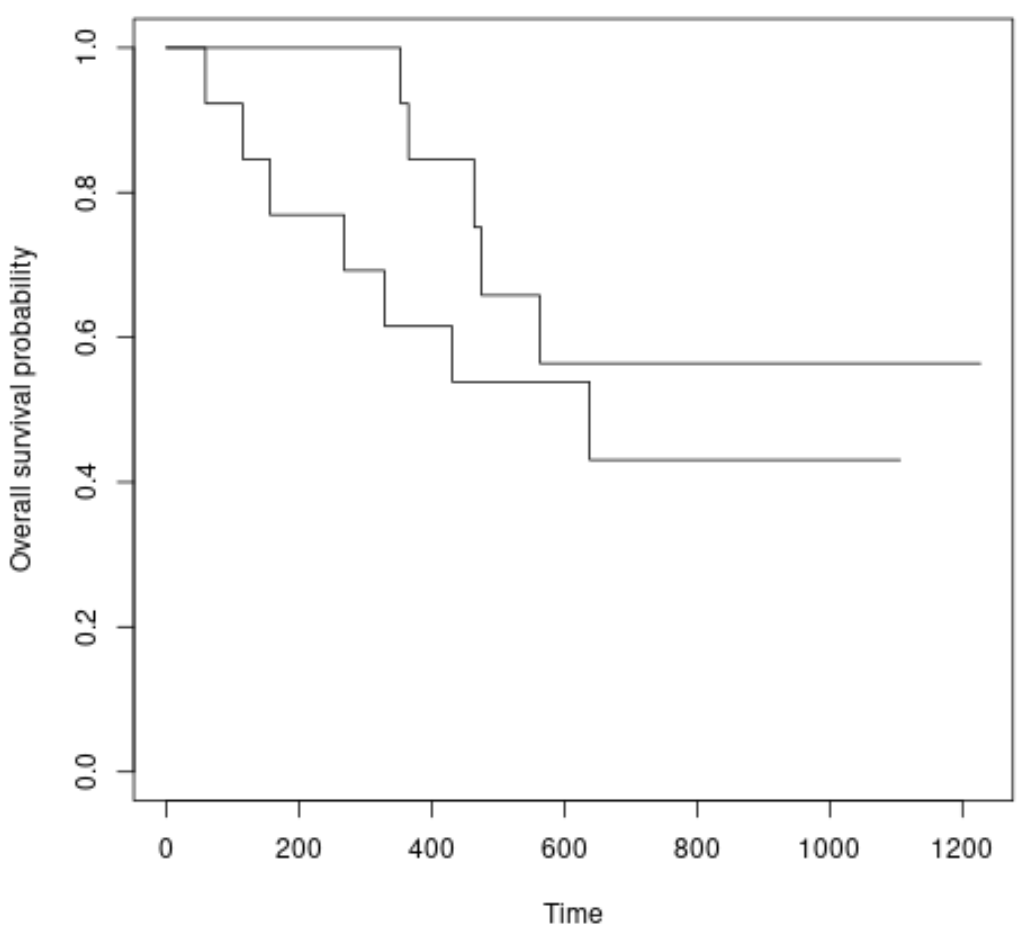
เราจะเห็นว่าเส้นโค้งการเอาชีวิตรอดแตกต่างกันเล็กน้อย แต่การทดสอบระดับบันทึกบอกเราว่าความแตกต่างนั้นไม่มีนัยสำคัญทางสถิติ