Python'da temel bileşen regresyon (adım adım)
Bir dizi p tahmin değişkeni ve bir yanıt değişkeni verildiğinde, çoklu doğrusal regresyon , kalan kareler toplamını (RSS) en aza indirmek için en küçük kareler olarak bilinen bir yöntemi kullanır:
RSS = Σ(y ben – ŷ ben ) 2
Altın:
- Σ : Toplam anlamına gelen bir Yunan sembolü
- y i : i’inci gözlem için gerçek yanıt değeri
- ŷ i : Çoklu doğrusal regresyon modeline dayalı olarak tahmin edilen yanıt değeri
Ancak yordayıcı değişkenler yüksek düzeyde korelasyona sahip olduğunda çoklu doğrusallık bir sorun haline gelebilir. Bu, model katsayı tahminlerini güvenilmez hale getirebilir ve yüksek varyans sergileyebilir.
Bu sorunu önlemenin bir yolu, orijinal p tahmincilerinin M doğrusal kombinasyonunu (“temel bileşenler” olarak adlandırılır) bulan ve ardından temel bileşenleri tahminci olarak kullanan doğrusal bir regresyon modeline uymak için en küçük kareleri kullanan temel bileşenler regresyonunu kullanmaktır.
Bu eğitimde Python’da temel bileşenlerin regresyonunun nasıl gerçekleştirileceğine ilişkin adım adım bir örnek sunulmaktadır.
Adım 1: Gerekli paketleri içe aktarın
İlk olarak Python’da temel bileşen regresyonunu (PCR) gerçekleştirmek için gereken paketleri içe aktaracağız:
import numpy as np
import pandas as pd
import matplotlib. pyplot as plt
from sklearn. preprocessing import scale
from sklearn import model_selection
from sklearn. model_selection import RepeatedKFold
from sklearn.model_selection import train_test_split
from sklearn. PCA import decomposition
from sklearn. linear_model import LinearRegression
from sklearn. metrics import mean_squared_error
2. Adım: Verileri yükleyin
Bu örnek için mtcars adı verilen ve 33 farklı araba hakkında bilgi içeren bir veri kümesi kullanacağız. Yanıt değişkeni olarak hp’yi ve yordayıcılar olarak aşağıdaki değişkenleri kullanacağız:
- mpg
- görüntülemek
- bok
- ağırlık
- qsec
Aşağıdaki kod bu veri kümesinin nasıl yüklenip görüntüleneceğini gösterir:
#define URL where data is located
url = "https://raw.githubusercontent.com/Statorials/Python-Guides/main/mtcars.csv"
#read in data
data_full = pd. read_csv (url)
#select subset of data
data = data_full[["mpg", "disp", "drat", "wt", "qsec", "hp"]]
#view first six rows of data
data[0:6]
mpg disp drat wt qsec hp
0 21.0 160.0 3.90 2.620 16.46 110
1 21.0 160.0 3.90 2.875 17.02 110
2 22.8 108.0 3.85 2.320 18.61 93
3 21.4 258.0 3.08 3.215 19.44 110
4 18.7 360.0 3.15 3.440 17.02 175
5 18.1 225.0 2.76 3.460 20.22 105
Adım 3: PCR modelini ayarlayın
Aşağıdaki kod PCR modelinin bu verilere nasıl sığdırılacağını gösterir. Aşağıdakilere dikkat et:
- pca.fit_transform(scale(X)) : Bu, Python’a tahmin değişkenlerinin her birinin ortalaması 0 ve standart sapması 1 olacak şekilde ölçeklendirilmesi gerektiğini söyler. Bu, hiçbir tahmin değişkeninin modelde çok fazla etkiye sahip olmamasını sağlar. bu meydana gelir. farklı birimlerle ölçülecektir.
- cv = RepeatedKFold() : Bu, Python’a model performansını değerlendirmek için k-katlı çapraz doğrulama kullanmasını söyler. Bu örnek için k = 10 kat seçiyoruz, bu 3 kez tekrarlanıyor.
#define predictor and response variables
X = data[["mpg", "disp", "drat", "wt", "qsec"]]
y = data[["hp"]]
#scale predictor variables
pca = pca()
X_reduced = pca. fit_transform ( scale (X))
#define cross validation method
cv = RepeatedKFold(n_splits= 10 , n_repeats= 3 , random_state= 1 )
regr = LinearRegression()
mse = []
# Calculate MSE with only the intercept
score = -1*model_selection. cross_val_score (regr,
n.p. ones ((len(X_reduced),1)), y, cv=cv,
scoring=' neg_mean_squared_error '). mean ()
mse. append (score)
# Calculate MSE using cross-validation, adding one component at a time
for i in np. arange (1, 6):
score = -1*model_selection. cross_val_score (regr,
X_reduced[:,:i], y, cv=cv, scoring=' neg_mean_squared_error '). mean ()
mse. append (score)
# Plot cross-validation results
plt. plot (mse)
plt. xlabel ('Number of Principal Components')
plt. ylabel ('MSE')
plt. title ('hp')
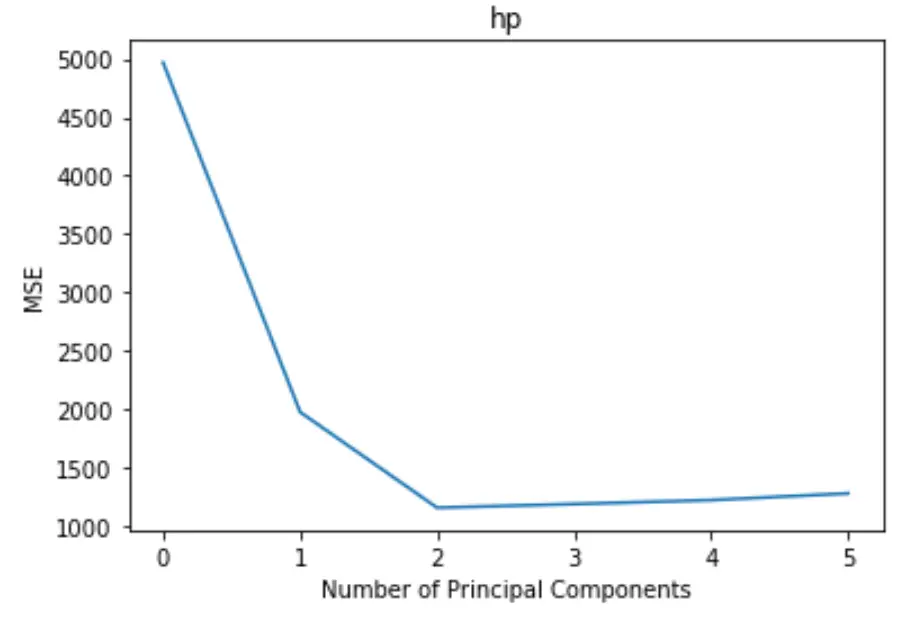
Grafik, x ekseni boyunca ana bileşenlerin sayısını ve y ekseni boyunca MSE (ortalama kare hatası) testini görüntüler.
Grafikten, testin MSE’sinin iki temel bileşen eklendiğinde azaldığını ancak ikiden fazla temel bileşen ekledikçe artmaya başladığını görebiliriz.
Bu nedenle optimal model yalnızca ilk iki temel bileşeni içerir.
Her bir temel bileşeni modele ekleyerek açıklanan yanıt değişkenindeki varyans yüzdesini hesaplamak için aşağıdaki kodu da kullanabiliriz:
n.p. cumsum (np. round (pca. explained_variance_ratio_ , decimals= 4 )* 100 )
array([69.83, 89.35, 95.88, 98.95, 99.99])
Aşağıdakileri görebiliriz:
- Yalnızca ilk temel bileşeni kullanarak yanıt değişkenindeki varyasyonun %69,83’ünü açıklayabiliriz.
- İkinci temel bileşeni ekleyerek yanıt değişkenindeki varyasyonun %89,35’ini açıklayabiliriz.
Daha fazla temel bileşen kullanarak hala daha fazla varyansı açıklayabileceğimizi unutmayın, ancak ikiden fazla temel bileşen eklemenin aslında açıklanan varyans yüzdesini çok fazla artırmadığını görebiliriz.
Adım 4: Tahminlerde bulunmak için son modeli kullanın
Yeni gözlemler hakkında tahminlerde bulunmak için son iki temel bileşenli PCR modelini kullanabiliriz.
Aşağıdaki kod, orijinal veri kümesinin bir eğitim ve test kümesine nasıl bölüneceğini ve test kümesi üzerinde tahminler yapmak için PCR modelinin iki temel bileşenle nasıl kullanılacağını gösterir.
#split the dataset into training (70%) and testing (30%) sets
X_train,X_test,y_train,y_test = train_test_split (X,y,test_size= 0.3 , random_state= 0 )
#scale the training and testing data
X_reduced_train = pca. fit_transform ( scale (X_train))
X_reduced_test = pca. transform ( scale (X_test))[:,:1]
#train PCR model on training data
regr = LinearRegression()
reg. fit (X_reduced_train[:,:1], y_train)
#calculate RMSE
pred = regr. predict (X_reduced_test)
n.p. sqrt ( mean_squared_error (y_test, pred))
40.2096
RMSE testinin 40.2096 çıktığını görüyoruz. Bu, test seti gözlemleri için tahmin edilen hp değeri ile gözlenen hp değeri arasındaki ortalama sapmadır.
Bu örnekte kullanılan Python kodunun tamamını burada bulabilirsiniz.