R'de temel bileşen regresyon (adım adım)
Bir dizi p tahmin değişkeni ve bir yanıt değişkeni verildiğinde, çoklu doğrusal regresyon , kalan kareler toplamını (RSS) en aza indirmek için en küçük kareler olarak bilinen bir yöntemi kullanır:
RSS = Σ(y ben – ŷ ben ) 2
Altın:
- Σ : Toplam anlamına gelen bir Yunan sembolü
- y i : i’inci gözlem için gerçek yanıt değeri
- ŷ i : Çoklu doğrusal regresyon modeline dayalı olarak tahmin edilen yanıt değeri
Ancak yordayıcı değişkenler yüksek düzeyde korelasyona sahip olduğunda çoklu doğrusallık bir sorun haline gelebilir. Bu, model katsayı tahminlerini güvenilmez hale getirebilir ve yüksek varyans sergileyebilir.
Bu sorunu önlemenin bir yolu, orijinal p tahmincilerinin M doğrusal kombinasyonunu (“temel bileşenler” olarak adlandırılır) bulan ve ardından temel bileşenleri tahminci olarak kullanan doğrusal bir regresyon modeline uymak için en küçük kareleri kullanan temel bileşenler regresyonunu kullanmaktır.
Bu eğitimde, R’de temel bileşenler regresyonunun nasıl gerçekleştirileceğine ilişkin adım adım bir örnek sağlanmaktadır.
Adım 1: Gerekli paketleri yükleyin
R’de temel bileşenler regresyonunu gerçekleştirmenin en kolay yolu pls paketindeki işlevleri kullanmaktır.
#install pls package (if not already installed) install.packages(" pls ") load pls package library(pls)
Adım 2: PCR modelini ayarlayın
Bu örnek için, farklı araba türlerine ilişkin verileri içeren mtcars adı verilen yerleşik R veri kümesini kullanacağız:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
Bu örnek için, yanıt değişkeni olarak hp’yi ve tahmin değişkenleri olarak aşağıdaki değişkenleri kullanarak bir temel bileşenler regresyon (PCR) modelini uygulayacağız:
- mpg
- görüntülemek
- bok
- ağırlık
- qsec
Aşağıdaki kod PCR modelinin bu verilere nasıl sığdırılacağını gösterir. Aşağıdaki argümanlara dikkat edin:
- ölçek=DOĞRU : Bu, R’ye yordayıcı değişkenlerin her birinin ortalaması 0 ve standart sapması 1 olacak şekilde ölçeklendirilmesi gerektiğini söyler. Bu, farklı birimlerle ölçüldüğünde hiçbir yordayıcı değişkenin modelde çok fazla etkiye sahip olmamasını sağlar. .
- validation=”CV” : Bu, R’ye model performansını değerlendirmek için k-katlı çapraz doğrulama kullanmasını söyler. Bunun varsayılan olarak k=10 kat kullandığını unutmayın. Ayrıca Birini Dışarıda Bırakma çapraz doğrulaması gerçekleştirmek yerine “LOOCV”yi belirtebileceğinizi de unutmayın.
#make this example reproducible set.seed(1) #fit PCR model model <- pcr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
3. Adım: Ana bileşenlerin sayısını seçin
Modeli ayarladıktan sonra, kaç temel bileşenin korunmaya değer olduğunu belirlememiz gerekiyor.
Bunu yapmak için k-çapraz doğrulamayla hesaplanan test kök ortalama kare hatasına (test RMSE) bakmanız yeterlidir:
#view summary of model fitting
summary(model)
Data:
Y dimension: 32 1
Fit method: svdpc
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comp 2 comps 3 comps 4 comps 5 comps
CV 69.66 44.56 35.64 35.83 36.23 36.67
adjCV 69.66 44.44 35.27 35.43 35.80 36.20
TRAINING: % variance explained
1 comp 2 comps 3 comps 4 comps 5 comps
X 69.83 89.35 95.88 98.96 100.00
hp 62.38 81.31 81.96 81.98 82.03
Sonuçta iki ilginç tablo var:
1. DOĞRULAMA: RMSEP
Bu tablo bize k-katlı çapraz doğrulamayla hesaplanan RMSE testini anlatır. Aşağıdakileri görebiliriz:
- Modelde sadece orijinal terimi kullanırsak testin RMSE’si 69,66 olur.
- İlk temel bileşeni de eklersek RMSE testi 44,56’ya düşüyor.
- İkinci temel bileşeni de eklersek RMSE testi 35,64’e düşüyor.
Ek temel bileşenlerin eklenmesinin aslında testin RMSE’sinde bir artışa yol açtığını görebiliriz. Bu nedenle nihai modelde yalnızca iki temel bileşenin kullanılmasının optimal olacağı görülmektedir.
2. EĞİTİM: açıklanan varyans yüzdesi
Bu tablo bize temel bileşenler tarafından açıklanan yanıt değişkenindeki varyansın yüzdesini gösterir. Aşağıdakileri görebiliriz:
- Yalnızca ilk temel bileşeni kullanarak yanıt değişkenindeki varyasyonun %69,83’ünü açıklayabiliriz.
- İkinci temel bileşeni ekleyerek yanıt değişkenindeki varyasyonun %89,35’ini açıklayabiliriz.
Daha fazla temel bileşen kullanarak hala daha fazla varyansı açıklayabileceğimizi unutmayın, ancak ikiden fazla temel bileşen eklemenin aslında açıklanan varyans yüzdesini çok fazla artırmadığını görebiliriz.
Validationplot() işlevini kullanarak RMSE testini (MSE ve R-kare testiyle birlikte) ana bileşenlerin sayısının bir fonksiyonu olarak da görselleştirebiliriz.
#visualize cross-validation plots
validationplot(model)
validationplot(model, val.type="MSEP")
validationplot(model, val.type="R2")
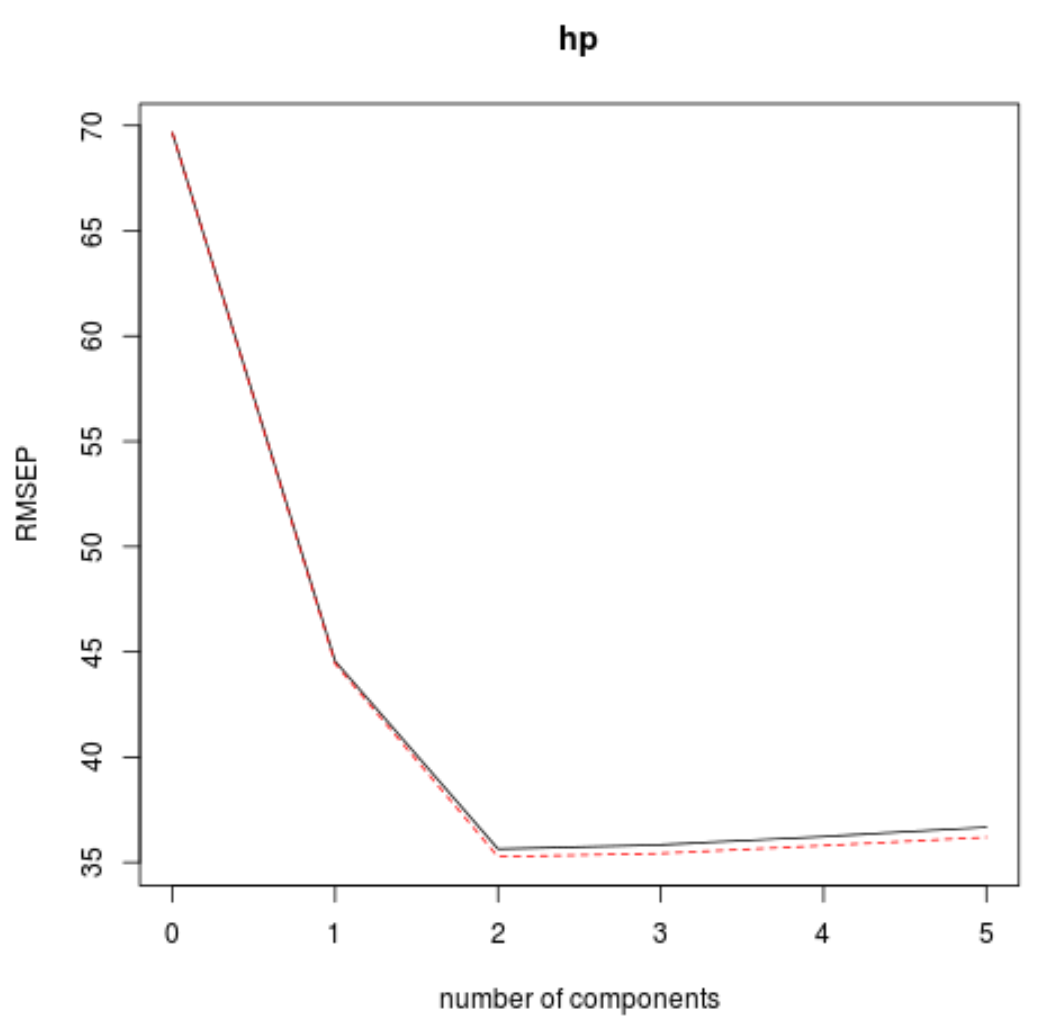
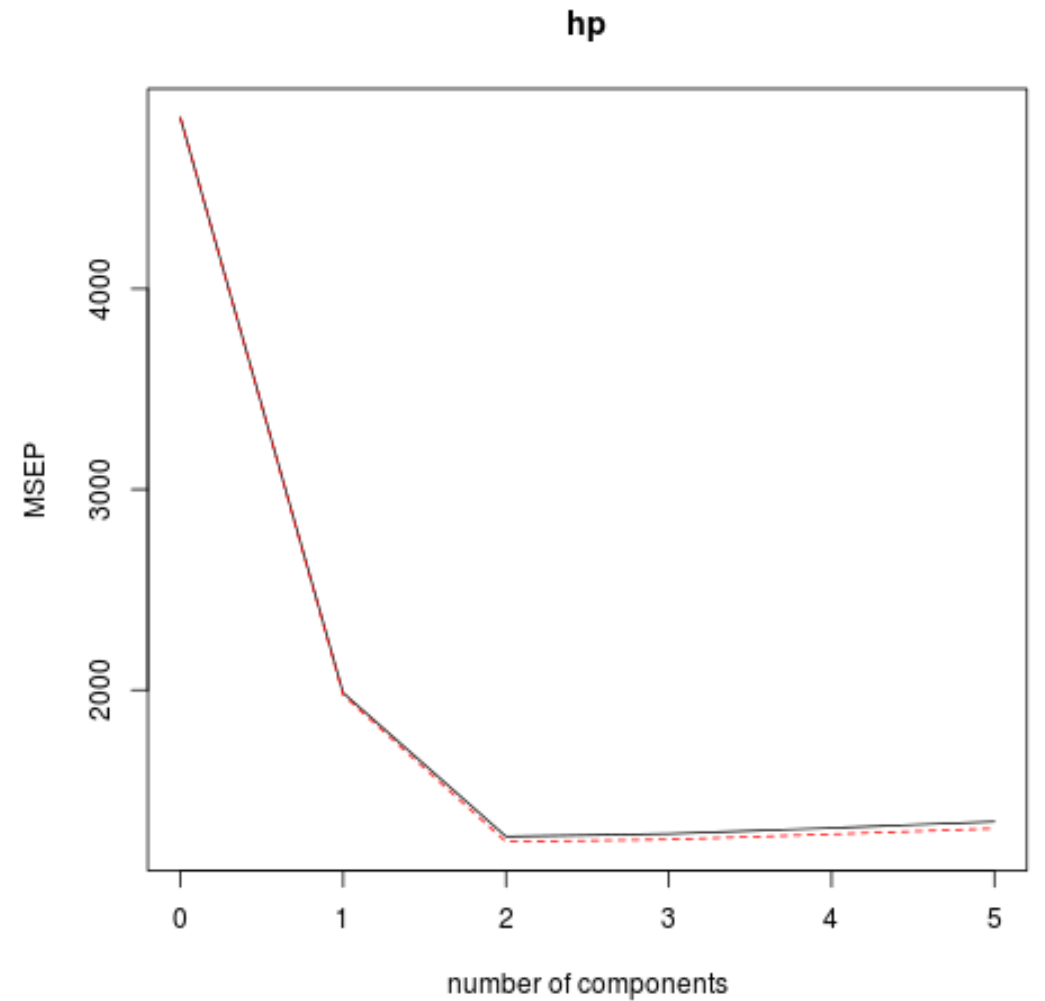
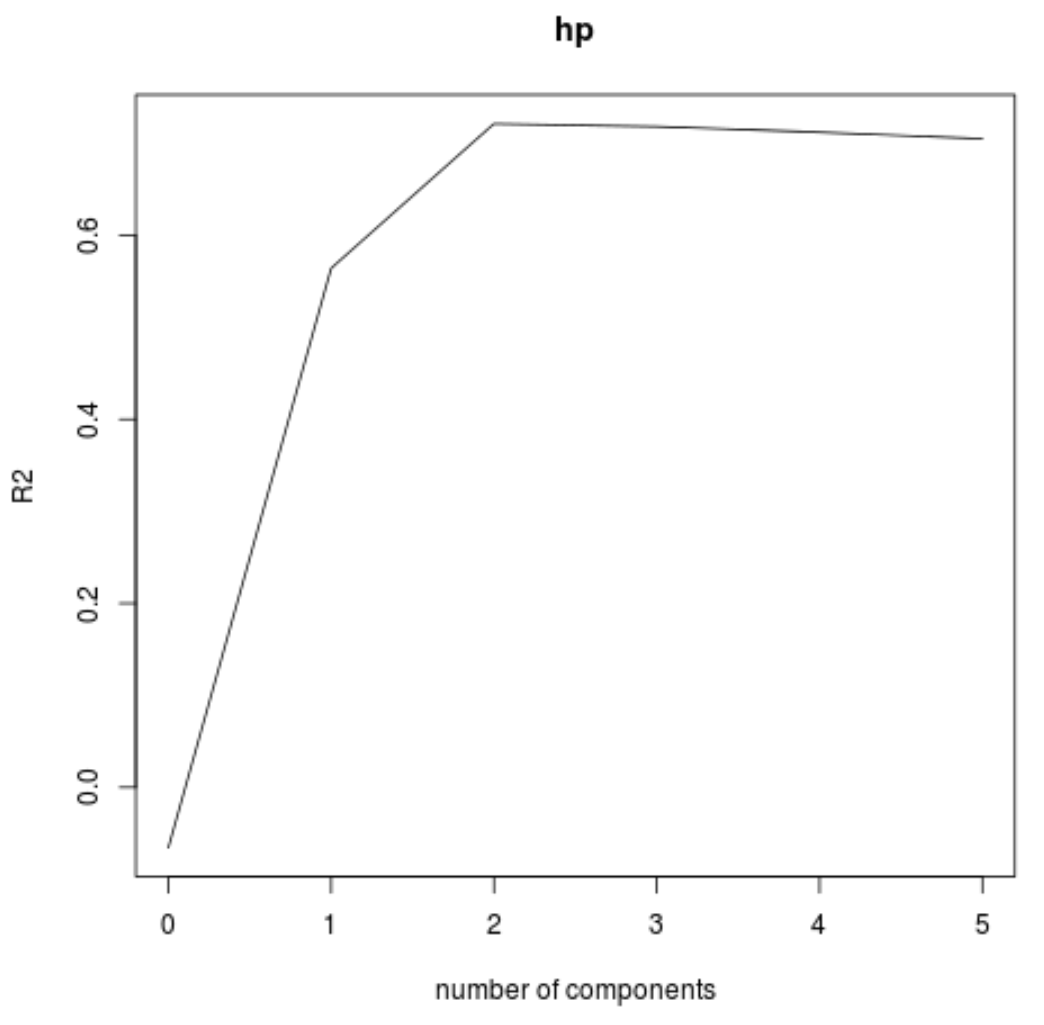
Her grafikte, model uyumunun iki temel bileşen eklendiğinde arttığını ancak daha fazla temel bileşen eklediğimizde bozulma eğiliminde olduğunu görebiliriz.
Bu nedenle optimal model yalnızca ilk iki temel bileşeni içerir.
Adım 4: Tahminlerde bulunmak için son modeli kullanın
Yeni gözlemler hakkında tahminlerde bulunmak için son iki temel bileşenli PCR modelini kullanabiliriz.
Aşağıdaki kod, orijinal veri kümesinin bir eğitim ve test kümesine nasıl bölüneceğini ve test kümesi üzerinde tahminler yapmak için PCR modelinin iki temel bileşenle nasıl kullanılacağını gösterir.
#define training and testing sets train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")] y_test <- mtcars[26: nrow (mtcars), c("hp")] test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")] #use model to make predictions on a test set model <- pcr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ") pcr_pred <- predict(model, test, ncomp= 2 ) #calculate RMSE sqrt ( mean ((pcr_pred - y_test)^2)) [1] 56.86549
Testin RMSE’sinin 56,86549 olduğunu görüyoruz. Bu, test seti gözlemleri için tahmin edilen hp değeri ile gözlenen hp değeri arasındaki ortalama sapmadır.
Bu örnekteki R kodunun tam kullanımını burada bulabilirsiniz.