Регресія головного компонента в r (покроково)
Враховуючи набір із p предикторних змінних і змінну відповіді, множинна лінійна регресія використовує метод найменших квадратів для мінімізації залишкової суми квадратів (RSS):
RSS = Σ(y i – ŷ i ) 2
золото:
- Σ : грецький символ, що означає суму
- y i : фактичне значення відповіді для i-го спостереження
- ŷ i : прогнозоване значення відповіді на основі моделі множинної лінійної регресії
Однак, коли прогностичні змінні сильно корельовані, мультиколінеарність може стати проблемою. Це може зробити оцінки коефіцієнтів моделі ненадійними та мати високу дисперсію.
Одним зі способів уникнути цієї проблеми є використання регресії головних компонентів , яка знаходить M лінійних комбінацій (званих «головними компонентами») вихідних p предикторів, а потім використовує найменші квадрати, щоб відповідати моделі лінійної регресії, використовуючи головні компоненти як предиктори.
Цей підручник надає покроковий приклад виконання регресії головних компонентів у R.
Крок 1: Завантажте необхідні пакети
Найпростіший спосіб виконати регресію головних компонентів у R – це використовувати функції в пакеті pls .
#install pls package (if not already installed) install.packages(" pls ") load pls package library(pls)
Крок 2: Налаштуйте модель ПЛР
Для цього прикладу ми використаємо вбудований набір даних R під назвою mtcars , який містить дані про різні типи автомобілів:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
Для цього прикладу ми підберемо модель регресії головних компонентів (PCR), використовуючи hp як змінну відповіді та наступні змінні як змінні предиктора:
- миль на галлон
- дисплей
- лайно
- вага
- qsec
У наведеному нижче коді показано, як адаптувати модель ПЛР до цих даних. Зверніть увагу на такі аргументи:
- scale=TRUE : це повідомляє R, що кожна змінна предиктора повинна бути масштабована, щоб мати середнє значення 0 і стандартне відхилення 1. Це гарантує, що жодна змінна предиктора не має надто сильного впливу на модель, якщо вона вимірюється в різних одиницях. .
- validation=”CV” : Це повідомляє R використовувати k-кратну перехресну перевірку для оцінки продуктивності моделі. Зауважте, що за умовчанням використовується k=10 згорток. Також зауважте, що замість цього можна вказати «LOOCV», щоб виконати перехресну перевірку Leave-One-Out .
#make this example reproducible set.seed(1) #fit PCR model model <- pcr(hp~mpg+disp+drat+wt+qsec, data=mtcars, scale= TRUE , validation=" CV ")
Крок 3: Виберіть кількість основних компонентів
Після того, як ми налаштували модель, нам потрібно визначити, скільки основних компонентів варто зберегти.
Для цього просто подивіться на тестову середню квадратичну помилку (тест RMSE), розраховану за допомогою k-cross перевірки:
#view summary of model fitting
summary(model)
Data:
Y dimension: 32 1
Fit method: svdpc
Number of components considered: 5
VALIDATION: RMSEP
Cross-validated using 10 random segments.
(Intercept) 1 comp 2 comps 3 comps 4 comps 5 comps
CV 69.66 44.56 35.64 35.83 36.23 36.67
adjCV 69.66 44.44 35.27 35.43 35.80 36.20
TRAINING: % variance explained
1 comp 2 comps 3 comps 4 comps 5 comps
X 69.83 89.35 95.88 98.96 100.00
hp 62.38 81.31 81.96 81.98 82.03
У результаті вийшли дві цікаві таблиці:
1. ПЕРЕВІРКА: RMSEP
У цій таблиці наведено тест RMSE, розрахований шляхом k-кратної перехресної перевірки. Ми бачимо наступне:
- Якщо ми використовуємо лише вихідний термін у моделі, RMSE тесту становить 69,66 .
- Якщо ми додамо перший головний компонент, тест RMSE падає до 44,56.
- Якщо ми додамо другий головний компонент, тест RMSE падає до 35,64.
Ми бачимо, що додавання додаткових головних компонентів фактично призводить до збільшення RMSE тесту. Таким чином, здається, що оптимальним було б використовувати лише два основних компоненти в кінцевій моделі.
2. НАВЧАННЯ: пояснення % відхилення
Ця таблиця повідомляє нам про відсоток дисперсії у змінній відповіді, що пояснюється головними компонентами. Ми бачимо наступне:
- Використовуючи лише перший головний компонент, ми можемо пояснити 69,83% варіації змінної відповіді.
- Додавши другий головний компонент, ми можемо пояснити 89,35% варіації змінної відповіді.
Зауважте, що ми все ще зможемо пояснити більшу дисперсію, використовуючи більше головних компонентів, але ми бачимо, що додавання більше двох головних компонентів фактично не значно збільшує відсоток поясненої дисперсії.
Ми також можемо візуалізувати тест RMSE (поряд із тестом MSE та R-квадрат) як функцію кількості основних компонентів за допомогою функції validationplot() .
#visualize cross-validation plots
validationplot(model)
validationplot(model, val.type="MSEP")
validationplot(model, val.type="R2")
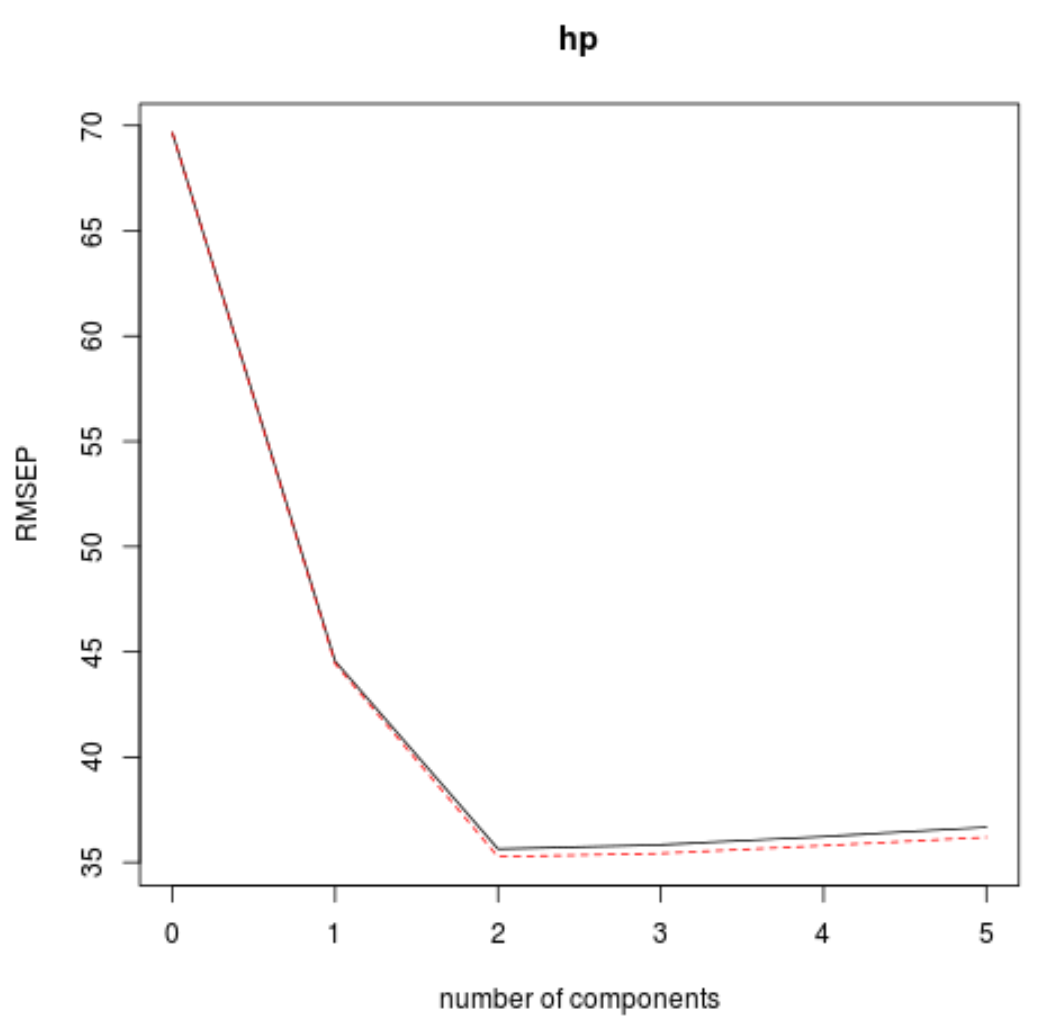
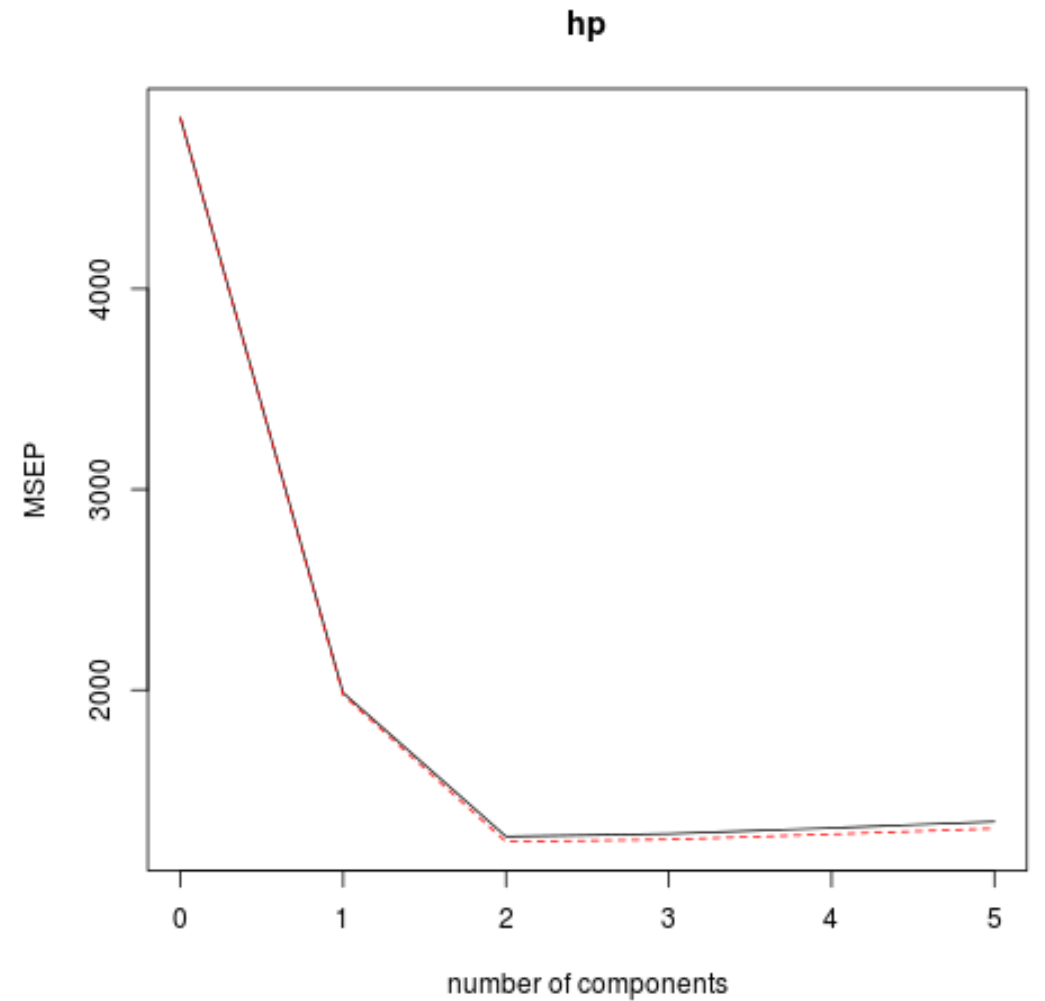
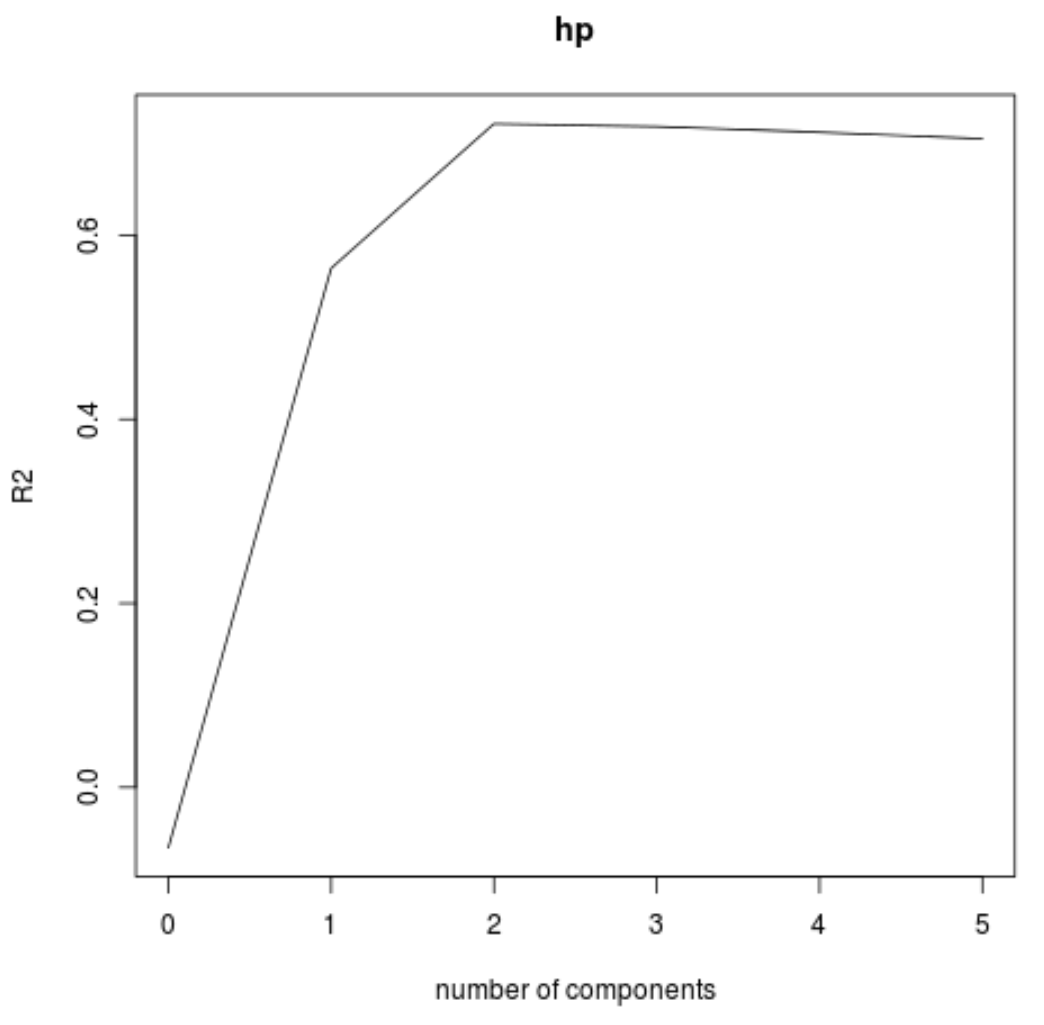
На кожному графіку ми бачимо, що підгонка моделі покращується шляхом додавання двох головних компонентів, але має тенденцію погіршуватися, коли ми додаємо більше головних компонентів.
Таким чином, оптимальна модель включає лише перші дві основні складові.
Крок 4. Використовуйте остаточну модель для прогнозування
Ми можемо використовувати остаточну двокомпонентну модель ПЛР, щоб зробити прогнози щодо нових спостережень.
У наведеному нижче коді показано, як розділити вихідний набір даних на навчальний і тестовий набір і використовувати модель ПЛР із двома основними компонентами для прогнозування на тестовому наборі.
#define training and testing sets train <- mtcars[1:25, c("hp", "mpg", "disp", "drat", "wt", "qsec")] y_test <- mtcars[26: nrow (mtcars), c("hp")] test <- mtcars[26: nrow (mtcars), c("mpg", "disp", "drat", "wt", "qsec")] #use model to make predictions on a test set model <- pcr(hp~mpg+disp+drat+wt+qsec, data=train, scale= TRUE , validation=" CV ") pcr_pred <- predict(model, test, ncomp= 2 ) #calculate RMSE sqrt ( mean ((pcr_pred - y_test)^2)) [1] 56.86549
Ми бачимо, що RMSE тесту виявляється 56,86549 . Це середнє відхилення між прогнозованим значенням hp і спостережуваним значенням hp для спостережень тестового набору.
Повне використання коду R у цьому прикладі можна знайти тут .