K-medoids в r: покроковий приклад
Кластеризація – це техніка машинного навчання, яка намагається знайти групи або кластери спостережень у наборі даних.
Мета полягає в тому, щоб знайти такі кластери, щоб спостереження всередині кожного кластера були досить подібними одне до одного, тоді як спостереження в різних кластерах сильно відрізнялися одне від одного.
Кластеризація є формою неконтрольованого навчання , оскільки ми просто намагаємося знайти структуру в наборі даних, а не передбачити значення змінної відповіді .
Кластеризація часто використовується в маркетингу, коли компанії мають доступ до такої інформації, як:
- Доходи домашніх господарств
- Розмір домогосподарства
- Голова домашнього господарства Професія
- Відстань до найближчого населеного пункту
Коли ця інформація доступна, кластеризацію можна використати для виявлення домогосподарств, які схожі та з більшою ймовірністю купуватимуть певні продукти або краще реагуватимуть на певний тип реклами.
Одна з найпоширеніших форм кластеризації відома як k-середнє кластеризування .
На жаль, на цей метод можуть впливати викиди, тому часто використовуваною альтернативою є кластеризація k-medoids .
Що таке кластеризація K-Medoids?
Кластеризація K-medoids — це техніка, за якої ми поміщаємо кожне спостереження в наборі даних в один із K кластерів.
Кінцева мета полягає в тому, щоб мати K кластерів, у яких спостереження всередині кожного кластера досить подібні одне до одного, тоді як спостереження в різних кластерах досить відрізняються одне від одного.
На практиці ми використовуємо такі кроки для кластеризації K-середніх:
1. Виберіть значення для K.
- По-перше, нам потрібно вирішити, скільки кластерів ми хочемо визначити в даних. Часто нам просто потрібно перевірити кілька різних значень K і проаналізувати результати, щоб побачити, яка кількість кластерів є найбільш доцільною для даної проблеми.
2. Випадково призначте кожне спостереження початковому кластеру від 1 до K.
3. Виконуйте наступну процедуру, доки призначення кластера не перестануть змінюватися.
- Для кожного з K кластерів обчисліть центр тяжіння кластера. Це вектор p медіан ознак для спостережень k- го кластера.
- Призначте кожне спостереження кластеру з найближчим центроїдом. Тут найближче визначається за допомогою евклідової відстані .
Технічна примітка:
Оскільки k-medoids обчислює центроїди кластерів, використовуючи медіани, а не середні значення, він, як правило, більш стійкий до викидів, ніж k-середні.
На практиці, якщо в наборі даних немає екстремальних викидів, k-середні та k-медоїди дадуть подібні результати.
Кластеризація K-Medoids у R
У наступному посібнику наведено покроковий приклад того, як виконати кластеризацію k-medoids у R.
Крок 1: Завантажте необхідні пакети
Спочатку ми завантажимо два пакунки, що містять кілька корисних функцій для кластеризації k-medoids у R.
library (factoextra) library (cluster)
Крок 2: Завантажте та підготуйте дані
Для цього прикладу ми використаємо набір даних USArrests , вбудований у R, який містить кількість арештів на 100 000 осіб у кожному штаті США у 1973 році за вбивства , напади та зґвалтування , а також відсоток населення кожного штату, що живе в містах. області. , UrbanPop .
Наступний код показує, як зробити наступне:
- Завантажити набір даних USArrests
- Видаліть усі рядки з відсутніми значеннями
- Масштабуйте кожну змінну в наборі даних, щоб мати середнє значення 0 і стандартне відхилення 1
#load data df <-USArrests #remove rows with missing values df <- na. omitted (df) #scale each variable to have a mean of 0 and sd of 1 df <- scale(df) #view first six rows of dataset head(df) Murder Assault UrbanPop Rape Alabama 1.24256408 0.7828393 -0.5209066 -0.003416473 Alaska 0.50786248 1.1068225 -1.2117642 2.484202941 Arizona 0.07163341 1.4788032 0.9989801 1.042878388 Arkansas 0.23234938 0.2308680 -1.0735927 -0.184916602 California 0.27826823 1.2628144 1.7589234 2.067820292 Colorado 0.02571456 0.3988593 0.8608085 1.864967207
Крок 3: Знайдіть оптимальну кількість кластерів
Щоб виконати кластеризацію k-medoid у R, ми можемо використати функцію pam() , яка означає «розбиття навколо медіан» і використовує такий синтаксис:
pam(дані, k, метрика = «евклідова», стандарт = FALSE)
золото:
- дані: назва набору даних.
- k: кількість кластерів.
- метрика: метрика для обчислення відстані. Типовим є Евклідове , але ви також можете вказати manhattan .
- stand: нормалізувати чи ні кожну змінну в наборі даних. Значення за замовчуванням – false.
Оскільки ми не знаємо заздалегідь, яка кількість кластерів є оптимальною, ми створимо два різних графіки, які допоможуть нам вирішити:
1. Кількість кластерів відносно загальної суми квадратів
Спочатку ми використаємо функцію fviz_nbclust() , щоб побудувати графік залежності кількості кластерів від загальної суми квадратів:
fviz_nbclust(df, pam, method = “ wss ”)
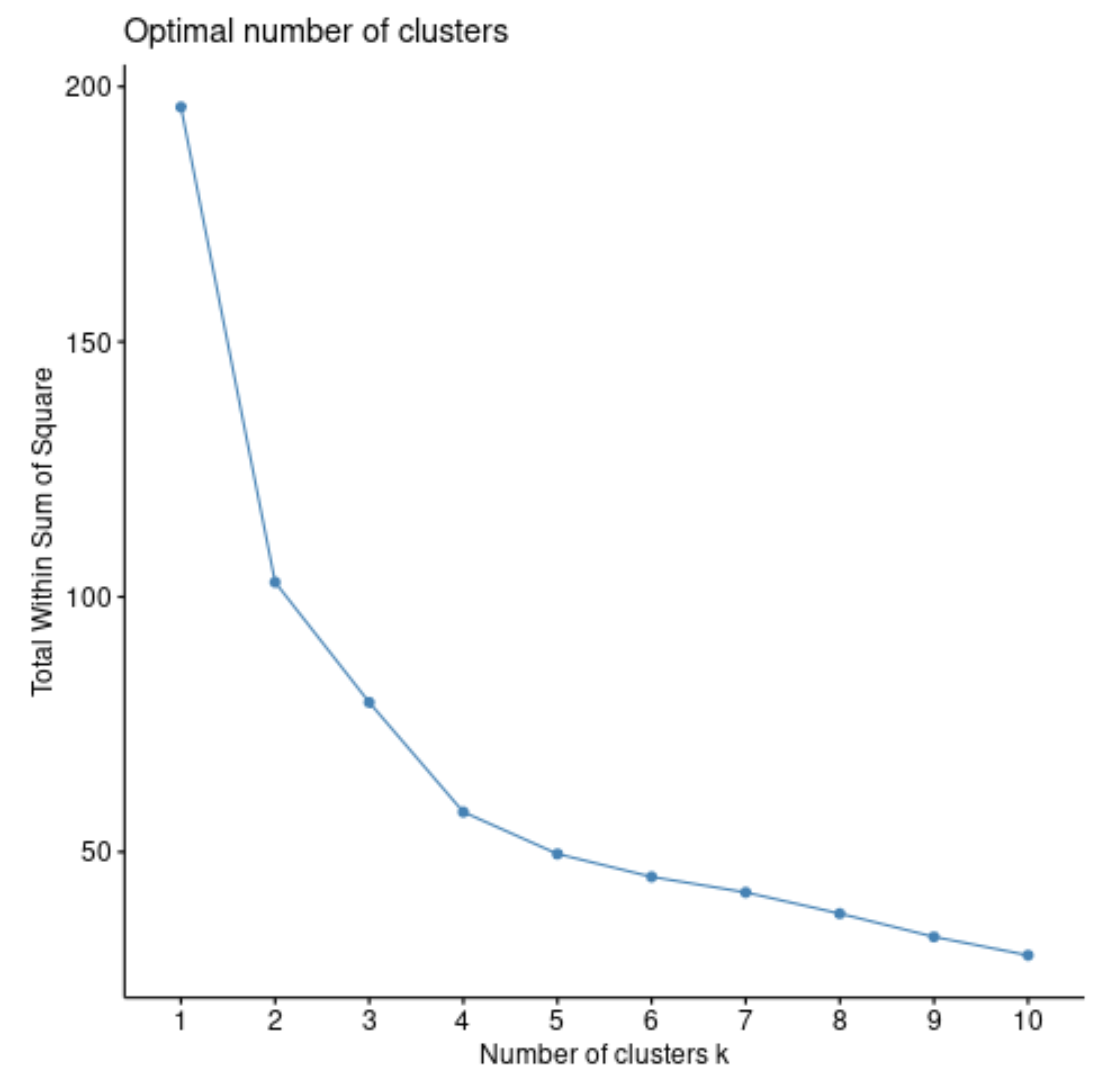
Загальна сума квадратів, як правило, завжди зростатиме зі збільшенням кількості кластерів. Отже, коли ми створюємо такий тип сюжету, ми шукаємо «коліно», де сума квадратів починає «згинатися» або вирівнюватися.
Точка кривизни графіка в цілому відповідає оптимальній кількості кластерів. Крім цієї цифри, ймовірно, відбудеться переобладнання .
Для цього графіка здається, що є невеликий перегин або «вигин» при k = 4 кластерах.
2. Кількість кластерів у порівнянні зі статистикою прогалин
Іншим способом визначення оптимальної кількості кластерів є використання метрики під назвою статистика відхилення , яка порівнює загальну внутрішньокластерну варіацію для різних значень k з їх очікуваними значеннями для розподілу без кластеризації.
Ми можемо обчислити статистику розриву для кожної кількості кластерів за допомогою функції clusGap() із пакета кластерів , а також графік залежності кластерів від статистики розриву за допомогою функції fviz_gap_stat() :
#calculate gap statistic based on number of clusters gap_stat <- clusGap(df, FUN = pam, K.max = 10, #max clusters to consider B = 50) #total bootstrapped iterations #plot number of clusters vs. gap statistic fviz_gap_stat(gap_stat)
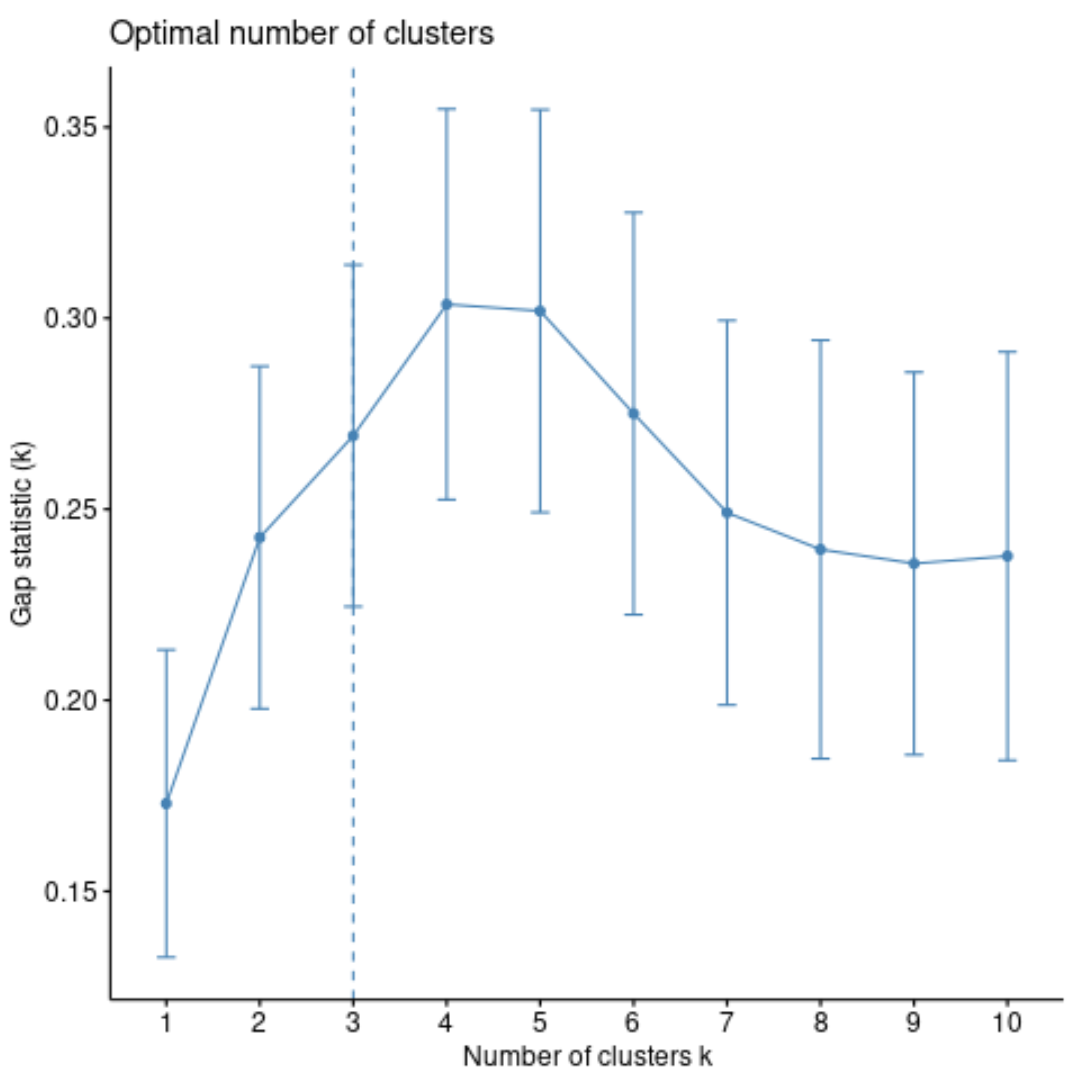
З графіка ми можемо побачити, що статистика розриву найвища при k = 4 кластерах, що відповідає методу ліктя, який ми використовували раніше.
Крок 4. Виконайте кластеризацію K-Medoids за допомогою Optimal K
Нарешті, ми можемо виконати кластеризацію k-medoids на наборі даних, використовуючи оптимальне значення для k , рівне 4:
#make this example reproducible set.seed(1) #perform k-medoids clustering with k = 4 clusters kmed <- pam(df, k = 4) #view results kmed ID Murder Assault UrbanPop Rape Alabama 1 1.2425641 0.7828393 -0.5209066 -0.003416473 Michigan 22 0.9900104 1.0108275 0.5844655 1.480613993 Oklahoma 36 -0.2727580 -0.2371077 0.1699510 -0.131534211 New Hampshire 29 -1.3059321 -1.3650491 -0.6590781 -1.252564419 Vector clustering: Alabama Alaska Arizona Arkansas California 1 2 2 1 2 Colorado Connecticut Delaware Florida Georgia 2 3 3 2 1 Hawaii Idaho Illinois Indiana Iowa 3 4 2 3 4 Kansas Kentucky Louisiana Maine Maryland 3 3 1 4 2 Massachusetts Michigan Minnesota Mississippi Missouri 3 2 4 1 3 Montana Nebraska Nevada New Hampshire New Jersey 3 3 2 4 3 New Mexico New York North Carolina North Dakota Ohio 2 2 1 4 3 Oklahoma Oregon Pennsylvania Rhode Island South Carolina 3 3 3 3 1 South Dakota Tennessee Texas Utah Vermont 4 1 2 3 4 Virginia Washington West Virginia Wisconsin Wyoming 3 3 4 4 3 Objective function: build swap 1.035116 1.027102 Available components: [1] "medoids" "id.med" "clustering" "objective" "isolation" [6] "clusinfo" "silinfo" "diss" "call" "data"
Зверніть увагу, що всі чотири центроїди кластера є фактичними спостереженнями в наборі даних. У верхній частині результату ми бачимо, що чотири центроїди є такими станами:
- Алабама
- Мічиган
- Оклахома
- Нью-Гемпшир
Ми можемо візуалізувати кластери на діаграмі розсіювання, яка відображає перші два головних компоненти на осях за допомогою функції fivz_cluster() :
#plot results of final k-medoids model
fviz_cluster(kmed, data = df)
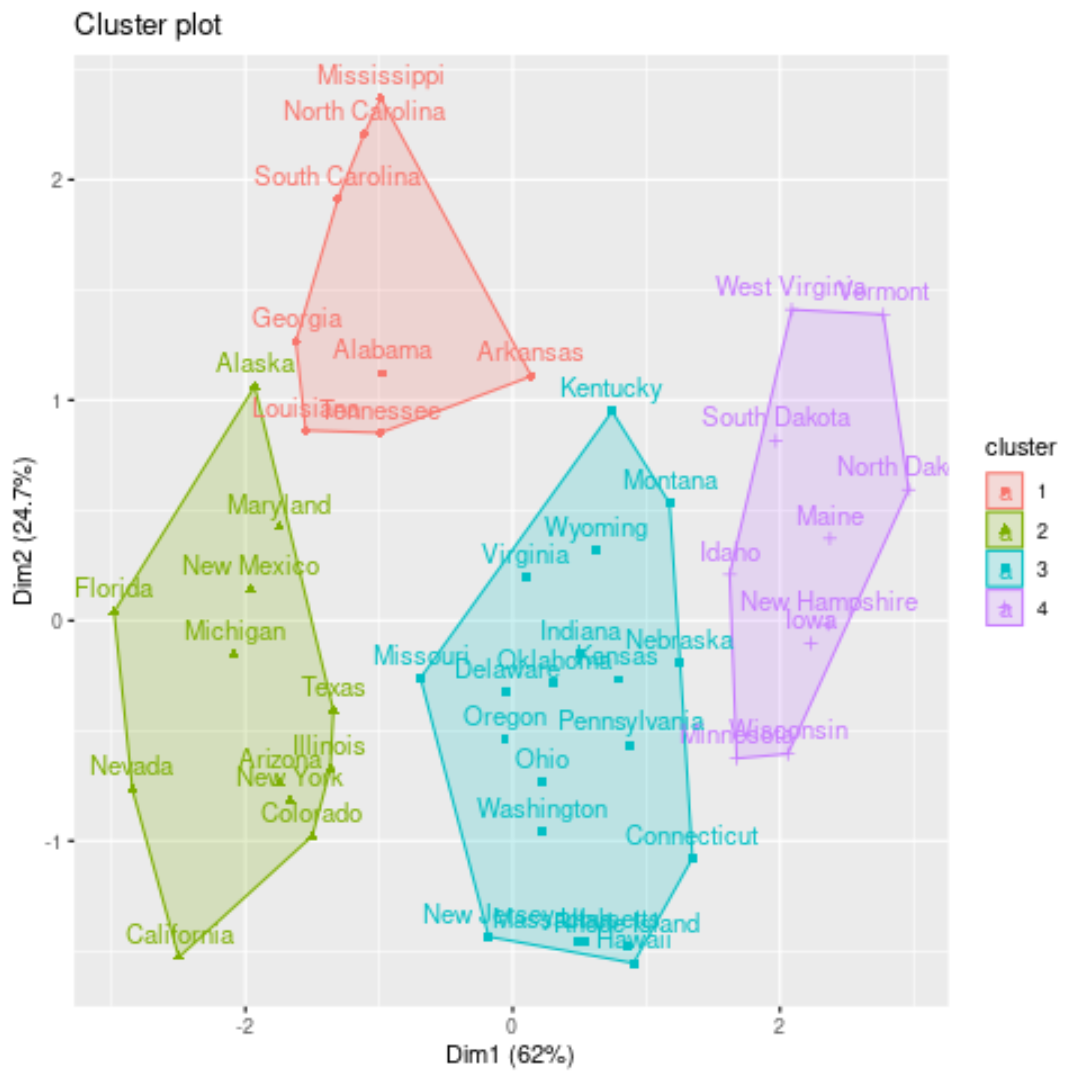
Ми також можемо додати кластерні призначення кожного стану до вихідного набору даних:
#add cluster assignment to original data
final_data <- cbind(USArrests, cluster = kmed$cluster)
#view final data
head(final_data)
Murder Assault UrbanPop Rape cluster
Alabama 13.2 236 58 21.2 1
Alaska 10.0 263 48 44.5 2
Arizona 8.1 294 80 31.0 2
Arkansas 8.8 190 50 19.5 1
California 9.0 276 91 40.6 2
Colorado 7.9 204 78 38.7 2
Ви можете знайти повний код R, використаний у цьому прикладі , тут .