Régression des composantes principales en Python (étape par étape)
Étant donné un ensemble de p variables prédictives et une variable de réponse, la régression linéaire multiple utilise une méthode connue sous le nom de moindres carrés pour minimiser la somme des carrés résiduels (RSS) :
RSS = Σ(y je – ŷ je ) 2
où:
- Σ : Un symbole grec qui signifie somme
- y i : la valeur de réponse réelle pour la ième observation
- ŷ i : La valeur de réponse prédite basée sur le modèle de régression linéaire multiple
Cependant, lorsque les variables prédictives sont fortement corrélées, la multicolinéarité peut devenir un problème. Cela peut rendre les estimations des coefficients du modèle peu fiables et présenter une variance élevée.
Une façon d’éviter ce problème consiste à utiliser la régression en composantes principales , qui trouve M combinaisons linéaires (appelées « composantes principales ») des p prédicteurs d’origine, puis utilise les moindres carrés pour ajuster un modèle de régression linéaire en utilisant les composantes principales comme prédicteurs.
Ce didacticiel fournit un exemple étape par étape de la façon d’effectuer une régression des composantes principales en Python.
Étape 1 : Importer les packages nécessaires
Tout d’abord, nous allons importer les packages nécessaires pour effectuer une régression en composantes principales (PCR) en Python :
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.preprocessing import scale
from sklearn import model_selection
from sklearn.model_selection import RepeatedKFold
from sklearn.model_selection import train_test_split
from sklearn.decomposition import PCA
from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error
Étape 2 : Charger les données
Pour cet exemple, nous utiliserons un ensemble de données appelé mtcars , qui contient des informations sur 33 voitures différentes. Nous utiliserons hp comme variable de réponse et les variables suivantes comme prédicteurs :
- mpg
- afficher
- merde
- poids
- qsec
Le code suivant montre comment charger et afficher cet ensemble de données :
#define URL where data is located
url = "https://raw.githubusercontent.com/Statology/Python-Guides/main/mtcars.csv"
#read in data
data_full = pd.read_csv(url)
#select subset of data
data = data_full[["mpg", "disp", "drat", "wt", "qsec", "hp"]]
#view first six rows of data
data[0:6]
mpg disp drat wt qsec hp
0 21.0 160.0 3.90 2.620 16.46 110
1 21.0 160.0 3.90 2.875 17.02 110
2 22.8 108.0 3.85 2.320 18.61 93
3 21.4 258.0 3.08 3.215 19.44 110
4 18.7 360.0 3.15 3.440 17.02 175
5 18.1 225.0 2.76 3.460 20.22 105
Étape 3 : Ajuster le modèle PCR
Le code suivant montre comment adapter le modèle PCR à ces données. Notez ce qui suit :
- pca.fit_transform(scale(X)) : cela indique à Python que chacune des variables prédictives doit être mise à l’échelle pour avoir une moyenne de 0 et un écart type de 1. Cela garantit qu’aucune variable prédictive n’a trop d’influence dans le modèle si cela se produit. à mesurer dans différentes unités.
- cv = RepeatedKFold() : cela indique à Python d’utiliser la validation croisée k-fold pour évaluer les performances du modèle. Pour cet exemple nous choisissons k = 10 plis, répété 3 fois.
#define predictor and response variables
X = data[["mpg", "disp", "drat", "wt", "qsec"]]
y = data[["hp"]]
#scale predictor variables
pca = PCA()
X_reduced = pca.fit_transform(scale(X))
#define cross validation method
cv = RepeatedKFold(n_splits=10, n_repeats=3, random_state=1)
regr = LinearRegression()
mse = []
# Calculate MSE with only the intercept
score = -1*model_selection.cross_val_score(regr,
np.ones((len(X_reduced),1)), y, cv=cv,
scoring='neg_mean_squared_error').mean()
mse.append(score)
# Calculate MSE using cross-validation, adding one component at a time
for i in np.arange(1, 6):
score = -1*model_selection.cross_val_score(regr,
X_reduced[:,:i], y, cv=cv, scoring='neg_mean_squared_error').mean()
mse.append(score)
# Plot cross-validation results
plt.plot(mse)
plt.xlabel('Number of Principal Components')
plt.ylabel('MSE')
plt.title('hp')
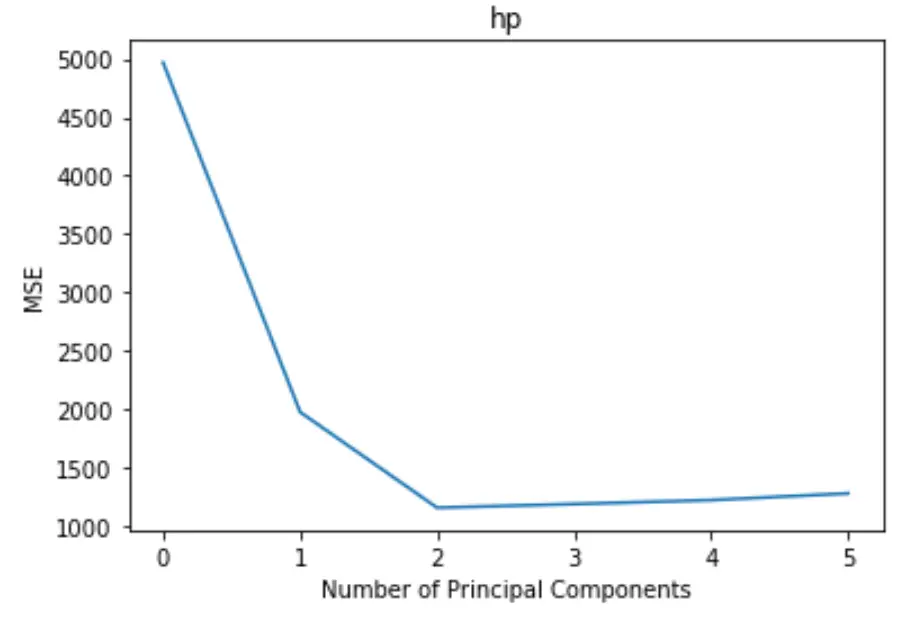
Le tracé affiche le nombre de composantes principales le long de l’axe des x et le test MSE (erreur quadratique moyenne) le long de l’axe des y.
À partir du graphique, nous pouvons voir que le MSE du test diminue en ajoutant deux composantes principales, mais il commence à augmenter à mesure que nous ajoutons plus de deux composantes principales.
Ainsi, le modèle optimal ne comprend que les deux premières composantes principales.
Nous pouvons également utiliser le code suivant pour calculer le pourcentage de variance de la variable de réponse expliqué en ajoutant chaque composante principale au modèle :
np.cumsum(np.round(pca.explained_variance_ratio_, decimals=4)*100)
array([69.83, 89.35, 95.88, 98.95, 99.99])
Nous pouvons voir ce qui suit :
- En utilisant uniquement la première composante principale, nous pouvons expliquer 69,83 % de la variation de la variable réponse.
- En ajoutant la deuxième composante principale, on peut expliquer 89,35 % de la variation de la variable réponse.
Notez que nous serons toujours en mesure d’expliquer plus de variance en utilisant plus de composantes principales, mais nous pouvons voir que l’ajout de plus de deux composantes principales n’augmente pas réellement le pourcentage de variance expliquée de beaucoup.
Étape 4 : Utiliser le modèle final pour faire des prédictions
Nous pouvons utiliser le modèle PCR final à deux composantes principales pour faire des prédictions sur de nouvelles observations.
Le code suivant montre comment diviser l’ensemble de données d’origine en un ensemble de formation et de test et utiliser le modèle PCR avec deux composants principaux pour faire des prédictions sur l’ensemble de test.
#split the dataset into training (70%) and testing (30%) sets
X_train,X_test,y_train,y_test = train_test_split(X,y,test_size=0.3,random_state=0)
#scale the training and testing data
X_reduced_train = pca.fit_transform(scale(X_train))
X_reduced_test = pca.transform(scale(X_test))[:,:1]
#train PCR model on training data
regr = LinearRegression()
regr.fit(X_reduced_train[:,:1], y_train)
#calculate RMSE
pred = regr.predict(X_reduced_test)
np.sqrt(mean_squared_error(y_test, pred))
40.2096
On voit que le test RMSE s’avère être 40.2096 . Il s’agit de l’écart moyen entre la valeur prédite de hp et la valeur observée de hp pour les observations de l’ensemble de tests.
Le code Python complet utilisé dans cet exemple peut être trouvé ici .